
| Sequence ID | X_DroMel_CAF1 |
|---|---|
| Location | 20,863,055 – 20,863,161 |
| Length | 106 |
| Max. P | 0.765668 |

| Location | 20,863,055 – 20,863,161 |
|---|---|
| Length | 106 |
| Sequences | 6 |
| Columns | 111 |
| Reading direction | reverse |
| Mean pairwise identity | 78.45 |
| Mean single sequence MFE | -33.18 |
| Consensus MFE | -22.98 |
| Energy contribution | -23.28 |
| Covariance contribution | 0.31 |
| Combinations/Pair | 1.21 |
| Mean z-score | -1.40 |
| Structure conservation index | 0.69 |
| SVM decision value | 0.51 |
| SVM RNA-class probability | 0.765668 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 20863055 106 - 22224390 CUGCGUCGGCA-CCAUUCUGUCGAUGCAGAUCUCGUUUGUGGGAUUCCAACCCAUCCAGAGCUCCGAACACAUGCUGGUGAGUGUAA--C-UACUUUGUGUAAUGCU-GUG (((((((((((-......))))))))))).........(((((.......))))).((.(((.....(((((...((((.......)--)-))...)))))...)))-.)) ( -34.00) >DroVir_CAF1 12169 109 - 1 UUGCGUUGGCA-CCAUUCUCUCGAUGCAGAUAUCGUUUGUGGGCUUCCAGCCCAUCCAGAGCUCCGAGCACAUGCUGGUAUGUCACAAACAUUUUUUGUUUAUUAUU-AUU .((((((((..-........))))))))(((((.((((((((((.....))))))...)))).((.(((....))))).)))))..(((((.....)))))......-... ( -27.10) >DroPse_CAF1 1937 108 - 1 CUGCGUGGGCA-CCAUUCUGUCCAUGCAGAUAUCGUUUGUGGGCUUCCAGCCCAUCCAGAGCUCCGAGCAUAUGCUGGUAAGUGCCAACC-GACCACGAGAACUGUC-CUC (((((((((((-......))))))))))).........((((((.....)))))).(((..(((..(((....)))(((..((....)).-.)))..)))..)))..-... ( -39.80) >DroYak_CAF1 2152 106 - 1 CUGCGUGGGCA-CCAUACUGUCGAUGCAGAUCUCAUUUGUGGGCUUCCAACCCAUCCAGAGCUCCGAGCACAUGCUGGUAAGUGUAA--C-UAUUUUGUGUAAUGCU-AUG ((((((.((((-......)))).)))))).........(((((.......))))).((.(((.....(((((...((((.......)--)-))...)))))...)))-.)) ( -27.50) >DroAna_CAF1 2056 107 - 1 UUGUGUGGGAA-CUAUACUGUCCAUGCAGAUAUCGUUCGUGGGCUUCCAGCCCAUACAGAGCUCGGAACACAUGCUGGUAAGGAUUG--C-AUUCCACUCUAGUUCUAGUU ..(((((.(((-(.(((((((....)))).))).))))((((((.....)))))).............)))))(((((...(((...--.-..)))...)))))....... ( -31.10) >DroPer_CAF1 1935 109 - 1 CUGCGUGGGCAACCAUUCUGUCCAUGCAGAUAUCGUUUGUGGGCUUCCAGCCCAUCCAGAGCUCCGAGCAUAUGCUGGUAAGUGCCAACC-GACCACGAGAACUGUC-UUA (((((((((((.......))))))))))).........((((((.....)))))).(((..(((..(((....)))(((..((....)).-.)))..)))..)))..-... ( -39.60) >consensus CUGCGUGGGCA_CCAUUCUGUCCAUGCAGAUAUCGUUUGUGGGCUUCCAGCCCAUCCAGAGCUCCGAGCACAUGCUGGUAAGUGUCA__C_UACCUUGUGUAAUGCU_AUG (((((((((((.......))))))))))).((((....((((((.....))))))....(((...........)))))))............................... (-22.98 = -23.28 + 0.31)

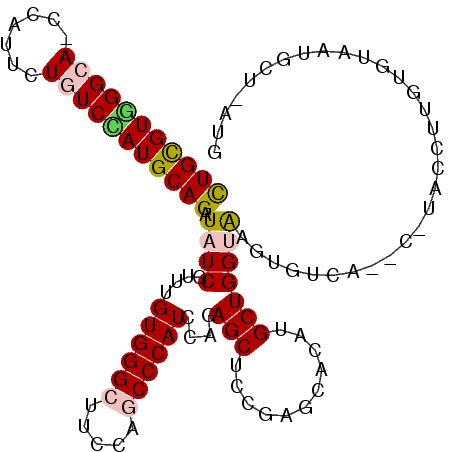

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 13:03:00 2006