
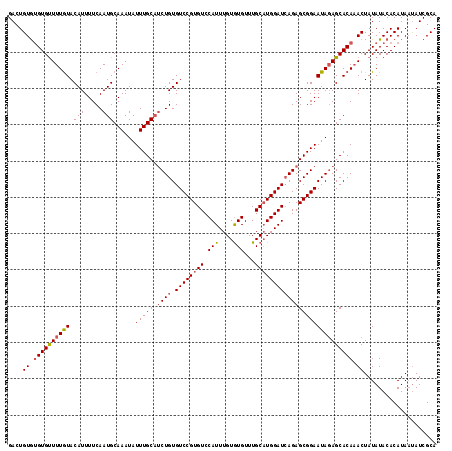
| Sequence ID | X_DroMel_CAF1 |
|---|---|
| Location | 20,792,973 – 20,793,091 |
| Length | 118 |
| Max. P | 0.978068 |

| Location | 20,792,973 – 20,793,091 |
|---|---|
| Length | 118 |
| Sequences | 5 |
| Columns | 118 |
| Reading direction | forward |
| Mean pairwise identity | 95.59 |
| Mean single sequence MFE | -31.93 |
| Consensus MFE | -26.58 |
| Energy contribution | -27.10 |
| Covariance contribution | 0.52 |
| Combinations/Pair | 1.09 |
| Mean z-score | -2.20 |
| Structure conservation index | 0.83 |
| SVM decision value | 1.81 |
| SVM RNA-class probability | 0.978068 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 20792973 118 + 22224390 GACUGUGUGUGCUUUGUACAUUUUCAAUGCAAAUAUUUGCAUCUGUGUCCGUGUCCAUUUGUGUGUUUGCAUGGAUCAGAGCGGAAUAGAGCACAAACUAUAUACACAUAAUAUCGCA ....((.((((((((((.(........(((((....)))))((((.((((((((.(((....)))...))))))))))))...).)))))))))).)).................... ( -32.20) >DroSec_CAF1 121489 118 + 1 GACUGUGUGUGUUUUGUACAUUUUCAAUGCAAAUAUUUGCAUCUGUGUCCGUGUCCAUUUGUGUGUUUGCAUGGAUAAGAGCGGAAUAGAGCACAAACUAUAUACACAUAAUAUCGCA ...((((((((((((((.(........(((((....)))))(((((.(((((....(((..(((....)))..)))....))))))))))).)))))..))))))))).......... ( -32.90) >DroSim_CAF1 58518 118 + 1 GACUGUGUGUGUUUUGUACAUUUUCAAUGCAAAUAUUUGCAUCUGUGUCCGUGUCCAUUUGUGUGUUUGCAUGGAUCAGAGCGGAAUAGAGCACAAACUAUAUACACAUAAUAUCGCA ...((((((((((((((.(........(((((....)))))(((((.(((((....(((..(((....)))..)))....))))))))))).)))))..))))))))).......... ( -32.90) >DroEre_CAF1 44099 118 + 1 GACUGUAUGUGUUUUAUACAUUUUCAAUGCAAAUAUUUGCUCCUGUGUCCGUGUCCAUUUGUGUGUUUGCAUGGAUCAGAGCGGAAUAGAGCACAAACUAUAUACACAUACCAUCGCA ((..((((((((..((((.........(((...(((((((((.((.((((((((.(((....)))...)))))))))))))).)))))..))).....)))).))))))))..))... ( -31.84) >DroYak_CAF1 96218 118 + 1 GACUGUGAGUGUUUUGUACAUUUUCAAUGCAAAUAUUUGCAUCUGUGUCCGUGUCCAUUUGUAUGUUUGCUUGGAUCAGAGCGGAAUACAGCACACACUAUAUACACAUAAUAUCGCA ...((((.((((..(((.........((((((....))))))(((((((((((((((...(((....))).)))))....)))).))))))...)))..)))).)))).......... ( -29.80) >consensus GACUGUGUGUGUUUUGUACAUUUUCAAUGCAAAUAUUUGCAUCUGUGUCCGUGUCCAUUUGUGUGUUUGCAUGGAUCAGAGCGGAAUAGAGCACAAACUAUAUACACAUAAUAUCGCA ....((.((((((((((.(((.....)))......(((((.((((.((((((((.(((....)))...))))))))))))))))))))))))))).)).................... (-26.58 = -27.10 + 0.52)


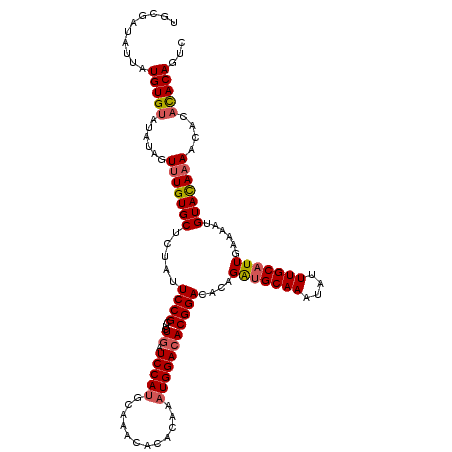
| Location | 20,792,973 – 20,793,091 |
|---|---|
| Length | 118 |
| Sequences | 5 |
| Columns | 118 |
| Reading direction | reverse |
| Mean pairwise identity | 95.59 |
| Mean single sequence MFE | -26.16 |
| Consensus MFE | -21.90 |
| Energy contribution | -22.42 |
| Covariance contribution | 0.52 |
| Combinations/Pair | 1.10 |
| Mean z-score | -1.61 |
| Structure conservation index | 0.84 |
| SVM decision value | 0.65 |
| SVM RNA-class probability | 0.812782 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 20792973 118 - 22224390 UGCGAUAUUAUGUGUAUAUAGUUUGUGCUCUAUUCCGCUCUGAUCCAUGCAAACACACAAAUGGACACGGACACAGAUGCAAAUAUUUGCAUUGAAAAUGUACAAAGCACACACAGUC ..........(((((......(((((((.....((((...((.(((((............)))))))))))....(((((((....)))))))......)))))))....)))))... ( -25.30) >DroSec_CAF1 121489 118 - 1 UGCGAUAUUAUGUGUAUAUAGUUUGUGCUCUAUUCCGCUCUUAUCCAUGCAAACACACAAAUGGACACGGACACAGAUGCAAAUAUUUGCAUUGAAAAUGUACAAAACACACACAGUC ..........(((((......(((((((.....((((......(((((............)))))..))))....(((((((....)))))))......)))))))....)))))... ( -24.60) >DroSim_CAF1 58518 118 - 1 UGCGAUAUUAUGUGUAUAUAGUUUGUGCUCUAUUCCGCUCUGAUCCAUGCAAACACACAAAUGGACACGGACACAGAUGCAAAUAUUUGCAUUGAAAAUGUACAAAACACACACAGUC ..........(((((......(((((((.....((((...((.(((((............)))))))))))....(((((((....)))))))......)))))))....)))))... ( -26.40) >DroEre_CAF1 44099 118 - 1 UGCGAUGGUAUGUGUAUAUAGUUUGUGCUCUAUUCCGCUCUGAUCCAUGCAAACACACAAAUGGACACGGACACAGGAGCAAAUAUUUGCAUUGAAAAUGUAUAAAACACAUACAGUC ...(((.((((((((..........((((((..((((...((.(((((............)))))))))))....))))))......(((((.....)))))....)))))))).))) ( -30.50) >DroYak_CAF1 96218 118 - 1 UGCGAUAUUAUGUGUAUAUAGUGUGUGCUGUAUUCCGCUCUGAUCCAAGCAAACAUACAAAUGGACACGGACACAGAUGCAAAUAUUUGCAUUGAAAAUGUACAAAACACUCACAGUC ((.((.......(((((((..(((((.((((.....(((........)))...(((....)))...)))))))))(((((((....)))))))....)))))))......)).))... ( -24.02) >consensus UGCGAUAUUAUGUGUAUAUAGUUUGUGCUCUAUUCCGCUCUGAUCCAUGCAAACACACAAAUGGACACGGACACAGAUGCAAAUAUUUGCAUUGAAAAUGUACAAAACACACACAGUC ..........(((((......(((((((.....((((...((.(((((............)))))))))))....(((((((....)))))))......)))))))....)))))... (-21.90 = -22.42 + 0.52)


Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 13:02:20 2006