
| Sequence ID | X_DroMel_CAF1 |
|---|---|
| Location | 20,790,311 – 20,790,479 |
| Length | 168 |
| Max. P | 0.883763 |

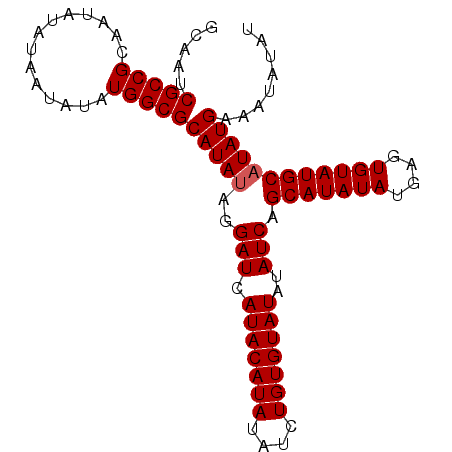
| Location | 20,790,311 – 20,790,405 |
|---|---|
| Length | 94 |
| Sequences | 4 |
| Columns | 95 |
| Reading direction | reverse |
| Mean pairwise identity | 97.02 |
| Mean single sequence MFE | -20.16 |
| Consensus MFE | -18.84 |
| Energy contribution | -19.09 |
| Covariance contribution | 0.25 |
| Combinations/Pair | 1.00 |
| Mean z-score | -2.07 |
| Structure conservation index | 0.93 |
| SVM decision value | 0.93 |
| SVM RNA-class probability | 0.883763 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 20790311 94 - 22224390 GCAAUCGCCGCAAUAUAUAAUAUAUGGCGCAUAUAGGAUCAUACAUAU-UCUGUGUAUAUAUCAGCAUAUAUGAGUGUAUGCAUAUGAAAUAUAU .....(((((..((((...)))).)))))(((((..(((.((((((..-...))))))..))).(((((((....))))))))))))........ ( -19.20) >DroSec_CAF1 118953 93 - 1 GCAAUCGCCGCAAUAUAUAAU--AUGGCGCAUAUAGGAUCAUACAUAUAUCUGUGUAUAUAUCAGCAUAUAUGAGUGUAUGCAUAUGAAAUAUAU .....(((((...........--.)))))(((((..(((.(((((((....)))))))..))).(((((((....))))))))))))........ ( -19.70) >DroSim_CAF1 54777 95 - 1 GCAAUCGCCGCAAUAUAUAAUAUAUGGCGCAUAUAGGAUCAUACAUAUAUCUGUGUAUAUAUCAGCAUAUAUGAGUGUAUGCAUAUGAAAUAUAU .....(((((..((((...)))).)))))(((((..(((.(((((((....)))))))..))).(((((((....))))))))))))........ ( -20.00) >DroEre_CAF1 41575 95 - 1 GCAAUCGCCGCAAUAUAUAAUAAAUGGCGCAUACACGAUCAUACAUAUAUCUGUGUAUAUAUCAGCAUAUAUGAGUGUAUGCAUAUGAAAUAUAU ....((((((..............))))((((((((....(((((((....)))))))...(((.......)))))))))))....))....... ( -21.74) >consensus GCAAUCGCCGCAAUAUAUAAUAUAUGGCGCAUAUAGGAUCAUACAUAUAUCUGUGUAUAUAUCAGCAUAUAUGAGUGUAUGCAUAUGAAAUAUAU .....(((((..............)))))(((((..(((.(((((((....)))))))..))).(((((((....))))))))))))........ (-18.84 = -19.09 + 0.25)

| Location | 20,790,365 – 20,790,479 |
|---|---|
| Length | 114 |
| Sequences | 5 |
| Columns | 119 |
| Reading direction | forward |
| Mean pairwise identity | 94.74 |
| Mean single sequence MFE | -25.40 |
| Consensus MFE | -21.90 |
| Energy contribution | -22.10 |
| Covariance contribution | 0.20 |
| Combinations/Pair | 1.00 |
| Mean z-score | -1.39 |
| Structure conservation index | 0.86 |
| SVM decision value | 0.49 |
| SVM RNA-class probability | 0.757374 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 20790365 114 + 22224390 GAUCCUAUAUGCGCCAUAUAUUAUAUAUUGCGGCGAUUGCAUCGCAACGACAAUUUUGGCUCAUUAUAAAUUUGCCUUAAUUUUAAUGCAAAAUGUC--UCUGCCGCAGUCAG---UCC ...........(((.(((((...))))).)))..((((((...(((..((((.(((((...(((((.(((((......)))))))))))))))))))--..))).))))))..---... ( -24.90) >DroSec_CAF1 119008 112 + 1 GAUCCUAUAUGCGCCAU--AUUAUAUAUUGCGGCGAUUGCAUCGCAACGACAAUUUUGGCUCAUUAUAAAUUUGCCUUAAUUUUAAUGCAAAAUGUC--UCUGCCGCAGUCCG---ACC .................--.......(((((((((.((((...)))).((((.(((((...(((((.(((((......)))))))))))))))))))--..)))))))))...---... ( -23.40) >DroSim_CAF1 54832 114 + 1 GAUCCUAUAUGCGCCAUAUAUUAUAUAUUGCGGCGAUUGCAUCGCAACGACAAUUUUGGCUCAUUAUAAAUUUGCCUUAAUUUUAAUGCAAAAUGUC--UCUGCCGCAGUCCG---GCC ............(((((((...))))(((((((((.((((...)))).((((.(((((...(((((.(((((......)))))))))))))))))))--..)))))))))..)---)). ( -26.40) >DroEre_CAF1 41630 116 + 1 GAUCGUGUAUGCGCCAUUUAUUAUAUAUUGCGGCGAUUGCAUCGCAACGACAAUUUUGGCUCAUUAUAAAUUUGCCUUAAUUUUAAUGCAAAAUGUCUCUCUGCCGCAGACCG---CCC ....(((....))).............((((((((.((((...)))).((((.(((((...(((((.(((((......)))))))))))))))))))....))))))))....---... ( -24.40) >DroYak_CAF1 93718 117 + 1 UAUCCUAUAUGCGCCAUUUAUUAUAUAUUGCGGCGAUUGCAUCGCAACGACAAUUUUGGCUCAUUAUAAAUUUGCCUUAAUUUUAAUGCAAAAUGUC--UCUGCCGCAGUCCGUGGCCC ............(((((.........(((((((((.((((...)))).((((.(((((...(((((.(((((......)))))))))))))))))))--..)))))))))..))))).. ( -27.90) >consensus GAUCCUAUAUGCGCCAUAUAUUAUAUAUUGCGGCGAUUGCAUCGCAACGACAAUUUUGGCUCAUUAUAAAUUUGCCUUAAUUUUAAUGCAAAAUGUC__UCUGCCGCAGUCCG___CCC ..........................(((((((((.((((...)))).((((.(((((...(((((.(((((......)))))))))))))))))))....)))))))))......... (-21.90 = -22.10 + 0.20)


Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 13:02:17 2006