
| Sequence ID | X_DroMel_CAF1 |
|---|---|
| Location | 20,583,250 – 20,583,461 |
| Length | 211 |
| Max. P | 0.998988 |

| Location | 20,583,250 – 20,583,369 |
|---|---|
| Length | 119 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 94.58 |
| Mean single sequence MFE | -22.53 |
| Consensus MFE | -20.85 |
| Energy contribution | -20.93 |
| Covariance contribution | 0.08 |
| Combinations/Pair | 1.11 |
| Mean z-score | -1.42 |
| Structure conservation index | 0.93 |
| SVM decision value | 1.29 |
| SVM RNA-class probability | 0.938295 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 20583250 119 + 22224390 CCAAUAUCAUGCAUCCUGUAUUUCACCUUUUAAGCCAGGCACCCUGUGGUGUUGGUUAUGUCCUAAACGAUUCGCUAAGUCCUGUACAACAUCACGAAAUAUAACUGUAUCUAUGUU-UU ..(((((.(((((...((((((((.(((........)))......((((((((((((........)))((((.....)))).....)))))))))))))))))..)))))..)))))-.. ( -24.00) >DroVir_CAF1 202687 119 + 1 CCAAUAUCAUGCAUCCUGUAUUUCACCUUUUAAUCCAGGCACCCUGUGGUGUUGGUUAUGUCCUAAACGAUUUGCUAAAUCCUGUACAACAUCACGAAAUAUAACUGUAUCUAUAUU-UU ..(((((.(((((...((((((((.(((........)))......((((((((((((........)))((((.....)))).....)))))))))))))))))..)))))..)))))-.. ( -24.10) >DroGri_CAF1 182527 119 + 1 CCAAUAUCAUGCAUCCUGUAUUUCACCUUUUAAUCCAGGCACCCUAUGGUGUUGGUUAUGUCCUAAACGAUUCGCUAAGUCCUGUACAACAUCACGAAAUAUAACUGUAUCUAUAUU-UU ..(((((.(((((...((((((((.............((((((....)))))).((.((((..((...((((.....))))...))..)))).))))))))))..)))))..)))))-.. ( -20.60) >DroWil_CAF1 188181 119 + 1 CCAAUAUCAUGCAUCCUGUAUUUCACCUUUCAAUCCAGGCACCCUAUGGUGUUGGUUAUGUCCUAAACGAUUCGCUAAAUCCUGUACAACAUCACGAAAUAUAACCGUAUCUAUGUU-UU .((((((((((...((((.(((.........))).)))).....))))))))))((.((((..((...((((.....))))...))..)))).))((((((((........))))))-)) ( -19.10) >DroMoj_CAF1 251847 119 + 1 CCAAUAUCAUGCAUCCUGUAUUUCACCUUUCAAUCCAGGCACCCUGUGGUGUUGGUUAUGUCCUAAGCGAUUUGCUAAAUCCUGUACAACAUCACGAAAUAUAACUGUAUCUAUAUU-UU ..(((((.(((((...((((((((.(((........)))......(((((((((...........(((.....)))..........)))))))))))))))))..)))))..)))))-.. ( -24.60) >DroAna_CAF1 160345 120 + 1 CCAAUAUCAUGCAUCCUGUAUUUCACCUUUUAAGCCAGGCACCCUGUGGUGUUGGUUAUGUCCUAAACGAUUCGGUAAAGUCUUGACAACAUCACGAAAUAUAACCGUAUUUAUGUUUUU ..(((((.((((....((((((((.(((........)))......((((((((((((........)))((((......))))....)))))))))))))))))...))))..)))))... ( -22.80) >consensus CCAAUAUCAUGCAUCCUGUAUUUCACCUUUUAAUCCAGGCACCCUGUGGUGUUGGUUAUGUCCUAAACGAUUCGCUAAAUCCUGUACAACAUCACGAAAUAUAACUGUAUCUAUAUU_UU ..(((((.(((((...((((((((.(((........)))......((((((((((((........)))((((.....)))).....)))))))))))))))))..)))))..)))))... (-20.85 = -20.93 + 0.08)


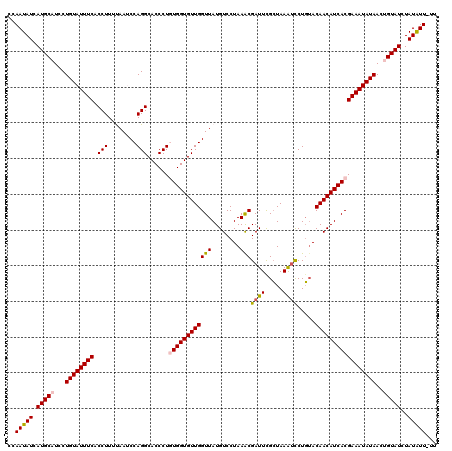
| Location | 20,583,330 – 20,583,447 |
|---|---|
| Length | 117 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 94.80 |
| Mean single sequence MFE | -31.97 |
| Consensus MFE | -29.75 |
| Energy contribution | -29.70 |
| Covariance contribution | -0.05 |
| Combinations/Pair | 1.14 |
| Mean z-score | -2.27 |
| Structure conservation index | 0.93 |
| SVM decision value | 3.31 |
| SVM RNA-class probability | 0.998988 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 20583330 117 - 22224390 UAGUGCGUCGUGACGCUAUUGAAAUAAUCGUUCGUCGU-UCAAAGAUGGGCGAUUGGUUUUUGUUGUAUUUAC-UAGGUGAA-AACAUAGAUACAGUUAUAUUUCGUGAUGUUGUACAGG ..(((((.(((.(((.........((((((((((((..-.....)))))))))))).......(((((((((.-...((...-.)).)))))))))........))).))).)))))... ( -33.40) >DroVir_CAF1 202767 117 - 1 UAGUGCGUCGUGACGCUAUUGAAAUAAUCGUUCGUCGU-UCCAAGAUGGGCGAUUGGUUUUUGUUGUAUUUAU-UAGGUGAA-AAUAUAGAUACAGUUAUAUUUCGUGAUGUUGUACAGG ..(((((.(((.(((.........((((((((((((..-.....)))))))))))).......((((((((((-...((...-.))))))))))))........))).))).)))))... ( -32.40) >DroGri_CAF1 182607 117 - 1 UAGUGCGUCGUGACGCAAUUGAAAUAAUCGUCCGUCGU-UCCAAGAUGGGCGAUUGGUUUUUGUUGUAUUUGC-UAGGUGAA-AAUAUAGAUACAGUUAUAUUUCGUGAUGUUGUACAGG ..(((((.(((.(((((((.((((((((((((((((..-.....))))))))))).))))).)))))......-.....(((-(.(((((......))))))))))).))).)))))... ( -36.10) >DroWil_CAF1 188261 117 - 1 UAGUGCGUCGUGACGCUAUUGAAAUAAUCGUUCGUCGU-UCAAAGAUGGGCGAUUGGUUUUUGUUGUAUUUAU-UAGGUGAA-AACAUAGAUACGGUUAUAUUUCGUGAUGUUGUACAGG ..(((((.(((.(((.........((((((((((((..-.....)))))))))))).......((((((((((-...((...-.))))))))))))........))).))).)))))... ( -32.90) >DroMoj_CAF1 251927 117 - 1 UAGUGCGUCGAGACGCAAUUGAAAUAAUCGUUCGUCGU-UCCAAGAUGGGCGAUUGGUUUUUGUUGUAUUUAU-UAGGUGAA-AAUAUAGAUACAGUUAUAUUUCGUGAUGUUGUACAGG ..(((((.(((((.((((..(...((((((((((((..-.....)))))))))))).)..))))(((((((((-...((...-.)))))))))))......)))))......)))))... ( -29.90) >DroAna_CAF1 160425 120 - 1 UAGUGCGUCGUGACGCUAUUGAAAUAAUCGUUCGUCGUUUCAAAGAUGGGCGAUUGGUUUUUGUUGUAUUUAUUUAGGUGAAAAACAUAAAUACGGUUAUAUUUCGUGAUGUUGUCAAGA .......((.(((((.((((((((((((((((((((........)))))))))).........((((((((((....((.....))))))))))))...))))))..)))).))))).)) ( -27.10) >consensus UAGUGCGUCGUGACGCUAUUGAAAUAAUCGUUCGUCGU_UCAAAGAUGGGCGAUUGGUUUUUGUUGUAUUUAU_UAGGUGAA_AACAUAGAUACAGUUAUAUUUCGUGAUGUUGUACAGG ..(((((.(((.(((.........((((((((((((........)))))))))))).......((((((((((....((.....))))))))))))........))).))).)))))... (-29.75 = -29.70 + -0.05)

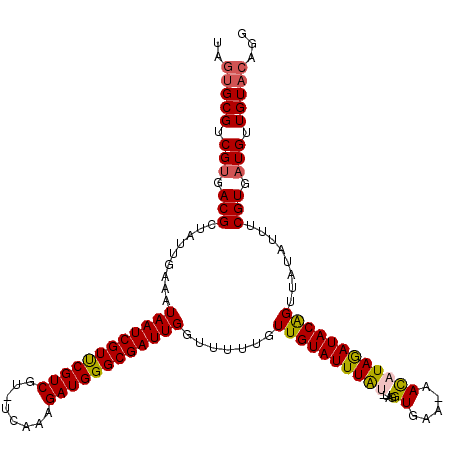
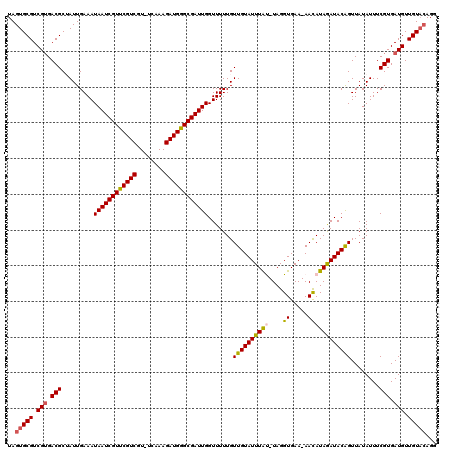
| Location | 20,583,369 – 20,583,461 |
|---|---|
| Length | 92 |
| Sequences | 6 |
| Columns | 118 |
| Reading direction | reverse |
| Mean pairwise identity | 82.04 |
| Mean single sequence MFE | -26.05 |
| Consensus MFE | -22.16 |
| Energy contribution | -21.63 |
| Covariance contribution | -0.53 |
| Combinations/Pair | 1.14 |
| Mean z-score | -1.56 |
| Structure conservation index | 0.85 |
| SVM decision value | 0.45 |
| SVM RNA-class probability | 0.739332 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 20583369 92 - 22224390 -CUUUAA-------------------------UUGCAGGUUAGUGCGUCGUGACGCUAUUGAAAUAAUCGUUCGUCGUUCAAAGAUGGGCGAUUGGUUUUUGUUGUAUUUACUAGGUG -((.(((-------------------------.(((((.(((((((((....)))).)))))..((((((((((((.......)))))))))))).......))))).)))..))... ( -24.80) >DroVir_CAF1 202806 99 - 1 -UUUCUAUUCGA------------------UUUUUCAGGUUAGUGCGUCGUGACGCUAUUGAAAUAAUCGUUCGUCGUUCCAAGAUGGGCGAUUGGUUUUUGUUGUAUUUAUUAGGUG -..((((...((------------------(....(((((((((((((....)))).)))))..((((((((((((.......))))))))))))...))))....)))...)))).. ( -23.30) >DroPse_CAF1 187567 93 - 1 AUUCCAA-------------------------UUGCAGGUUAGUGCGUCGUGACGCUAUUGAAAUAAUCGUUCGUCGUUCGAAGAUGGGCGAUUGGUUUUUGUUGUAUUUACUAGGUG ((..(((-------------------------..((...(((((((((....)))).)))))..((((((((((((.......))))))))))))))..)))..))............ ( -24.50) >DroGri_CAF1 182646 117 - 1 -UCUCUUUUUCUCAUCCUUUAUUCAUCGAUCAUUGUAGGUUAGUGCGUCGUGACGCAAUUGAAAUAAUCGUCCGUCGUUCCAAGAUGGGCGAUUGGUUUUUGUUGUAUUUGCUAGGUG -...........((((......(((.(((((((((.....))))).))))))).(((((.((((((((((((((((.......))))))))))).))))).)))))........)))) ( -32.10) >DroWil_CAF1 188300 91 - 1 -UCCCAA-------------------------UU-CAGGUUAGUGCGUCGUGACGCUAUUGAAAUAAUCGUUCGUCGUUCAAAGAUGGGCGAUUGGUUUUUGUUGUAUUUAUUAGGUG -..((..-------------------------((-(((......((((....))))..))))).((((((((((((.......))))))))))))...................)).. ( -22.90) >DroMoj_CAF1 251966 99 - 1 -UCUCCGUUUCA------------------CUUCACAGGUUAGUGCGUCGAGACGCAAUUGAAAUAAUCGUUCGUCGUUCCAAGAUGGGCGAUUGGUUUUUGUUGUAUUUAUUAGGUG -...((((((((------------------(..(((......))).)).)))))(((((.((((((((((((((((.......))))))))))).))))).)))))........)).. ( -28.70) >consensus _UCUCAA_________________________UUGCAGGUUAGUGCGUCGUGACGCUAUUGAAAUAAUCGUUCGUCGUUCCAAGAUGGGCGAUUGGUUUUUGUUGUAUUUACUAGGUG .....................................(.(((((((((....))))........((((((((((((.......))))))))))))...............))))).). (-22.16 = -21.63 + -0.53)



Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 12:59:52 2006