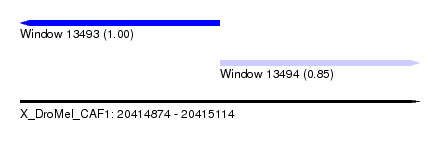
| Sequence ID | X_DroMel_CAF1 |
|---|---|
| Location | 20,414,874 – 20,415,114 |
| Length | 240 |
| Max. P | 0.995796 |

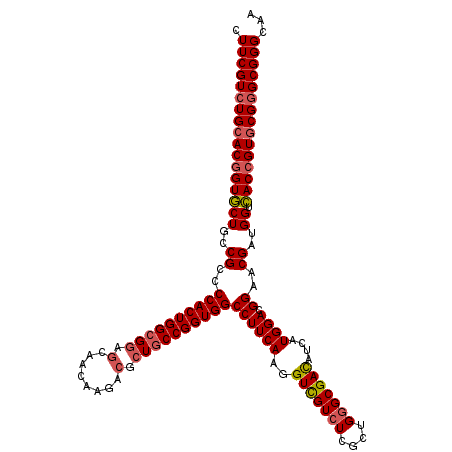
| Location | 20,414,874 – 20,414,994 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 94.06 |
| Mean single sequence MFE | -57.67 |
| Consensus MFE | -53.50 |
| Energy contribution | -54.12 |
| Covariance contribution | 0.61 |
| Combinations/Pair | 1.07 |
| Mean z-score | -1.98 |
| Structure conservation index | 0.93 |
| SVM decision value | 2.62 |
| SVM RNA-class probability | 0.995796 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 20414874 120 - 22224390 CUUCGUCUGCACGGUGCUCCCGCCCCACUGGCGGAGCAACAAGACGCUGCCGGUGGCCUUCAAGGUCGUCUCGCUGGGCGACAUUAUGGACGGAACGAUGGUCACCGUGCGGGCGGGCAA .(((((((((((((((((..((..((((((((((..(.....)...))))))))))((((((..(((((((....)))))))....)))).))..))..)).)))))))))))))))... ( -59.10) >DroSec_CAF1 3549 120 - 1 CUUCGUCUGCACGGUGCUGCCGCCCCACUGGCGGAGCAACAAGACGCUGCCGGUGGCCUUCAAGGUCGUCUCGCUGGGCGACAUCAUGGACGGAACGAUGGUCACCGUGCGGGCGGGCAA .(((((((((((((((((..((..((((((((((..(.....)...))))))))))((((((..(((((((....)))))))....)))).))..))..)).)))))))))))))))... ( -60.30) >DroAna_CAF1 4571 120 - 1 CUUCGUCUGCACCGUCCUACCGCCCCACUGGCGAUCGAACAAAACGCUACCGGUGGCCUUCAAGGUUGUGUCCUUGGGCGAUAUCAUGGACGGAACGAUGGUGACUGUCCGGGCGGGCAA .......(((.((((((...(((((((((((....((.......))...)))))))....(((((......))))))))).......((((((.((....))..))))))))))))))). ( -47.40) >DroSim_CAF1 174 120 - 1 CUUCGUCUGCACGGUGCUGCCGCCCCACUGGCGGAGCAACAAGACGCUGCCGGUGGCCUUCAAGGUCGUCUCGCUGGGCGACAUCAUGGACGGAACGAUGGUCACCGUGCGGGCGGGCAA .(((((((((((((((((..((..((((((((((..(.....)...))))))))))((((((..(((((((....)))))))....)))).))..))..)).)))))))))))))))... ( -60.30) >DroEre_CAF1 3526 120 - 1 CUUCGUCUGCACGGUGCUGCCGCCCCACUGGCGGAGCAACAAGACGCUGCCGGUGGCCUUCAAGGUCGUCUCGCUGGGCGACAUCAUGGACGGCACGAUGGUCACCGUGCGGGCGGGCAA .(((((((((((((((((((((((((((((((((..(.....)...))))))))))........(((((((....))))))).....)).)))).....)).)))))))))))))))... ( -62.10) >DroYak_CAF1 185 120 - 1 CUUCGUAUGCACGGUGCUGCCGCCCCACUGGCGGAGCAACAAGACGCUGCCGGUGGCCUUCAAGGUCGUCUCGCUGGGCGACAUCAUGGACGGCACGAUGGUCACCGUGCGGGCGGGCAA .(((((.(((((((((((((((((((((((((((..(.....)...))))))))))........(((((((....))))))).....)).)))).....)).))))))))).)))))... ( -56.80) >consensus CUUCGUCUGCACGGUGCUGCCGCCCCACUGGCGGAGCAACAAGACGCUGCCGGUGGCCUUCAAGGUCGUCUCGCUGGGCGACAUCAUGGACGGAACGAUGGUCACCGUGCGGGCGGGCAA .(((((((((((((((((..((..((((((((((.(........).))))))))))((((((..(((((((....)))))))....)))).))..))..)).)))))))))))))))... (-53.50 = -54.12 + 0.61)

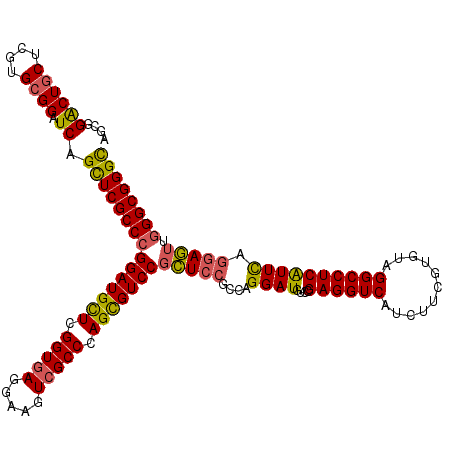
| Location | 20,414,994 – 20,415,114 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 93.94 |
| Mean single sequence MFE | -55.92 |
| Consensus MFE | -49.11 |
| Energy contribution | -48.78 |
| Covariance contribution | -0.33 |
| Combinations/Pair | 1.20 |
| Mean z-score | -1.64 |
| Structure conservation index | 0.88 |
| SVM decision value | 0.80 |
| SVM RNA-class probability | 0.853307 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 20414994 120 + 22224390 AGCGGACUGCUCGUGCGGAUCAGCUCGCCCGGAUGCUCGGUGAGGAAGUCGCCCAGCGUCCGCUCCGCCAGGAUGUCCGAGGUCAUCUUCGUGUAGGCCUCAUUUAGGAGUUGGGCGGGC ....((((((....)))).)).(((((((((((((((.(((((.....))))).)))))))(((((....((....))((((((...........)))))).....))))).)))))))) ( -59.50) >DroSec_CAF1 3669 120 + 1 AGCGGACUGCUCGUGCGGAUCAGCUCGCCCGGAUGCUCGGUGAGGAAGUCGCCCAGCGUCCGCUCCGCCAGGAUGUCCGAGGUCAUCUUCGUGUAGGCCUCAUUCAGGAGUUGGGCGGGC ....((((((....)))).)).(((((((((((((((.(((((.....))))).)))))))(((((....((....))((((((...........)))))).....))))).)))))))) ( -59.50) >DroAna_CAF1 4691 120 + 1 AGCGGGCUACUGGUGCGGAUCAGUUCGCCGGGAUGUUCGGUUAGGAAGUCGCCCAGUGUCCUUUCCGCCAGGAUGUCGGAGGUCAUUUUGGUGUAGGCCUCGUUUAAUAAUUGGGCGGGU .((((((.(((((.((((.....((((((((.....)))))..)))..)))))))))))))....((((((((((.(....).))))))))))...))((((((((.....)))))))). ( -44.40) >DroSim_CAF1 294 120 + 1 AGCGGACUGCUCGUGCGGAUCAGCUCGCCCGGAUGCUCGGUGAGGAAGUCGCCCAGCGUCCGCUCCGCCAGGAUGUCCGAGGUCAUCUUCGUGUAGGCCUCAUUCAGGAGUUGGGCGGGC ....((((((....)))).)).(((((((((((((((.(((((.....))))).)))))))(((((....((....))((((((...........)))))).....))))).)))))))) ( -59.50) >DroEre_CAF1 3646 120 + 1 AGCGGACUGCUCGUGCGGAUCAGCUCGCCCGGAUGCUCGGUGAGGAAGUCGCCCAGCGUCCGCUCCGCCAGGAUGUCAGAGGUCAUCUUCGUAUAGGCCUCAUUCAGGAGUUGGGCGGGC ....((((((....)))).)).(((((((((((((((.(((((.....))))).)))))))(((((....((((....((((((...........)))))))))).))))).)))))))) ( -56.10) >DroYak_CAF1 305 120 + 1 AGCGGACUGCUCGUGCGGAUCAGCUCGCCCGGAUGCUCGGUGAGGAAGUCGCCCAGCGUCCGCUCCGCCAGGAUGUCGGAGGUCAUCUUCGUGUAGGCCUCAUUCAGGAGUUGGGCGGGC ....((((((....)))).)).(((((((((((((((.(((((.....))))).)))))))(((((.....((.....((((((...........))))))..)).))))).)))))))) ( -56.50) >consensus AGCGGACUGCUCGUGCGGAUCAGCUCGCCCGGAUGCUCGGUGAGGAAGUCGCCCAGCGUCCGCUCCGCCAGGAUGUCCGAGGUCAUCUUCGUGUAGGCCUCAUUCAGGAGUUGGGCGGGC ....((((((....)))).)).(((((((((((((((.(((((.....))))).)))))))(((((....((((....((((((...........)))))))))).))))).)))))))) (-49.11 = -48.78 + -0.33)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 12:58:32 2006