
| Sequence ID | X_DroMel_CAF1 |
|---|---|
| Location | 20,285,973 – 20,286,212 |
| Length | 239 |
| Max. P | 0.951690 |

| Location | 20,285,973 – 20,286,081 |
|---|---|
| Length | 108 |
| Sequences | 6 |
| Columns | 114 |
| Reading direction | forward |
| Mean pairwise identity | 77.98 |
| Mean single sequence MFE | -46.48 |
| Consensus MFE | -19.38 |
| Energy contribution | -19.68 |
| Covariance contribution | 0.31 |
| Combinations/Pair | 1.21 |
| Mean z-score | -2.31 |
| Structure conservation index | 0.42 |
| SVM decision value | 0.23 |
| SVM RNA-class probability | 0.643800 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 20285973 108 + 22224390 AUCACCGGCGAAAUGGAGUCGCUGGUGGCGGCGACAAUAGCCGCAGCUGCUGGUAGCGCAGAAACAUG---AUGACUGCCACUAGCGUCUGCAAUGC---CACCGGAAUGGGAG .((.(((((........))).(((((((((((.......)))((((.(((((((.(.((((.......---....)))))))))))).))))...))---))))))...)))). ( -47.30) >DroPse_CAF1 24661 102 + 1 AUCACCGGCGAUAUAGAGUCACUGGUGGCGGCCACAAUGGCUGCGGCAGCGGGCAAGGCAGCCACAUG---GUGG-UGC--------UCCGCCAUGCCCCCACCCGAAUGUGAG ..((((((.(((.....))).))))))((((((.....))))))((((...(((..((((.((((...---))))-)))--------)..))).))))..(((......))).. ( -45.60) >DroEre_CAF1 10539 108 + 1 AUCACCGGCGAAAUAGAGUCGCUAGUGGCAGCGACAAUAGCCGCAGCUGCAGGCAGCGCAGAAACAUG---GUGGCUGCCACUAGCGUCUGCAAUGC---CACCCGAAUGAGAG ......((((...((((..(((((((((((((.((.....(.((.((((....)))))).).......---)).)))))))))))))))))...)))---)............. ( -46.50) >DroYak_CAF1 10581 108 + 1 AUCACCGGCGAAAUGGAGUCACUGGUAGCAGCUACAAUAGCCGCAGCUGCUGGUAGCGCAGAAACAUG---GUGACUGCCACUAGCGUCUGCAAUGC---CACCAGAAUGAGAG .(((..(((........))).((((((((((((.(.......).)))))))((((..(((((....((---(((.....)))))...)))))..)))---))))))..)))... ( -39.50) >DroAna_CAF1 13368 111 + 1 AUCACCGGGGAAAUGGAGUCACUGGUGGCGGCCACGAUGGCGGCUGCGGCCGGCAGGGCCGAGACAUGAUGGUGGCCGCCAGCCGCCUCGGCCAGGC---CGCCGGAAUGGGAG .(((((((.((.......)).)))))(((((((.(((.((((((((((((((.((.(.......)....)).)))))).)))))))))))....)))---)))).))....... ( -58.90) >DroPer_CAF1 24718 102 + 1 AUCACCGGCGAUAUAGAGUCACUGGUGGCGGCCACAAUUGCUGCGGCAGCGGGCAAGGCAGCCACAUG---GUGG-UGC--------UCCGCCAUGCCCCCACCCGAAUGUGAG ..((((((.(((.....))).))))))((.(((.((.....)).))).))(((((.(((.(((((...---))))-)..--------...))).))))).(((......))).. ( -41.10) >consensus AUCACCGGCGAAAUAGAGUCACUGGUGGCGGCCACAAUAGCCGCAGCUGCGGGCAGCGCAGAAACAUG___GUGGCUGCCACUAGCGUCUGCAAUGC___CACCCGAAUGAGAG ..((((((.((.......)).))))))((((........(((((....)).)))...((((...(((....))).)))).........))))...................... (-19.38 = -19.68 + 0.31)

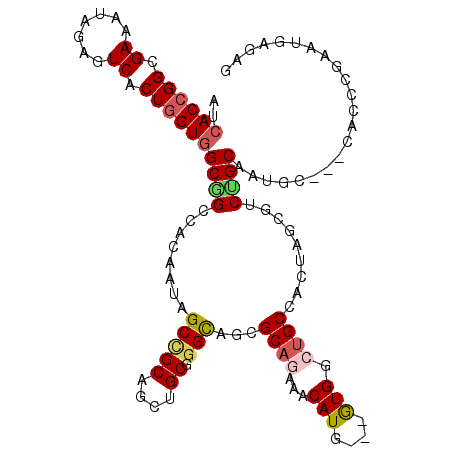
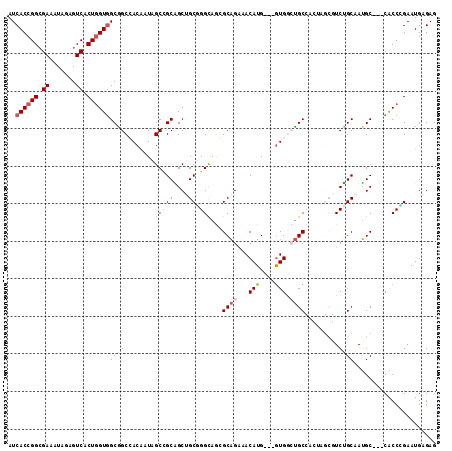
| Location | 20,285,973 – 20,286,081 |
|---|---|
| Length | 108 |
| Sequences | 6 |
| Columns | 114 |
| Reading direction | reverse |
| Mean pairwise identity | 77.98 |
| Mean single sequence MFE | -46.00 |
| Consensus MFE | -25.19 |
| Energy contribution | -23.78 |
| Covariance contribution | -1.41 |
| Combinations/Pair | 1.43 |
| Mean z-score | -2.46 |
| Structure conservation index | 0.55 |
| SVM decision value | 1.41 |
| SVM RNA-class probability | 0.951690 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 20285973 108 - 22224390 CUCCCAUUCCGGUG---GCAUUGCAGACGCUAGUGGCAGUCAU---CAUGUUUCUGCGCUACCAGCAGCUGCGGCUAUUGUCGCCGCCACCAGCGACUCCAUUUCGCCGGUGAU ........((((((---(...((.((.((((.(((((......---.......((((.......))))..(((((....))))).))))).)))).)).))..))))))).... ( -41.00) >DroPse_CAF1 24661 102 - 1 CUCACAUUCGGGUGGGGGCAUGGCGGA--------GCA-CCAC---CAUGUGGCUGCCUUGCCCGCUGCCGCAGCCAUUGUGGCCGCCACCAGUGACUCUAUAUCGCCGGUGAU ..........((..((((((.(((((.--------.((-(...---...))).))))).))))).)..))((.(((.....))).))((((.((((.......)))).)))).. ( -45.10) >DroEre_CAF1 10539 108 - 1 CUCUCAUUCGGGUG---GCAUUGCAGACGCUAGUGGCAGCCAC---CAUGUUUCUGCGCUGCCUGCAGCUGCGGCUAUUGUCGCUGCCACUAGCGACUCUAUUUCGCCGGUGAU ...(((((..((((---(.((.(.((.(((((((((((((.((---.(((...((((((((....)))).)))).))).)).))))))))))))).))).)).)))))))))). ( -48.70) >DroYak_CAF1 10581 108 - 1 CUCUCAUUCUGGUG---GCAUUGCAGACGCUAGUGGCAGUCAC---CAUGUUUCUGCGCUACCAGCAGCUGCGGCUAUUGUAGCUGCUACCAGUGACUCCAUUUCGCCGGUGAU .........(((((---((...(((((.((..((((......)---))))).))))))))))))(((((((((.....)))))))))((((.((((.......)))).)))).. ( -42.20) >DroAna_CAF1 13368 111 - 1 CUCCCAUUCCGGCG---GCCUGGCCGAGGCGGCUGGCGGCCACCAUCAUGUCUCGGCCCUGCCGGCCGCAGCCGCCAUCGUGGCCGCCACCAGUGACUCCAUUUCCCCGGUGAU .((((.....((((---(((....(((((((((((.(((((((......))...(((...)))))))))))))))).))).)))))))...((((....)))).....)).)). ( -54.20) >DroPer_CAF1 24718 102 - 1 CUCACAUUCGGGUGGGGGCAUGGCGGA--------GCA-CCAC---CAUGUGGCUGCCUUGCCCGCUGCCGCAGCAAUUGUGGCCGCCACCAGUGACUCUAUAUCGCCGGUGAU .((((.....((((.(((((.(((((.--------.((-(...---...))).))))).)))))((.(((((((...))))))).)))))).((((.......))))..)))). ( -44.80) >consensus CUCACAUUCGGGUG___GCAUGGCAGACGCUAGUGGCAGCCAC___CAUGUUUCUGCCCUGCCAGCAGCUGCGGCUAUUGUGGCCGCCACCAGUGACUCCAUUUCGCCGGUGAU ........((((((.......((((((.......((((..........)))))))))).))))))..((.((.((....)).)).))((((.((((.......)))).)))).. (-25.19 = -23.78 + -1.41)

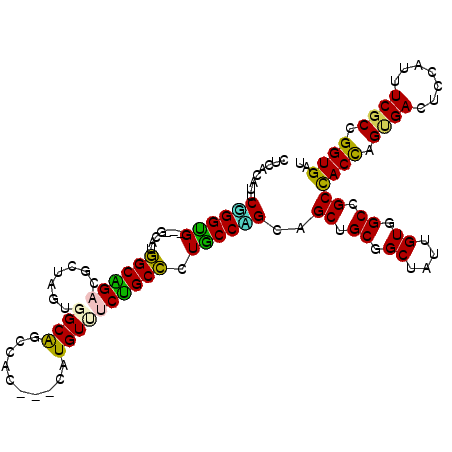

| Location | 20,286,081 – 20,286,183 |
|---|---|
| Length | 102 |
| Sequences | 6 |
| Columns | 108 |
| Reading direction | forward |
| Mean pairwise identity | 79.10 |
| Mean single sequence MFE | -40.85 |
| Consensus MFE | -25.51 |
| Energy contribution | -26.21 |
| Covariance contribution | 0.70 |
| Combinations/Pair | 1.43 |
| Mean z-score | -2.30 |
| Structure conservation index | 0.62 |
| SVM decision value | 1.24 |
| SVM RNA-class probability | 0.934626 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 20286081 102 + 22224390 CUCAAGGGUGAGCUGUCGGCUGCACUCGCUCCACC---UUGGACGUCGUCCAGAUA---ACUGGCGCUGGCCACUGUUUUCUGUGGUUCUGCCAACUGCUUGCCCUGU ....((((..(((.((.(((.((....)).((((.---..((((...))))(((.(---((((((....))))..))).)))))))....))).)).)))..)))).. ( -39.60) >DroPse_CAF1 24763 105 + 1 CUCAACGGGGAGCUAUCCAC---GCCGGCUCCACCAGUCACCACAUCGUCGAGAUAGUGGCUGGACGUGGCCACCGUUUUCUGUGGCUCCGCCAAUUGUUUGCCCUGC .....(((((.((.......---)).(((....((((((((...(((.....))).))))))))..(..(((((.(....).)))))..)))).........))))). ( -37.40) >DroSim_CAF1 18023 102 + 1 CUCAAGGGUGAGCUGUCGGCUGCACUUGCUCCACC---UUGGACAUCGUCCAGGUA---ACUGGCGCUAGCCACUGUUUUCUGUGGUUCUGCCAACUGCUUGCCCUGU ....((((..(((.((.(((.......((.(((.(---((((((...)))))))..---..))).)).((((((........))))))..))).)).)))..)))).. ( -42.50) >DroEre_CAF1 10647 102 + 1 CUCAAGGGUGAGCUGUCGGCUGCAAUUGCUCCGCC---UUGGACAUCGUCCAGAUA---GCUGGCGCUGGCCACUGUUUUCUGCGGUUCUGCCAACUGCUUGCCCUGU ....((((..(((.((.(((.((....)).((((.---.(((((...)))))((((---(.((((....)))))))))....))))....))).)).)))..)))).. ( -43.10) >DroYak_CAF1 10689 102 + 1 CUCAAGGGUGAGCUGUCGGCUGCACUCGCUGCACC---UUGGACAUCGUCCAGAUA---GCUGGCGCUGGCCACUGUUUUCUGCGGUUCUGCCAACUGCUUGCCUUGU ..(((((((.(((((((((.((((.....))))))---.(((((...)))))))))---))).))((((((.(((((.....)))))...))))...))...))))). ( -40.50) >DroMoj_CAF1 11509 91 + 1 CUCAACGGCGAGCUGUCCGCAGCGCCGGC------------CACGUCGUCCAGAUA---GCUGGACGUGGCUGCUGUUUUCUGUGGCUCCGCCAGCUGUUUGCCCG-- .....((((..((((....))))))))..------------.....((..((((((---(((((.((..((..(........)..))..)))))))))))))..))-- ( -42.00) >consensus CUCAAGGGUGAGCUGUCGGCUGCACUCGCUCCACC___UUGGACAUCGUCCAGAUA___GCUGGCGCUGGCCACUGUUUUCUGUGGUUCUGCCAACUGCUUGCCCUGU ....(((((((((.((.(((......((((........((((((...)))))).........))))..((((((........))))))..))).)).))))))))).. (-25.51 = -26.21 + 0.70)

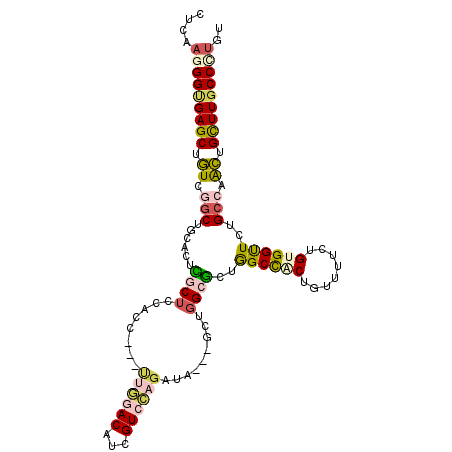

| Location | 20,286,115 – 20,286,212 |
|---|---|
| Length | 97 |
| Sequences | 6 |
| Columns | 105 |
| Reading direction | forward |
| Mean pairwise identity | 79.36 |
| Mean single sequence MFE | -33.62 |
| Consensus MFE | -22.74 |
| Energy contribution | -24.04 |
| Covariance contribution | 1.31 |
| Combinations/Pair | 1.29 |
| Mean z-score | -1.83 |
| Structure conservation index | 0.68 |
| SVM decision value | 0.85 |
| SVM RNA-class probability | 0.867323 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 20286115 97 + 22224390 CUUGGACGUCGUCCAGAUAACUGGCGCUGGCCACUGUUUUCUGUGGUUCUGCCAACUGCUUGCCCUGUGGCUGGUGCUCCUGUUAA-A--AAUGUAAU---GU-- .((((((...)))))).((((.(((((..(((((........(..(((.....)))..).......)))))..)))))...)))).-.--........---..-- ( -32.16) >DroSec_CAF1 15994 96 + 1 CUUGGACGUCGUCCAGGUAACUUGCGCUAGCCACUGUUUUCUGUGGUUCUGCCAACUGCUUGCCCUGUGGCUGAUGCUCCUGUUAA-A--AGU-UAAU---GU-- (((((((...)))))))((((..(((.(((((((........(..(((.....)))..).......))))))).)))....)))).-.--...-....---..-- ( -28.06) >DroSim_CAF1 18057 95 + 1 CUUGGACAUCGUCCAGGUAACUGGCGCUAGCCACUGUUUUCUGUGGUUCUGCCAACUGCUUGCCCUGUGGCUGAUGCUCCUGUUAA-A--AGU--AAU---GU-- (((((((...)))))))((((.((.(((((((((........(..(((.....)))..).......)))))))..)).)).)))).-.--...--...---..-- ( -29.56) >DroEre_CAF1 10681 99 + 1 CUUGGACAUCGUCCAGAUAGCUGGCGCUGGCCACUGUUUUCUGCGGUUCUGCCAACUGCUUGCCCUGUGGCUGGUGCUCCUGUUGA-AAUGGAUGCAU---GU-- .....((((((((((..((((.(((((..(((((........((((((.....)))))).......)))))..)))))...)))).-..)))))).))---))-- ( -40.36) >DroYak_CAF1 10723 100 + 1 CUUGGACAUCGUCCAGAUAGCUGGCGCUGGCCACUGUUUUCUGCGGUUCUGCCAACUGCUUGCCUUGUGGUUGGUGCUCCUGUUGUGGAAAGUAGUAU---GU-- .((((((...))))))...((((((((..(((((........((((((.....)))))).......)))))..))))(((......)))...))))..---..-- ( -34.56) >DroMoj_CAF1 11538 95 + 1 ----CACGUCGUCCAGAUAGCUGGACGUGGCUGCUGUUUUCUGUGGCUCCGCCAGCUGUUUGCCCG---CCGGCUGCUCCUAUACG-A--AACAUAAGCGUGUAA ----..((.((..(((((((((((.((..((..(........)..))..)))))))))))))..))---.))........((((((-.--........)))))). ( -37.00) >consensus CUUGGACAUCGUCCAGAUAACUGGCGCUGGCCACUGUUUUCUGUGGUUCUGCCAACUGCUUGCCCUGUGGCUGGUGCUCCUGUUAA_A__AGU_UAAU___GU__ .((((((...)))))).((((.((((((((((((........((((((.....)))))).......))))))))))))...)))).................... (-22.74 = -24.04 + 1.31)


Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 12:57:15 2006