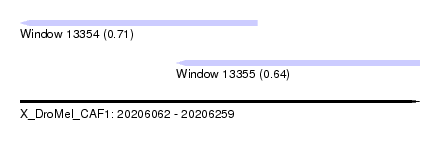
| Sequence ID | X_DroMel_CAF1 |
|---|---|
| Location | 20,206,062 – 20,206,259 |
| Length | 197 |
| Max. P | 0.707214 |

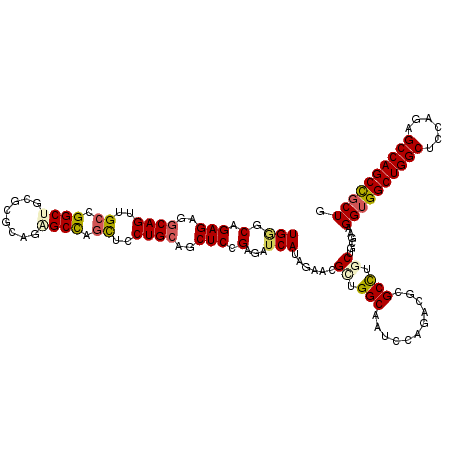
| Location | 20,206,062 – 20,206,179 |
|---|---|
| Length | 117 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 83.47 |
| Mean single sequence MFE | -60.48 |
| Consensus MFE | -42.35 |
| Energy contribution | -41.52 |
| Covariance contribution | -0.83 |
| Combinations/Pair | 1.22 |
| Mean z-score | -2.35 |
| Structure conservation index | 0.70 |
| SVM decision value | 0.37 |
| SVM RNA-class probability | 0.707214 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 20206062 117 - 22224390 CGCCUGCUGCGAAGGUGGCUGGCUCCAGAGCCAGCCGCUCAGAACCAUGUCGGUGGCCUGGCUAAGAUG---GGCUGCUUGCUGGGCGGCAGCUUGGGUGGUAAGCUUUUUGAUGGCCGU .(((((((((...(((((((((((....))))))))))).....(((.((.((..(((((.......))---)))..)).)))))))))))(((((.....)))))........)))... ( -55.30) >DroPse_CAF1 3486 117 - 1 GGCUGUCAGGGUGGGUGGCUGGCUCCACUGCCAGCUGCUGAGGAUCAGAUCGGUGGCCUGGCUGCCUUGUGCGGCUGCCUGCUGAGCGGCCGCC---GAGGUGAGCUUUUUGAUGGCCGU (((((((((....((..((((((......))))))..))((((.(((..(((((((((.(((.(((......))).))).((...)))))))))---))..))).))))))))))))).. ( -65.50) >DroSim_CAF1 33942 117 - 1 CGCCUGCUGCGAAGGUGGCUGGCUCCAGAGCCAGCCGCUCAGAACCAUGUCGGUGGCCUGGCUGAGAUG---GGCCGCUUGCUGUGCGGCGGCUUGGGUGGUAAGCUUCUUAAUGGCCGU ((((.((.(((..(((((((((((....))))))))))).....((((.(((((......))))).)))---)..)))..)).).)))((((((((((.((....)).))))..)))))) ( -55.20) >DroEre_CAF1 22497 117 - 1 CGCCUGCUGCGACGGUGGCUGGCUCCAGAGCCAGCCGCUGAGGACCAUGUCGGUGGCCUGGCUGAGAUG---GGCUGCCUGCUGGGCGGCGGCCUGGGUGGUGAGCUUCUUGAUGGCCGU (((((...((..((((((((((((....))))))))))))(((.((((.(((((......))))).)))---)....))))).)))))((((((.(((.((....)).)))...)))))) ( -62.00) >DroYak_CAF1 24512 117 - 1 CGCCUGCUGCGACGGGGGCUGGCUCCAAAGCCAGCCGCUAAGGACCAUGUCGGUGGCCUGGCUGAGAUG---GGCUGCUUGCUGGGCGGCGGCUUGGGUGGUGAGCUUUUUGAUGGCCGU .(((.(((((...((.((((((((....)))))))).)).....(((.((.((..(((((.......))---)))..)).)))))))))))))...(((.((.((....)).)).))).. ( -54.00) >DroPer_CAF1 2462 117 - 1 GGCUGUCAGGGUGGGUGGCUGGCUCCACUGCCAGCUGCUGAGGAUCAGAUCGGUGGCCUGGCUGCCGUGUGCGGCUGCCUGCUGGGCGGCCGCC---GAGGUGAGCUUCUUGAUGGCCGU (((((((((....((..((((((......))))))..))((((.(((..(((((((((.((((((.....))))))(((.....))))))))))---))..))).))))))))))))).. ( -70.90) >consensus CGCCUGCUGCGAAGGUGGCUGGCUCCAGAGCCAGCCGCUGAGGACCAUGUCGGUGGCCUGGCUGAGAUG___GGCUGCCUGCUGGGCGGCGGCCUGGGUGGUGAGCUUCUUGAUGGCCGU .(((.........((((((((((......))))))))))..((......)))))((((.((((.........(((((((.((...))))))))).........)))).......)))).. (-42.35 = -41.52 + -0.83)



| Location | 20,206,139 – 20,206,259 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 83.50 |
| Mean single sequence MFE | -59.98 |
| Consensus MFE | -40.21 |
| Energy contribution | -40.47 |
| Covariance contribution | 0.25 |
| Combinations/Pair | 1.22 |
| Mean z-score | -2.34 |
| Structure conservation index | 0.67 |
| SVM decision value | 0.23 |
| SVM RNA-class probability | 0.643442 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 20206139 120 - 22224390 UGGGCAGAGAGACAGUUGCCGGCUGCGCGUAGAGCUAGCUCCUGCAGCUCCGAGAUCAUAGAACGCUGGCAAUCCAAACGCGCCUGCUGCGAAGGUGGCUGGCUCCAGAGCCAGCCGCUC .((((......((..((((.(((.((((((.(((((.((....))))))).(.(((..(((....)))...))))..))))))..)))))))..))((((((((....)))))))))))) ( -52.30) >DroPse_CAF1 3563 120 - 1 UGGGCGGAGAGGCAGUUGCCGGCUGCCCGCAGUGCCAGUUCCUGCAGCUCUGCGAUCAGUGUGCGCUGGCAGUCCUGCUUGGCUGUCAGGGUGGGUGGCUGGCUCCACUGCCAGCUGCUG ..(((((...((((((.((((((..(((((..((.(((((...((((..((((...((((....))))))))..))))..))))).))..)))))..))))))...))))))..))))). ( -67.70) >DroSim_CAF1 34019 120 - 1 UGGGCAGAGAGGCAGUUGCGGGCUGCGCGCAGAGCCAGCUCCUGCAGCUCCGAGAUCAUAGAACGUUGGCAGUCCAGACGCGCCUGCUGCGAAGGUGGCUGGCUCCAGAGCCAGCCGCUC ...((((..((((.(((..(((((((((((((((....)).)))).))........((........))))))))).)))..)))).))))...(((((((((((....))))))))))). ( -58.80) >DroEre_CAF1 22574 120 - 1 UGAGCAGAGAGGCAGUUGCCGGCUGCGCGCAGGGCCAGCUCCUGCAGCUCCGAGAUCAUGGAACGCUGGCAAUCCAGGCGCGCCUGCUGCGACGGUGGCUGGCUCCAGAGCCAGCCGCUG ...((((...(((....)))..)))).(((((.(((((((((.....(.....).....)))..))))))....((((....))))))))).((((((((((((....)))))))))))) ( -62.50) >DroYak_CAF1 24589 120 - 1 UGUGCAGAGAGGCAGUUGCCGGCUGCGCGCAAAGCCAACUCCUGCAACUCCGAGAUCAUGGAACGCUGGCAAUCCAGGCGCGCCUGCUGCGACGGGGGCUGGCUCCAAAGCCAGCCGCUA .((((((..((((.(((((.((.((.((.....))))...)).)))))((((......)))).((((((....))).))).)))).)))).))((.((((((((....)))))))).)). ( -50.90) >DroPer_CAF1 2539 120 - 1 UGGGCGGAGAGGCAGUUGCCGGCUGCCCGCAGUGCCAGUUCCUGCAGCUCUGCGAUCAGUGUGCGCUGGCAGUCCUGCUUGGCUGUCAGGGUGGGUGGCUGGCUCCACUGCCAGCUGCUG ..(((((...((((((.((((((..(((((..((.(((((...((((..((((...((((....))))))))..))))..))))).))..)))))..))))))...))))))..))))). ( -67.70) >consensus UGGGCAGAGAGGCAGUUGCCGGCUGCGCGCAGAGCCAGCUCCUGCAGCUCCGAGAUCAUAGAACGCUGGCAAUCCAGACGCGCCUGCUGCGAAGGUGGCUGGCUCCAGAGCCAGCCGCUG (((.(.(((..((((..((.((((........)))).))..))))..))).)...)))......((.(((...........))).))......((((((((((......)))))))))). (-40.21 = -40.47 + 0.25)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 12:56:20 2006