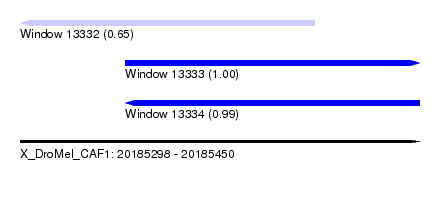
| Sequence ID | X_DroMel_CAF1 |
|---|---|
| Location | 20,185,298 – 20,185,450 |
| Length | 152 |
| Max. P | 0.999898 |

| Location | 20,185,298 – 20,185,410 |
|---|---|
| Length | 112 |
| Sequences | 5 |
| Columns | 112 |
| Reading direction | reverse |
| Mean pairwise identity | 89.91 |
| Mean single sequence MFE | -41.28 |
| Consensus MFE | -33.16 |
| Energy contribution | -33.84 |
| Covariance contribution | 0.68 |
| Combinations/Pair | 1.15 |
| Mean z-score | -1.49 |
| Structure conservation index | 0.80 |
| SVM decision value | 0.24 |
| SVM RNA-class probability | 0.650782 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 20185298 112 - 22224390 CGGGCGGUAUGAGCAAUUUCGCAUUCCUCAUACGCCGCGUGGGUCGCAAAGGGAGUGGGGUAGGUGCAAGGUAAGAAAUGCGGGUGCCACUUUAAGCUUGCGUCCACAAAAC ..((((.((((((.(((.....))).))))))))))..(((((.((((...((..((((((.((..(...(((.....)))..)..))))))))..)))))))))))..... ( -39.50) >DroSec_CAF1 6957 111 - 1 CGGGCGGUAUGAGUAAUUUCGCAUUCCUCAUACGCCGCGUGGGCCGCAAAGAG-GCAGGGCAGCUGCAAGGUAAGAAGUGCGGGUGCCACUUUAAGCUUGCGUCCACAAAAC ..((((.((((((.(((.....))).))))))))))..((((((.((((((((-....((((.(((((..........))))).)))).))))....))))))))))..... ( -43.10) >DroSim_CAF1 6524 112 - 1 CGGGCGGUAUGAGCAAUUUCGCAUUCCUCAUACGCCGCGUGGGUCGCAAAGAGAGUGGGGCAGCUGCAAGGUAAGAAGUGCGGGUGCCACUUUAAGCUUGCGUCCACAAAAC ..((((.((((((.(((.....))).))))))))))..(((((.(((((..(((((..((((.(((((..........))))).)))))))))....))))))))))..... ( -44.40) >DroEre_CAF1 7238 110 - 1 CGGGCGGUAUGAGCAAUUUCGCAUUCCUCAUACGCCGCGUGGGUUGCAAGGGGAGUGGG-CAACUGCAAGGUAGGAAAUGCGGGUGCCACUUUAAC-UUGCGUCCCCAAAAC ((((((.((((((.(((.....))).)))))))))).))((((.((((((..((((((.-((.(((((..........))))).))))))))...)-)))))..)))).... ( -43.50) >DroYak_CAF1 6184 112 - 1 CGGGCGGUAUGAGCAAUUUCGCAUUCCUCAUACGCCGCGUGGGUCGCAAGCAGAAUGGGGGCACUGCCAGGGAAGAAAUGCGGGUGCCACUUUAAGUUUGCGUUCGAAAAAC ((((((.((((((.(((.....))).)))))))))).))((((.(((((((.....(((((((((((............)).)))))).)))...)))))))))))...... ( -35.90) >consensus CGGGCGGUAUGAGCAAUUUCGCAUUCCUCAUACGCCGCGUGGGUCGCAAAGAGAGUGGGGCAGCUGCAAGGUAAGAAAUGCGGGUGCCACUUUAAGCUUGCGUCCACAAAAC ..((((.((((((.(((.....))).))))))))))..(((((.(((((.......((((((.(((((..........))))).)))).))......))))))))))..... (-33.16 = -33.84 + 0.68)



| Location | 20,185,338 – 20,185,450 |
|---|---|
| Length | 112 |
| Sequences | 5 |
| Columns | 112 |
| Reading direction | forward |
| Mean pairwise identity | 93.12 |
| Mean single sequence MFE | -44.78 |
| Consensus MFE | -40.46 |
| Energy contribution | -40.70 |
| Covariance contribution | 0.24 |
| Combinations/Pair | 1.03 |
| Mean z-score | -2.82 |
| Structure conservation index | 0.90 |
| SVM decision value | 4.44 |
| SVM RNA-class probability | 0.999898 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 20185338 112 + 22224390 ACCUUGCACCUACCCCACUCCCUUUGCGACCCACGCGGCGUAUGAGGAAUGCGAAAUUGCUCAUACCGCCCGUGGUCCCGCAUCUGAGCAACUGGGUGACAGUUGAUUUGCC .....(((.........(((....((((..(((((.((((((((((.(((.....))).)))))).)))))))))...))))...)))((((((.....))))))...))). ( -42.50) >DroSec_CAF1 6997 111 + 1 ACCUUGCAGCUGCCCUGC-CUCUUUGCGGCCCACGCGGCGUAUGAGGAAUGCGAAAUUACUCAUACCGCCCGUGGUCCCGCAUCUGAGCAACUGGGUGACAGUUGAUUUGCC ......((((((((((((-.((..(((((.(((((.((((((((((.(((.....))).)))))).)))))))))..)))))...)))))...)))...))))))....... ( -47.00) >DroSim_CAF1 6564 112 + 1 ACCUUGCAGCUGCCCCACUCUCUUUGCGACCCACGCGGCGUAUGAGGAAUGCGAAAUUGCUCAUACCGCCCGUGGUCCCGCAUCUGAGCAACUGGGUGACAGUUGAUUUGCC ......(((((((((((..(((..((((..(((((.((((((((((.(((.....))).)))))).)))))))))...))))...)))....)))).).))))))....... ( -48.10) >DroEre_CAF1 7277 111 + 1 ACCUUGCAGUUG-CCCACUCCCCUUGCAACCCACGCGGCGUAUGAGGAAUGCGAAAUUGCUCAUACCGCCCGUGGUCCCGCAUCUGAGCAACUGGGUGACAGUUGAUUUGCC ........((..-(((((((....(((...(((((.((((((((((.(((.....))).)))))).)))))))))....)))...)))....))))..))............ ( -42.00) >DroYak_CAF1 6224 112 + 1 CCCUGGCAGUGCCCCCAUUCUGCUUGCGACCCACGCGGCGUAUGAGGAAUGCGAAAUUGCUCAUACCGCCCGUGGUCCCGCAUCUGAGCAACUGGGUGACAGUUGAUUUGCC ....(((((........(((....((((..(((((.((((((((((.(((.....))).)))))).)))))))))...))))...)))((((((.....))))))..))))) ( -44.30) >consensus ACCUUGCAGCUGCCCCACUCUCUUUGCGACCCACGCGGCGUAUGAGGAAUGCGAAAUUGCUCAUACCGCCCGUGGUCCCGCAUCUGAGCAACUGGGUGACAGUUGAUUUGCC .....(((.........(((....((((..(((((.((((((((((.(((.....))).)))))).)))))))))...))))...)))((((((.....))))))...))). (-40.46 = -40.70 + 0.24)



| Location | 20,185,338 – 20,185,450 |
|---|---|
| Length | 112 |
| Sequences | 5 |
| Columns | 112 |
| Reading direction | reverse |
| Mean pairwise identity | 93.12 |
| Mean single sequence MFE | -45.40 |
| Consensus MFE | -40.18 |
| Energy contribution | -40.50 |
| Covariance contribution | 0.32 |
| Combinations/Pair | 1.10 |
| Mean z-score | -2.00 |
| Structure conservation index | 0.89 |
| SVM decision value | 2.52 |
| SVM RNA-class probability | 0.994888 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 20185338 112 - 22224390 GGCAAAUCAACUGUCACCCAGUUGCUCAGAUGCGGGACCACGGGCGGUAUGAGCAAUUUCGCAUUCCUCAUACGCCGCGUGGGUCGCAAAGGGAGUGGGGUAGGUGCAAGGU .(((......(((.(.((((.((.((....(((((..(((((((((.((((((.(((.....))).)))))))))).))))).))))).)).)).)))))))).)))..... ( -43.30) >DroSec_CAF1 6997 111 - 1 GGCAAAUCAACUGUCACCCAGUUGCUCAGAUGCGGGACCACGGGCGGUAUGAGUAAUUUCGCAUUCCUCAUACGCCGCGUGGGCCGCAAAGAG-GCAGGGCAGCUGCAAGGU ((((.......))))((((((((((((.(.(((((..(((((((((.((((((.(((.....))).)))))))))).))))).))))).....-.).)))))))))...))) ( -47.30) >DroSim_CAF1 6564 112 - 1 GGCAAAUCAACUGUCACCCAGUUGCUCAGAUGCGGGACCACGGGCGGUAUGAGCAAUUUCGCAUUCCUCAUACGCCGCGUGGGUCGCAAAGAGAGUGGGGCAGCUGCAAGGU ((((.......))))((((((((((((...(((((..(((((((((.((((((.(((.....))).)))))))))).))))).))))).........)))))))))...))) ( -44.80) >DroEre_CAF1 7277 111 - 1 GGCAAAUCAACUGUCACCCAGUUGCUCAGAUGCGGGACCACGGGCGGUAUGAGCAAUUUCGCAUUCCUCAUACGCCGCGUGGGUUGCAAGGGGAGUGGG-CAACUGCAAGGU ((((.......))))(((((((((((((..(((((..(((((((((.((((((.(((.....))).)))))))))).))))).))))).......))))-))))))...))) ( -43.90) >DroYak_CAF1 6224 112 - 1 GGCAAAUCAACUGUCACCCAGUUGCUCAGAUGCGGGACCACGGGCGGUAUGAGCAAUUUCGCAUUCCUCAUACGCCGCGUGGGUCGCAAGCAGAAUGGGGGCACUGCCAGGG ((((.......((((.((((.(((((....(((((..(((((((((.((((((.(((.....))).)))))))))).))))).))))))))))..)))))))).)))).... ( -47.70) >consensus GGCAAAUCAACUGUCACCCAGUUGCUCAGAUGCGGGACCACGGGCGGUAUGAGCAAUUUCGCAUUCCUCAUACGCCGCGUGGGUCGCAAAGAGAGUGGGGCAGCUGCAAGGU ((((.......))))((((((((((((...(((((..(((((((((.((((((.(((.....))).)))))))))).))))).))))).........)))))))))...))) (-40.18 = -40.50 + 0.32)


Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 12:56:01 2006