
| Sequence ID | X_DroMel_CAF1 |
|---|---|
| Location | 20,180,695 – 20,180,955 |
| Length | 260 |
| Max. P | 0.999488 |

| Location | 20,180,695 – 20,180,803 |
|---|---|
| Length | 108 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 91.10 |
| Mean single sequence MFE | -47.04 |
| Consensus MFE | -36.32 |
| Energy contribution | -36.48 |
| Covariance contribution | 0.16 |
| Combinations/Pair | 1.03 |
| Mean z-score | -2.32 |
| Structure conservation index | 0.77 |
| SVM decision value | 1.35 |
| SVM RNA-class probability | 0.944669 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 20180695 108 + 22224390 ------UCCCACUGCCGC------AGCCCUGCUCGAUUGCAAAGACAAUGGAUGCAGGCGAGCGGAGCCGAUGGGAUUGGAUCGGGACAGACAGCUGGCAUCCACUGGGAAGCCAGACAG ------((((.((.((((------.(((.(((((.((((......)))).)).))))))..))))))(((((...)))))...))))......((((((.(((....))).))))).).. ( -43.10) >DroSec_CAF1 2365 114 + 1 ACCGCAUCCCGCUGCCGC------AGCCCUGCUCGAUUGCAAAGACAAUGGAUGCAGGCGAGCGGAGCCGAUGGGAUUGGAUCGGGACAGACAGCUGGCAUCCACUGGGAAGCCAGACAG .(((.((((((((.((((------.(((.(((((.((((......)))).)).))))))..)))))))....)))))))).............((((((.(((....))).))))).).. ( -47.30) >DroSim_CAF1 1783 114 + 1 UCCGCGUCCCGCUGCCGC------AGCCCUGCUCGAUUGCAAAGACAAUGGAUGCAGGCGAGCGGAGCCGAUGGGAUUGGAUCGGGACAGACAUCUGGCAUCCACUGGGAAGCCAGACAG .....((((((((.((((------.(((.(((((.((((......)))).)).))))))..))))))).(((........)))))))).....((((((.(((....))).))))))... ( -50.50) >DroEre_CAF1 785 114 + 1 ------UUCCACUUCCACUUGCAGAGCCCUGCUCGAUUGCAAAGACAAUAGAUGCAGGCGAGCGGAGGCGAUGGGAUUGGACCGGGACAGACAGCUGGCAUCCACUGGGAAGCCAGACAG ------.(((...((((.((.((..((((((((((.(((((...........))))).))))))).)))..)).)).))))...)))......((((((.(((....))).))))).).. ( -48.60) >DroYak_CAF1 2362 108 + 1 ------UUCCACUGCCGC------AGCUCUGCUCGAUUGCAAAGACAAUGGAUGCAGGCGAGCGGAGCCGAUGGGAUUGGAUCGGGACAGACAGCUGGCAUCCACUGGGAAGCCAGACAG ------.((((.(.((((------.((((((((((.(((((...........))))).)))))))))).).))).).))))............((((((.(((....))).))))).).. ( -45.70) >consensus ______UCCCACUGCCGC______AGCCCUGCUCGAUUGCAAAGACAAUGGAUGCAGGCGAGCGGAGCCGAUGGGAUUGGAUCGGGACAGACAGCUGGCAUCCACUGGGAAGCCAGACAG ......((((.((.((((.......((.((((((.((((......)))).)).))))))..))))))(((((...)))))...)))).......(((((.(((....))).))))).... (-36.32 = -36.48 + 0.16)



| Location | 20,180,695 – 20,180,803 |
|---|---|
| Length | 108 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 91.10 |
| Mean single sequence MFE | -43.60 |
| Consensus MFE | -36.42 |
| Energy contribution | -36.46 |
| Covariance contribution | 0.04 |
| Combinations/Pair | 1.09 |
| Mean z-score | -1.74 |
| Structure conservation index | 0.84 |
| SVM decision value | 1.15 |
| SVM RNA-class probability | 0.922785 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 20180695 108 - 22224390 CUGUCUGGCUUCCCAGUGGAUGCCAGCUGUCUGUCCCGAUCCAAUCCCAUCGGCUCCGCUCGCCUGCAUCCAUUGUCUUUGCAAUCGAGCAGGGCU------GCGGCAGUGGGA------ ..(.(((((.(((....))).))))))......(((((((........))).(((((((...(((((.((.(((((....))))).)))))))...------)))).)))))))------ ( -40.40) >DroSec_CAF1 2365 114 - 1 CUGUCUGGCUUCCCAGUGGAUGCCAGCUGUCUGUCCCGAUCCAAUCCCAUCGGCUCCGCUCGCCUGCAUCCAUUGUCUUUGCAAUCGAGCAGGGCU------GCGGCAGCGGGAUGCGGU ....(((((.(((....))).)))))((((..((((((((........))).(((((((...(((((.((.(((((....))))).)))))))...------)))).)))))))))))). ( -47.10) >DroSim_CAF1 1783 114 - 1 CUGUCUGGCUUCCCAGUGGAUGCCAGAUGUCUGUCCCGAUCCAAUCCCAUCGGCUCCGCUCGCCUGCAUCCAUUGUCUUUGCAAUCGAGCAGGGCU------GCGGCAGCGGGACGCGGA ..(((((((.(((....))).)))))))....((((((((........))).(((((((...(((((.((.(((((....))))).)))))))...------)))).))))))))..... ( -50.30) >DroEre_CAF1 785 114 - 1 CUGUCUGGCUUCCCAGUGGAUGCCAGCUGUCUGUCCCGGUCCAAUCCCAUCGCCUCCGCUCGCCUGCAUCUAUUGUCUUUGCAAUCGAGCAGGGCUCUGCAAGUGGAAGUGGAA------ ..(.(((((.(((....))).))))))......(((...((((....((..((((..(((((..((((...........))))..))))).))))..))....))))...))).------ ( -39.20) >DroYak_CAF1 2362 108 - 1 CUGUCUGGCUUCCCAGUGGAUGCCAGCUGUCUGUCCCGAUCCAAUCCCAUCGGCUCCGCUCGCCUGCAUCCAUUGUCUUUGCAAUCGAGCAGAGCU------GCGGCAGUGGAA------ ..(.(((((.(((....))).))))))............((((...((..((((((.(((((..((((...........))))..))))).)))))------).))...)))).------ ( -41.00) >consensus CUGUCUGGCUUCCCAGUGGAUGCCAGCUGUCUGUCCCGAUCCAAUCCCAUCGGCUCCGCUCGCCUGCAUCCAUUGUCUUUGCAAUCGAGCAGGGCU______GCGGCAGUGGGA______ ....(((((.(((....))).)))))(((.(((((.((((........))))((((.(((((..((((...........))))..))))).)))).........))))))))........ (-36.42 = -36.46 + 0.04)



| Location | 20,180,723 – 20,180,840 |
|---|---|
| Length | 117 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 93.74 |
| Mean single sequence MFE | -44.70 |
| Consensus MFE | -39.02 |
| Energy contribution | -39.50 |
| Covariance contribution | 0.48 |
| Combinations/Pair | 1.03 |
| Mean z-score | -1.16 |
| Structure conservation index | 0.87 |
| SVM decision value | -0.06 |
| SVM RNA-class probability | 0.500000 |
| Prediction | OTHER |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 20180723 117 + 22224390 AAAGACAAUGGAUGCAGGCGAGCGGAGCCGAUGGGAUUGGAUCGGGACAGACAGCUGGCAUCCACUGGGAAGCCAGACAGCUGGUACUUUCCAGGACC---ACCCCGAUGCUGCCGCCCC ................((((.((((...((.(((..(((((..(...(((....(((((.(((....))).)))))....)))...)..)))))..))---)...))...)))))))).. ( -44.10) >DroSec_CAF1 2399 117 + 1 AAAGACAAUGGAUGCAGGCGAGCGGAGCCGAUGGGAUUGGAUCGGGACAGACAGCUGGCAUCCACUGGGAAGCCAGACAGCUGGUACUUUCCAGGACC---AACCCGAAGCUGCCGCCCC ................((((.((((...((.(((..(((((..(...(((....(((((.(((....))).)))))....)))...)..)))))..))---)...))...)))))))).. ( -44.10) >DroSim_CAF1 1817 117 + 1 AAAGACAAUGGAUGCAGGCGAGCGGAGCCGAUGGGAUUGGAUCGGGACAGACAUCUGGCAUCCACUGGGAAGCCAGACAGCUGGUACUUUCCAGGACC---AACCCGAAGCUGCCGCCCC ................((((.((((...((.(((..(((((..(...(((...((((((.(((....))).))))))...)))...)..)))))..))---)...))...)))))))).. ( -45.00) >DroEre_CAF1 819 117 + 1 AAAGACAAUAGAUGCAGGCGAGCGGAGGCGAUGGGAUUGGACCGGGACAGACAGCUGGCAUCCACUGGGAAGCCAGACAGCUGGGACUUCCCAGGAGC---AUACCGAAGCUGCCGCCCC ................((.(.((((.(((..(((...((((((((.........))))..))))((((((((((((....))))..))))))))....---...)))..))).))))))) ( -41.80) >DroYak_CAF1 2390 120 + 1 AAAGACAAUGGAUGCAGGCGAGCGGAGCCGAUGGGAUUGGAUCGGGACAGACAGCUGGCAUCCACUGGGAAGCCAGACAGCUGGGACAACCCAGGAGCGCCAUCCCGGAGCUGCCGCCCC ................((((.((((..((...(((((.((.((..........((((((.(((....))).))))).)..(((((....)))))))...)))))))))..)))))))).. ( -48.50) >consensus AAAGACAAUGGAUGCAGGCGAGCGGAGCCGAUGGGAUUGGAUCGGGACAGACAGCUGGCAUCCACUGGGAAGCCAGACAGCUGGUACUUUCCAGGACC___AACCCGAAGCUGCCGCCCC ................((((.((((..(((((...))))).(((((........(((((.(((....))).)))))....((((......)))).........)))))..)))))))).. (-39.02 = -39.50 + 0.48)

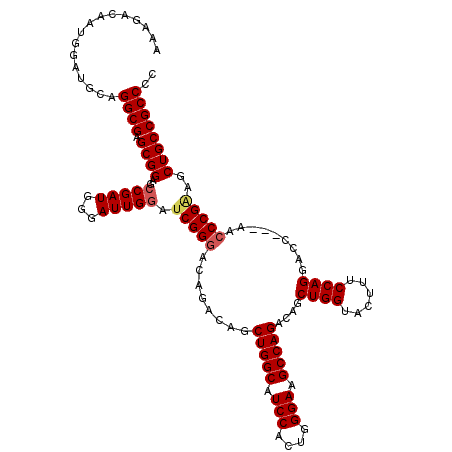
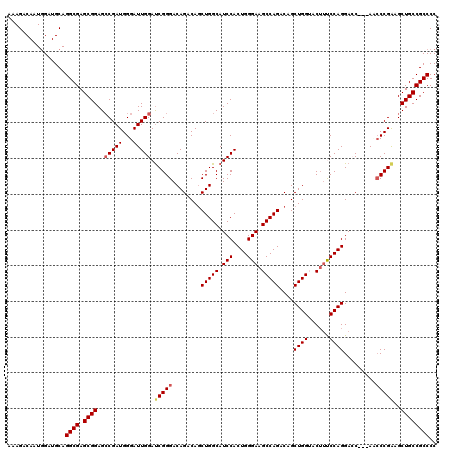
| Location | 20,180,723 – 20,180,840 |
|---|---|
| Length | 117 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 93.74 |
| Mean single sequence MFE | -46.98 |
| Consensus MFE | -37.40 |
| Energy contribution | -37.52 |
| Covariance contribution | 0.12 |
| Combinations/Pair | 1.08 |
| Mean z-score | -1.72 |
| Structure conservation index | 0.80 |
| SVM decision value | 0.53 |
| SVM RNA-class probability | 0.771488 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 20180723 117 - 22224390 GGGGCGGCAGCAUCGGGGU---GGUCCUGGAAAGUACCAGCUGUCUGGCUUCCCAGUGGAUGCCAGCUGUCUGUCCCGAUCCAAUCCCAUCGGCUCCGCUCGCCUGCAUCCAUUGUCUUU .((((((.(((...(((((---((..(.(((.((...(((((....(((.(((....))).)))))))).)).))).)..)).)))))....)))))))))................... ( -43.60) >DroSec_CAF1 2399 117 - 1 GGGGCGGCAGCUUCGGGUU---GGUCCUGGAAAGUACCAGCUGUCUGGCUUCCCAGUGGAUGCCAGCUGUCUGUCCCGAUCCAAUCCCAUCGGCUCCGCUCGCCUGCAUCCAUUGUCUUU .((((((.((((..(((((---((..(.(((.((...(((((....(((.(((....))).)))))))).)).))).)..)))))))....))))))))))................... ( -45.00) >DroSim_CAF1 1817 117 - 1 GGGGCGGCAGCUUCGGGUU---GGUCCUGGAAAGUACCAGCUGUCUGGCUUCCCAGUGGAUGCCAGAUGUCUGUCCCGAUCCAAUCCCAUCGGCUCCGCUCGCCUGCAUCCAUUGUCUUU .((((((.((((..(((((---((..(.(((......(((.((((((((.(((....))).)))))))).)))))).)..)))))))....))))))))))................... ( -46.90) >DroEre_CAF1 819 117 - 1 GGGGCGGCAGCUUCGGUAU---GCUCCUGGGAAGUCCCAGCUGUCUGGCUUCCCAGUGGAUGCCAGCUGUCUGUCCCGGUCCAAUCCCAUCGCCUCCGCUCGCCUGCAUCUAUUGUCUUU ((((((((((((..(((((---.(..((((((((..((((....))))))))))))..))))))))))).)))))))(((...........)))...((......))............. ( -48.50) >DroYak_CAF1 2390 120 - 1 GGGGCGGCAGCUCCGGGAUGGCGCUCCUGGGUUGUCCCAGCUGUCUGGCUUCCCAGUGGAUGCCAGCUGUCUGUCCCGAUCCAAUCCCAUCGGCUCCGCUCGCCUGCAUCCAUUGUCUUU ((((((((((((..((((..((.(....).))..)))))))))))..))))).((((((((((..((.((..(..(((((........)))))..).))..))..))))))))))..... ( -50.90) >consensus GGGGCGGCAGCUUCGGGAU___GGUCCUGGAAAGUACCAGCUGUCUGGCUUCCCAGUGGAUGCCAGCUGUCUGUCCCGAUCCAAUCCCAUCGGCUCCGCUCGCCUGCAUCCAUUGUCUUU (((((((((((((((((........)))))........)))))))..))))).((((((((((.((.((..((..(((((........)))))...))..)).))))))))))))..... (-37.40 = -37.52 + 0.12)

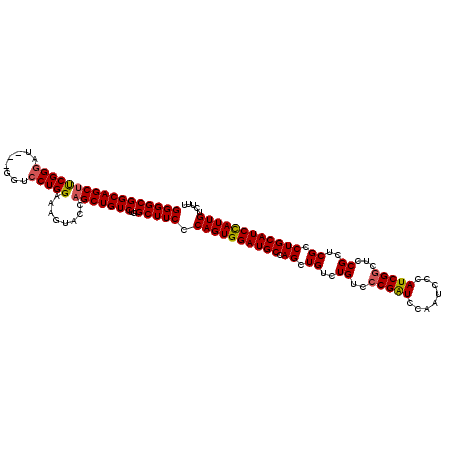
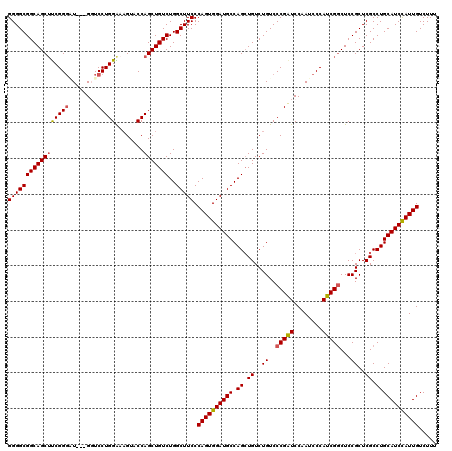
| Location | 20,180,763 – 20,180,880 |
|---|---|
| Length | 117 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 93.91 |
| Mean single sequence MFE | -42.28 |
| Consensus MFE | -38.36 |
| Energy contribution | -38.56 |
| Covariance contribution | 0.20 |
| Combinations/Pair | 1.00 |
| Mean z-score | -1.27 |
| Structure conservation index | 0.91 |
| SVM decision value | 0.56 |
| SVM RNA-class probability | 0.782550 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 20180763 117 + 22224390 AUCGGGACAGACAGCUGGCAUCCACUGGGAAGCCAGACAGCUGGUACUUUCCAGGACC---ACCCCGAUGCUGCCGCCCCGUUGUAAAUCUCUUCACAUGUGCCCAGGUGGGCAACUUGU ...((((...(((((.(((.....((((....)))).(((((((......)))((...---...))...))))..)))..)))))...))))...(((..(((((....)))))...))) ( -39.70) >DroSec_CAF1 2439 117 + 1 AUCGGGACAGACAGCUGGCAUCCACUGGGAAGCCAGACAGCUGGUACUUUCCAGGACC---AACCCGAAGCUGCCGCCCCGUUGUAAAUCUCCUCACAUGUGCCCAGGUGGGCAACUUGU ..((((.(.(.(((((((......((((((((((((....))))..))))))))....---..))...)))))).).))))..............(((..(((((....)))))...))) ( -40.40) >DroSim_CAF1 1857 117 + 1 AUCGGGACAGACAUCUGGCAUCCACUGGGAAGCCAGACAGCUGGUACUUUCCAGGACC---AACCCGAAGCUGCCGCCCCGUUGUAAAUCUCCUCACAUGUGCCCAGGUGGGCAACUUGU ..((((.(.....((((((.(((....))).))))))(((((((......)).((...---...))..)))))..).))))..............(((..(((((....)))))...))) ( -40.10) >DroEre_CAF1 859 117 + 1 ACCGGGACAGACAGCUGGCAUCCACUGGGAAGCCAGACAGCUGGGACUUCCCAGGAGC---AUACCGAAGCUGCCGCCCCGUUGUAAAUCUCUUCACAUGUGCCCAGGUGGGCAACUUGU ...((((...(((((.(((.....((((((((((((....))))..)))))))).(((---........)))...)))..)))))...))))...(((..(((((....)))))...))) ( -45.40) >DroYak_CAF1 2430 120 + 1 AUCGGGACAGACAGCUGGCAUCCACUGGGAAGCCAGACAGCUGGGACAACCCAGGAGCGCCAUCCCGGAGCUGCCGCCCCGUUGUAAAUCUCUUCACAUGUGCCCAGGUGGGCAACUUGU ...((((...(((((((((.(((.((((....)))).....((((....)))))))..))))....((.((....)).)))))))...))))...(((..(((((....)))))...))) ( -45.80) >consensus AUCGGGACAGACAGCUGGCAUCCACUGGGAAGCCAGACAGCUGGUACUUUCCAGGACC___AACCCGAAGCUGCCGCCCCGUUGUAAAUCUCUUCACAUGUGCCCAGGUGGGCAACUUGU ...((((...(((((.(((.....((((....)))).(((((((......)))((.........))...))))..)))..)))))...))))...(((..(((((....)))))...))) (-38.36 = -38.56 + 0.20)


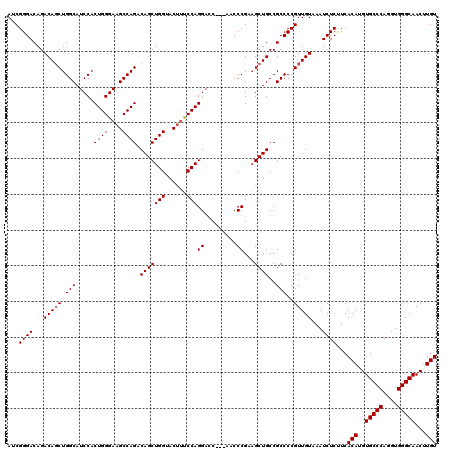
| Location | 20,180,763 – 20,180,880 |
|---|---|
| Length | 117 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 93.91 |
| Mean single sequence MFE | -55.76 |
| Consensus MFE | -46.86 |
| Energy contribution | -46.90 |
| Covariance contribution | 0.04 |
| Combinations/Pair | 1.08 |
| Mean z-score | -3.60 |
| Structure conservation index | 0.84 |
| SVM decision value | 3.65 |
| SVM RNA-class probability | 0.999488 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 20180763 117 - 22224390 ACAAGUUGCCCACCUGGGCACAUGUGAAGAGAUUUACAACGGGGCGGCAGCAUCGGGGU---GGUCCUGGAAAGUACCAGCUGUCUGGCUUCCCAGUGGAUGCCAGCUGUCUGUCCCGAU ....((.((((....)))))).((((((....)))))).((((((((((((....((.(---(((.((....)).)))).))...((((.(((....))).)))))))).)))))))).. ( -53.40) >DroSec_CAF1 2439 117 - 1 ACAAGUUGCCCACCUGGGCACAUGUGAGGAGAUUUACAACGGGGCGGCAGCUUCGGGUU---GGUCCUGGAAAGUACCAGCUGUCUGGCUUCCCAGUGGAUGCCAGCUGUCUGUCCCGAU ....((.((((....)))))).((((((....)))))).((((((((((((....((((---(((.((....)).)))))))...((((.(((....))).)))))))).)))))))).. ( -54.50) >DroSim_CAF1 1857 117 - 1 ACAAGUUGCCCACCUGGGCACAUGUGAGGAGAUUUACAACGGGGCGGCAGCUUCGGGUU---GGUCCUGGAAAGUACCAGCUGUCUGGCUUCCCAGUGGAUGCCAGAUGUCUGUCCCGAU ....((.((((....)))))).((((((....)))))).(((((((((.......((((---(((.((....)).)))))))(((((((.(((....))).)))))))).)))))))).. ( -53.10) >DroEre_CAF1 859 117 - 1 ACAAGUUGCCCACCUGGGCACAUGUGAAGAGAUUUACAACGGGGCGGCAGCUUCGGUAU---GCUCCUGGGAAGUCCCAGCUGUCUGGCUUCCCAGUGGAUGCCAGCUGUCUGUCCCGGU ....((.((((....)))))).((((((....)))))).(((((((((((((..(((((---.(..((((((((..((((....))))))))))))..))))))))))).)))))))).. ( -60.40) >DroYak_CAF1 2430 120 - 1 ACAAGUUGCCCACCUGGGCACAUGUGAAGAGAUUUACAACGGGGCGGCAGCUCCGGGAUGGCGCUCCUGGGUUGUCCCAGCUGUCUGGCUUCCCAGUGGAUGCCAGCUGUCUGUCCCGAU ....((.((((....)))))).((((((....)))))).((((((((((((...((((..((.(....).))..)))).......((((.(((....))).)))))))).)))))))).. ( -57.40) >consensus ACAAGUUGCCCACCUGGGCACAUGUGAAGAGAUUUACAACGGGGCGGCAGCUUCGGGAU___GGUCCUGGAAAGUACCAGCUGUCUGGCUUCCCAGUGGAUGCCAGCUGUCUGUCCCGAU ....((.((((....)))))).((((((....)))))).((((((((((((((((((........))))))..............((((.(((....))).)))))))).)))))))).. (-46.86 = -46.90 + 0.04)

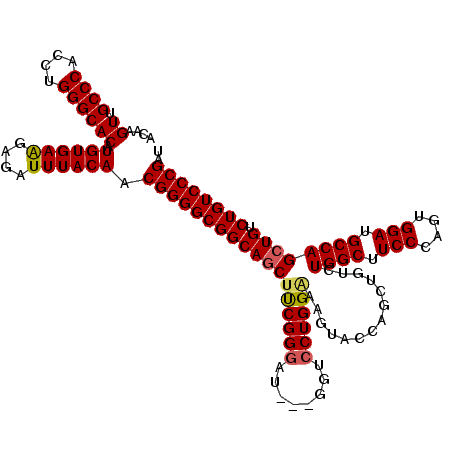

| Location | 20,180,840 – 20,180,955 |
|---|---|
| Length | 115 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 96.41 |
| Mean single sequence MFE | -41.44 |
| Consensus MFE | -40.54 |
| Energy contribution | -40.50 |
| Covariance contribution | -0.04 |
| Combinations/Pair | 1.03 |
| Mean z-score | -1.60 |
| Structure conservation index | 0.98 |
| SVM decision value | 2.17 |
| SVM RNA-class probability | 0.989533 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 20180840 115 - 22224390 -----CGGCGGUCGGGAGAACCAUGAACCGAUUUGCCAUGUGUCAGGUGGAUGAAGCGCAGCAGCUACGGAGCCACGUUAACAAGUUGCCCACCUGGGCACAUGUGAAGAGAUUUACAAC -----.(((((((((............)))).)))))(((((((((((((.((.....))(((((((((......))).....)))))))))))).)))))))(((((....)))))... ( -41.40) >DroSec_CAF1 2516 115 - 1 -----CGGCGGUCGGGAGAACCAUGAACCGAUUUGCCAUGUGUCAGGUGGAUGAAGCGCAGCAGCUACGAAGCCACGUUAACAAGUUGCCCACCUGGGCACAUGUGAGGAGAUUUACAAC -----.(((((((((............)))).)))))(((((((((((((.((.....))(((((((((......))).....)))))))))))).)))))))(((((....)))))... ( -39.60) >DroSim_CAF1 1934 115 - 1 -----CGGCGGUCGGGAGAACCAUGAACCGAUUUGCCAUGUGUCAGGUGGAUGAAGCGCAGCAGCUACGGAGCCACGUUAACAAGUUGCCCACCUGGGCACAUGUGAGGAGAUUUACAAC -----.(((((((((............)))).)))))(((((((((((((.((.....))(((((((((......))).....)))))))))))).)))))))(((((....)))))... ( -40.90) >DroEre_CAF1 936 120 - 1 GGCGUCGGCGGUCGGGAGAACCAUGAACCGAUUUGCCAUGUGUCAGGUGGAUGAAGCGCAGCAGCUCCGGAGCCACGUUAACAAGUUGCCCACCUGGGCACAUGUGAAGAGAUUUACAAC ((((....((((((((....)).)).))))...))))(((((((((((((.((.(((......))).))..((.((........)).)))))))).)))))))(((((....)))))... ( -41.20) >DroYak_CAF1 2510 115 - 1 -----CGGCGGUCGGGAUAACCAUGAACCGAUUUGCCAUGUGUCAGGUGGAUGAAGCGCAGCAGCUACGGAGCAACGUUAACAAGUUGCCCACCUGGGCACAUGUGAAGAGAUUUACAAC -----.(((((((((............)))).)))))(((((((((((((.((.(((......))).))..(((((........))))))))))).)))))))(((((....)))))... ( -44.10) >consensus _____CGGCGGUCGGGAGAACCAUGAACCGAUUUGCCAUGUGUCAGGUGGAUGAAGCGCAGCAGCUACGGAGCCACGUUAACAAGUUGCCCACCUGGGCACAUGUGAAGAGAUUUACAAC ......(((((((((............)))).)))))(((((((((((((.((.....))(((((((((......))).....)))))))))))).)))))))(((((....)))))... (-40.54 = -40.50 + -0.04)


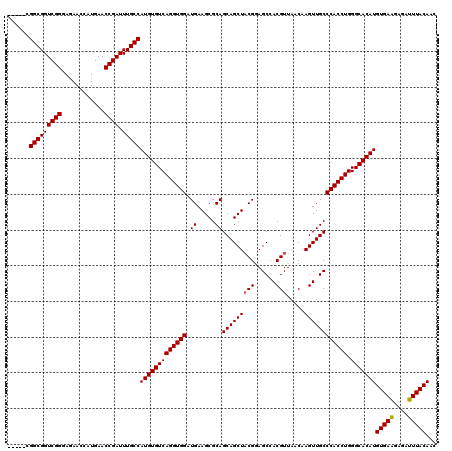
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 12:55:50 2006