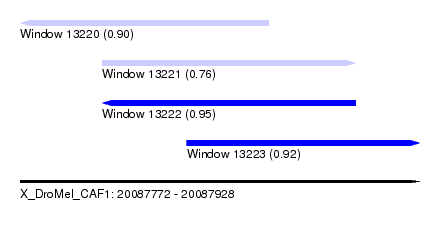
| Sequence ID | X_DroMel_CAF1 |
|---|---|
| Location | 20,087,772 – 20,087,928 |
| Length | 156 |
| Max. P | 0.946764 |

| Location | 20,087,772 – 20,087,869 |
|---|---|
| Length | 97 |
| Sequences | 5 |
| Columns | 105 |
| Reading direction | reverse |
| Mean pairwise identity | 91.84 |
| Mean single sequence MFE | -35.86 |
| Consensus MFE | -24.76 |
| Energy contribution | -24.72 |
| Covariance contribution | -0.04 |
| Combinations/Pair | 1.12 |
| Mean z-score | -3.93 |
| Structure conservation index | 0.69 |
| SVM decision value | 1.01 |
| SVM RNA-class probability | 0.899077 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 20087772 97 - 22224390 AUGUGCA-G----UAUAUACAGCUUUGAUGUGGCCUGUGUGCACACGCCACACAUGUGAGUGUG--UGCAAAAGGAUCUAAUCGAAUGCAAGGGAUA-AUAUGCA ...((((-.----(((.....((((((((....(((...((((((((((((....))).)))))--))))..))).....)))))).)).....)))-...)))) ( -35.00) >DroSec_CAF1 10205 97 - 1 AGGUGCA-G----UAUAUACAGCUUUGAUGUGGCCUGUGUGCACACGCCACACAUGUGAGUGUG--UGCGAAAGGAUCUAAUCGAAUGCAAGGGAUA-AUAUGCA ...((((-.----(((.....((((((((....(((...((((((((((((....))).)))))--))))..))).....)))))).)).....)))-...)))) ( -34.30) >DroSim_CAF1 12500 97 - 1 AGGUGCA-G----UAUAUACAGCUUUGAUGUGGACUGUGUGCACACGCCACACAUGUGAGUGUG--UGCGAAAGGAUCUAAUCGAAUGCAAGGGAUA-AUAUGCA ...((((-.----(((.....((((((((.((((((...((((((((((((....))).)))))--))))...)).)))))))))).)).....)))-...)))) ( -33.70) >DroEre_CAF1 8724 98 - 1 AUGUGCAAG----UAUAUACAGCUUUGAUGUGGCCUGUGUGCACACACCACACAUGUGAGUGUG--UGCGCAAGGACCUAAUCGAAUGCAAAGGAUA-AUAUGCA ...((((..----(((.....((((((((..(((((.((((((((((((((....))).)))))--))))))))).))..)))))).)).....)))-...)))) ( -39.60) >DroYak_CAF1 9481 105 - 1 AUGUGCAAGUAUGUACAUACAGCUUUGAUGUGGCCUGUGUGCACACGCCACACAUGUGAGUGUGUGUGCGAAAGGAUCUAAUCGAAUGCAAAGGACAUGUAUGUA (((((((.((((....)))).((((((((....(((.((..((((((((((....))).)))))))..))..))).....)))))).))........))))))). ( -36.70) >consensus AUGUGCA_G____UAUAUACAGCUUUGAUGUGGCCUGUGUGCACACGCCACACAUGUGAGUGUG__UGCGAAAGGAUCUAAUCGAAUGCAAGGGAUA_AUAUGCA ...((((......(((.....((((((((..(((((...((((((((((((....))).)))))..))))..))).))..)))))).)).....)))....)))) (-24.76 = -24.72 + -0.04)


| Location | 20,087,804 – 20,087,903 |
|---|---|
| Length | 99 |
| Sequences | 5 |
| Columns | 106 |
| Reading direction | forward |
| Mean pairwise identity | 84.96 |
| Mean single sequence MFE | -29.74 |
| Consensus MFE | -18.73 |
| Energy contribution | -20.64 |
| Covariance contribution | 1.91 |
| Combinations/Pair | 1.08 |
| Mean z-score | -2.34 |
| Structure conservation index | 0.63 |
| SVM decision value | 0.49 |
| SVM RNA-class probability | 0.756764 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 20087804 99 + 22224390 UUUGCA--CACACUCACAUGUGUGGCGUGUGCACACAGGCCACAUCAAAGCUGUAUAUA----C-UGCACAUACAUAUGUAUGUAUGUAUAUGUACUCGGUUGGGC ..((((--(((.(.(((....)))).))))))).....(((.((.(..((.((((((((----(-...((((((....))))))..)))))))))))..).))))) ( -32.40) >DroSec_CAF1 10237 97 + 1 UUCGCA--CACACUCACAUGUGUGGCGUGUGCACACAGGCCACAUCAAAGCUGUAUAUA----C-UGCACCUACAUAUG--UGUAUGUAUAUGUACUCGGUUGGGC ...(((--(((.(.(((....)))).))))))......(((.((.(..((.((((((((----(-(((((........)--)))).)))))))))))..).))))) ( -30.60) >DroSim_CAF1 12532 97 + 1 UUCGCA--CACACUCACAUGUGUGGCGUGUGCACACAGUCCACAUCAAAGCUGUAUAUA----C-UGCACCUACAUAUG--UGUACGUAUAUGUAAUCGCUCGGGU ...(((--(((.(.(((....)))).))))))...........(((..(((((((((((----(-(((((........)--)))).)))))))))...)))..))) ( -27.30) >DroEre_CAF1 8756 88 + 1 UGCGCA--CACACUCACAUGUGUGGUGUGUGCACACAGGCCACAUCAAAGCUGUAUAUA----CUUGCACAUA------------UGUAUUGGUAUUCGGUCGGGC ((.(((--(((((.(((....))))))))))).))..((((..(((((.((((((....----..))))....------------.)).)))))....)))).... ( -27.80) >DroYak_CAF1 9514 99 + 1 UUCGCACACACACUCACAUGUGUGGCGUGUGCACACAGGCCACAUCAAAGCUGUAUGUACAUACUUGCACAUAUA-------GUAUGUAUAUGUAUUCGGUCGGGC ...((((((.(((.(....).)))..)))))).....(((((((.......)))(((((((((((.........)-------))))))))))......)))).... ( -30.60) >consensus UUCGCA__CACACUCACAUGUGUGGCGUGUGCACACAGGCCACAUCAAAGCUGUAUAUA____C_UGCACAUACAUAUG__UGUAUGUAUAUGUACUCGGUCGGGC ............(((....(((((((.(((....))).)))))))....................((((.((((((((....)))))))).)))).......))). (-18.73 = -20.64 + 1.91)


| Location | 20,087,804 – 20,087,903 |
|---|---|
| Length | 99 |
| Sequences | 5 |
| Columns | 106 |
| Reading direction | reverse |
| Mean pairwise identity | 84.96 |
| Mean single sequence MFE | -32.74 |
| Consensus MFE | -21.04 |
| Energy contribution | -22.66 |
| Covariance contribution | 1.62 |
| Combinations/Pair | 1.07 |
| Mean z-score | -3.01 |
| Structure conservation index | 0.64 |
| SVM decision value | 1.37 |
| SVM RNA-class probability | 0.946764 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 20087804 99 - 22224390 GCCCAACCGAGUACAUAUACAUACAUACAUAUGUAUGUGCA-G----UAUAUACAGCUUUGAUGUGGCCUGUGUGCACACGCCACACAUGUGAGUGUG--UGCAAA (((((.(.((((..((((((.(((((((....)))))))..-)----)))))...)))).).)).))).....((((((((((((....))).)))))--)))).. ( -37.80) >DroSec_CAF1 10237 97 - 1 GCCCAACCGAGUACAUAUACAUACA--CAUAUGUAGGUGCA-G----UAUAUACAGCUUUGAUGUGGCCUGUGUGCACACGCCACACAUGUGAGUGUG--UGCGAA ..........((((...((((((..--..)))))).)))).-(----(((((((.((..((.((((((.(((....))).)))))))).))..)))))--)))... ( -32.40) >DroSim_CAF1 12532 97 - 1 ACCCGAGCGAUUACAUAUACGUACA--CAUAUGUAGGUGCA-G----UAUAUACAGCUUUGAUGUGGACUGUGUGCACACGCCACACAUGUGAGUGUG--UGCGAA ......(((.((((((((.......--.)))))))).))).-.----...((((((((.......)).))))))(((((((((((....))).)))))--)))... ( -32.80) >DroEre_CAF1 8756 88 - 1 GCCCGACCGAAUACCAAUACA------------UAUGUGCAAG----UAUAUACAGCUUUGAUGUGGCCUGUGUGCACACACCACACAUGUGAGUGUG--UGCGCA (((.......((((...(((.------------...)))...)----)))..(((.......))))))...((((((((((((((....))).)))))--)))))) ( -27.40) >DroYak_CAF1 9514 99 - 1 GCCCGACCGAAUACAUAUACAUAC-------UAUAUGUGCAAGUAUGUACAUACAGCUUUGAUGUGGCCUGUGUGCACACGCCACACAUGUGAGUGUGUGUGCGAA .......((.((((((((.(((((-------(.((((((((....)))))))).))...((.((((((.(((....))).)))))))))))).)))))))).)).. ( -33.30) >consensus GCCCGACCGAAUACAUAUACAUACA__CAUAUGUAUGUGCA_G____UAUAUACAGCUUUGAUGUGGCCUGUGUGCACACGCCACACAUGUGAGUGUG__UGCGAA .............((((((((((......))))))))))...........((((((((.......)).))))))(((((((((((....))).)))))..)))... (-21.04 = -22.66 + 1.62)


| Location | 20,087,837 – 20,087,928 |
|---|---|
| Length | 91 |
| Sequences | 5 |
| Columns | 103 |
| Reading direction | forward |
| Mean pairwise identity | 73.12 |
| Mean single sequence MFE | -31.52 |
| Consensus MFE | -13.67 |
| Energy contribution | -16.46 |
| Covariance contribution | 2.79 |
| Combinations/Pair | 1.19 |
| Mean z-score | -2.03 |
| Structure conservation index | 0.43 |
| SVM decision value | 1.13 |
| SVM RNA-class probability | 0.919859 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 20087837 91 + 22224390 CAGGCCACAUCAAAGCUGUAUAUA----C-UGCACAUACAUAUGUAUGUAUGUAUAUGUACUCGGUUGGGCGGUGGCCGUGGGCGUGGCAGCUGUG------- ..((((((.((((....(((((((----.-(((((((((....))))))..))))))))))....))))...))))))...(((......)))...------- ( -32.10) >DroSec_CAF1 10270 83 + 1 CAGGCCACAUCAAAGCUGUAUAUA----C-UGCACCUACAUAUG--UGUAUGUAUAUGUACUCGGUUGGGCGGGGGCUGGGGGCGUGGCC------------- ..((((((.((..((((.((((((----(-(((((........)--)))).)))))))..((((......))))))))..))..))))))------------- ( -31.60) >DroSim_CAF1 12565 83 + 1 CAGUCCACAUCAAAGCUGUAUAUA----C-UGCACCUACAUAUG--UGUACGUAUAUGUAAUCGCUCGGGUGUGGUCUCGGGGCUUGGCC------------- .((.(((((((..(((((((((((----(-(((((........)--)))).)))))))))...)))..))))))).))...(((...)))------------- ( -26.50) >DroEre_CAF1 8789 85 + 1 CAGGCCACAUCAAAGCUGUAUAUA----CUUGCACAUA------------UGUAUUGGUAUUCGGUCGGGCGG-CACUGGGGGCGUGGCAGCCG-GGGUGGCA ...(((((......((((.((((.----..((((....------------))))...)))).))))(.((((.-(((.......))).).))).-).))))). ( -26.90) >DroYak_CAF1 9549 95 + 1 CAGGCCACAUCAAAGCUGUAUGUACAUACUUGCACAUAUA-------GUAUGUAUAUGUAUUCGGUCGGGCGG-GACUGGGGGCGUGGCAGCUGUGGGUGGCA ...(((((.(((.(((((((((((((((((.........)-------))))))))))((.(((((((......-))))))).))...)))))).)))))))). ( -40.50) >consensus CAGGCCACAUCAAAGCUGUAUAUA____C_UGCACAUACAUAUG__UGUAUGUAUAUGUACUCGGUCGGGCGG_GACUGGGGGCGUGGCAGC_G_G_______ ...(((((.((...................((((.((((((((....)))))))).))))(((((((.......))))))))).))))).............. (-13.67 = -16.46 + 2.79)


Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 12:54:23 2006