
| Sequence ID | X_DroMel_CAF1 |
|---|---|
| Location | 20,002,377 – 20,002,497 |
| Length | 120 |
| Max. P | 0.802281 |

| Location | 20,002,377 – 20,002,497 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 77.83 |
| Mean single sequence MFE | -46.23 |
| Consensus MFE | -23.64 |
| Energy contribution | -24.50 |
| Covariance contribution | 0.86 |
| Combinations/Pair | 1.32 |
| Mean z-score | -2.11 |
| Structure conservation index | 0.51 |
| SVM decision value | 0.62 |
| SVM RNA-class probability | 0.802281 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 20002377 120 + 22224390 UGUCGCUGGUGUAGCCGCCAUACUCGCAGCCGGCGUCCAUCAGCACGAGAUCCUGCUGACCCAACAACUGGCUGUUGGCCACGUAGUGGAUCACCGUGGCGUUUUUACCGGCAGCCACCA .((.(((((((.((.((((((.((.((((((((.((...((((((.(....).))))))....))..)))))))).))((((...))))......)))))).)).))))))).))..... ( -46.60) >DroVir_CAF1 1788 120 + 1 UGUCGCUGGUAUAGCCGCCGUAUUCGCAGCCGGCAUCCAUCAGCAGCAGCUCCUGCGGCUGCAGCAACUGUGUGUUGUUCACAUAGUGUAUAACGGUGGCAUUUUUGCCGGCGGCCACCA .((((((((((..((((((((....((((((.((........)).((((...)))))))))).(((.(((((((.....))))))))))...)))))))).....))))))))))..... ( -60.40) >DroPse_CAF1 1826 120 + 1 UGUCGCUGGUGUAUCCGCCGUACUCGCAGCCAGCAUCCAUGAGGACCAGAUCUUGCGGCUGCAACAGCUGGCUGUUGUCCACAUAGUGGAUGAUGGUGGCAUUGCGACCGGCGGCCACAA .((((((((((((.(((((((....(((((((((.(((....))).......((((....))))..))))))))).((((((...)))))).)))))))...))).)))))))))..... ( -56.20) >DroWil_CAF1 1773 120 + 1 UAUCACUCGUAUAGCCGCCGUACUCACAGCCAGCGUCCAUGAGAACCAAAUCCUUUGGCUGCAAUAACUGUGUAUUGUUCACAUAAUGGAUAAUUGUGGCAUUUUUGCCGGCAGCAACGA ......((((...((.(((.....(((((...(((.(((.(((.........)))))).))).....))))).((((((((.....))))))))...((((....))))))).)).)))) ( -29.80) >DroMoj_CAF1 1836 120 + 1 UAUCGCUCGUAUAGCCGCCAUAUUCACAGCCCGCAUCCAUCAGCAGCAGCUCCUGCGGCUGCAGAAUCUGUGUGUUGUUCACAUAGUGUAUGACGGUGGCAUUUCGGCCAGCUGCCACAA ........((...((((((.......(((((.((........)).((((...)))))))))((....(((((((.....)))))))....))..)))))).....(((.....))))).. ( -39.60) >DroAna_CAF1 1777 120 + 1 UGUCGCUGGUGUAGCCGCCGUACUCGCAGCCGGCGUCCAUCAGCACCAUGUCCUGCACCCCCACGACUUGAUUAUUGGUCACGUAGUGGAUGAUGGUGGCGUUCCGGCCGGAGGCUACCA .((((((((((..(((((.((....)).).))))...)))))))................((((.((.((((.....)))).)).))))..)))((((((.((((....)))))))))). ( -44.80) >consensus UGUCGCUGGUAUAGCCGCCGUACUCGCAGCCGGCAUCCAUCAGCACCAGAUCCUGCGGCUGCAACAACUGUGUGUUGUUCACAUAGUGGAUGACGGUGGCAUUUCGGCCGGCGGCCACCA ....((((((...((((((((....((.....))((((((..(((........)))....((((((......)))))).......)))))).))))))))......))))))........ (-23.64 = -24.50 + 0.86)


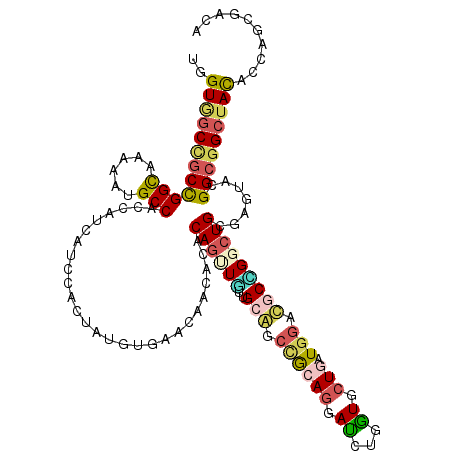
| Location | 20,002,377 – 20,002,497 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 77.83 |
| Mean single sequence MFE | -45.80 |
| Consensus MFE | -25.56 |
| Energy contribution | -25.73 |
| Covariance contribution | 0.17 |
| Combinations/Pair | 1.48 |
| Mean z-score | -1.79 |
| Structure conservation index | 0.56 |
| SVM decision value | 0.35 |
| SVM RNA-class probability | 0.698512 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 20002377 120 - 22224390 UGGUGGCUGCCGGUAAAAACGCCACGGUGAUCCACUACGUGGCCAACAGCCAGUUGUUGGGUCAGCAGGAUCUCGUGCUGAUGGACGCCGGCUGCGAGUAUGGCGGCUACACCAGCGACA ((((((((((((.((....(((..(((((.(((.........(((((((....)))))))(((((((.(....).)))))))))))))))...)))..)))))))))..)))))...... ( -54.10) >DroVir_CAF1 1788 120 - 1 UGGUGGCCGCCGGCAAAAAUGCCACCGUUAUACACUAUGUGAACAACACACAGUUGCUGCAGCCGCAGGAGCUGCUGCUGAUGGAUGCCGGCUGCGAAUACGGCGGCUAUACCAGCGACA (((((((((((((((....))))..........(((.((((.....)))).)))...(((((((((((......))))....((...))))))))).....)))))))..))))...... ( -46.80) >DroPse_CAF1 1826 120 - 1 UUGUGGCCGCCGGUCGCAAUGCCACCAUCAUCCACUAUGUGGACAACAGCCAGCUGUUGCAGCCGCAAGAUCUGGUCCUCAUGGAUGCUGGCUGCGAGUACGGCGGAUACACCAGCGACA ..(((.((((((.(((((..((((....((((((...((.(((((((((....))))).(((.(....)..))))))).)))))))).)))))))))...))))))...)))........ ( -50.80) >DroWil_CAF1 1773 120 - 1 UCGUUGCUGCCGGCAAAAAUGCCACAAUUAUCCAUUAUGUGAACAAUACACAGUUAUUGCAGCCAAAGGAUUUGGUUCUCAUGGACGCUGGCUGUGAGUACGGCGGCUAUACGAGUGAUA ((((.((((((((((....))))((............((....))...((((((((..((..(((..(((......)))..)))..)))))))))).))..))))))...))))...... ( -39.30) >DroMoj_CAF1 1836 120 - 1 UUGUGGCAGCUGGCCGAAAUGCCACCGUCAUACACUAUGUGAACAACACACAGAUUCUGCAGCCGCAGGAGCUGCUGCUGAUGGAUGCGGGCUGUGAAUAUGGCGGCUAUACGAGCGAUA ..((((((.(.....)...))))))(((((((.....((....))...(((((...(((((.((((((.((...)).))).))).))))).)))))..)))))))(((.....))).... ( -40.80) >DroAna_CAF1 1777 120 - 1 UGGUAGCCUCCGGCCGGAACGCCACCAUCAUCCACUACGUGACCAAUAAUCAAGUCGUGGGGGUGCAGGACAUGGUGCUGAUGGACGCCGGCUGCGAGUACGGCGGCUACACCAGCGACA ..(((((((((((((((....(((.((((.(((((.((.(((.......))).)).)))))))))(((.((...)).))).)))...))))))).))(.....))))))).......... ( -43.00) >consensus UGGUGGCCGCCGGCAAAAAUGCCACCAUCAUCCACUAUGUGAACAACACACAGUUGUUGCAGCCGCAGGAUCUGGUGCUGAUGGACGCCGGCUGCGAGUACGGCGGCUACACCAGCGACA ..((((((((((((......)))...........................((((((..(((.((((((.((...)).))).))).))))))))).......))))))))).......... (-25.56 = -25.73 + 0.17)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 12:53:57 2006