
| Sequence ID | X_DroMel_CAF1 |
|---|---|
| Location | 19,970,828 – 19,970,952 |
| Length | 124 |
| Max. P | 0.993655 |

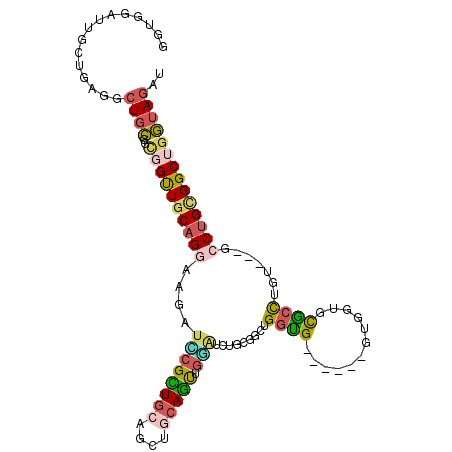
| Location | 19,970,828 – 19,970,927 |
|---|---|
| Length | 99 |
| Sequences | 6 |
| Columns | 108 |
| Reading direction | reverse |
| Mean pairwise identity | 72.94 |
| Mean single sequence MFE | -41.05 |
| Consensus MFE | -17.88 |
| Energy contribution | -17.83 |
| Covariance contribution | -0.05 |
| Combinations/Pair | 1.58 |
| Mean z-score | -2.27 |
| Structure conservation index | 0.44 |
| SVM decision value | 1.47 |
| SVM RNA-class probability | 0.956398 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
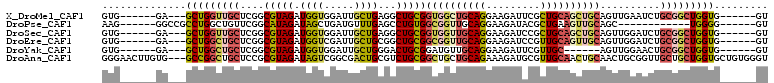
>X_DroMel_CAF1 19970828 99 - 22224390 GGUGGAUUGCUGAGGCUGCGGUGGCUGCAGGAAGAUUCGCUGCAGCUGCAGUUGAAUCUGCGGCUGGUG------GUGGUGCGCCUGU---GCCUGCGGCUGGUAGAU ......(((((..(((((((..(((.(((((.....((((..(((((((((......)))))))).)..------))))....)))))---))))))))))))))).. ( -46.80) >DroPse_CAF1 1456 87 - 1 AGCUGAUGUUUGAGCCUGUGGCGGUUGCAGGAAGAUACGCUGAAGUUGCAGC------------UGGGG------GUGGUGCUGUGGU---GCCUGUGGCUGAUAGAU .(((.(....).)))((((..((((..((((....(((((((......))))------------....(------(.....)))))..---.))))..)))))))).. ( -25.80) >DroSec_CAF1 5587 99 - 1 GGUGGAUUGCUGAGGCUGCGGUGGUUGCAGGAAGAUCCGCUGCAGCUGCAGUUGGAUCUGCGGCUGGUG------GUGGUGCGCCUGU---GCCUGCGGCUGGUAGAU .(..((((((((.(((((((((((...(.....)..))))))))))).))))..))))..).((..((.------(..(.(((....)---)))..).))..)).... ( -47.30) >DroEre_CAF1 5629 99 - 1 GGUCGAUUGCUGCGGCUGCGGCGGUUGCAGGAAGAUCCGUUGCAGUUGCAGUUGGAUCUGCGGCUGGUG------GUGGUGUGCCUGG---GCCUGCGGCUGGUAGAU ((((((((((((((((((((((((...(.....)..))))))))))))))))..)))).((((((.(.(------((.....)))).)---))).))))))....... ( -45.90) >DroYak_CAF1 5745 93 - 1 GGUGGAUUGCUGGGACUGCGGAUGUUGCAGGAAGAUUCGUUGC------AGUUGGAACUGCGGCUGGUG------GUGGUGCACCUGU---GGCUGCGGCUGGUAGAU ......((((..(((((((((..(((.......)))...))))------))))....(((((((((..(------(((...))))..)---)))))))))..)))).. ( -32.00) >DroAna_CAF1 1383 108 - 1 AGUCGGCGACUGCGUCUGCGGCUGCUGCAGAAAGAUGCGUUGCAACUGCAACUGCGGUUGCUGCUGGUGCUGUGGGUGCUGCACUUGCGAGGCCAGGGGCUGGAAAAU ((((((((((((((..(((((.(((((((......))))..))).)))))..)))))))))).(((((..((..((((...))))..))..))))).))))....... ( -48.50) >consensus GGUGGAUUGCUGAGGCUGCGGCGGUUGCAGGAAGAUCCGCUGCAGCUGCAGUUGGAUCUGCGGCUGGUG______GUGGUGCGCCUGU___GCCUGCGGCUGGUAGAU ...............((((..((((((((((....((((((((....))))).))).........((((............)))).......)))))))))))))).. (-17.88 = -17.83 + -0.05)

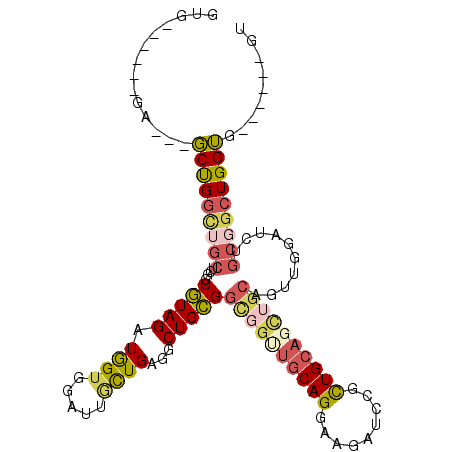
| Location | 19,970,856 – 19,970,952 |
|---|---|
| Length | 96 |
| Sequences | 6 |
| Columns | 111 |
| Reading direction | reverse |
| Mean pairwise identity | 72.15 |
| Mean single sequence MFE | -39.53 |
| Consensus MFE | -23.90 |
| Energy contribution | -24.57 |
| Covariance contribution | 0.67 |
| Combinations/Pair | 1.39 |
| Mean z-score | -2.38 |
| Structure conservation index | 0.60 |
| SVM decision value | 2.41 |
| SVM RNA-class probability | 0.993655 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 19970856 96 - 22224390 GUG------GA---GCUGGUUGCUCGGCGUAGAUGGUGGAUUGCUGAGGCUGCGGUGGCUGCAGGAAGAUUCGCUGCAGCUGCAGUUGAAUCUGCGGCUGGUG------GU ((.------((---((.....)))).))......((((((((.(((.((((.....)))).)))...)))))))).(((((((((......)))))))))...------.. ( -40.90) >DroPse_CAF1 1484 87 - 1 AAG------GGCCGCCUGGCUGUUCGGCAUAGAUAGCUGAUGUUUGAGCCUGUGGCGGUUGCAGGAAGAUACGCUGAAGUUGCAGC------------UGGGG------GU ...------.(((.((..((((((((((.(((....))).(((((...((((..(...)..)))).))))).)))))....)))))------------..)))------)) ( -32.90) >DroSec_CAF1 5615 96 - 1 GUG------GA---GCUGGUUGCUCGGCGUAGAUGGUGGAUUGCUGAGGCUGCGGUGGUUGCAGGAAGAUCCGCUGCAGCUGCAGUUGGAUCUGCGGCUGGUG------GU ((.------((---((.....)))).)).(((.(.(..((((((((.(((((((((((...(.....)..))))))))))).))))..))))..).))))...------.. ( -43.00) >DroEre_CAF1 5657 96 - 1 GUG------GA---GCUGGCUGCUCGGCGUAGAUGGUCGAUUGCUGCGGCUGCGGCGGUUGCAGGAAGAUCCGUUGCAGUUGCAGUUGGAUCUGCGGCUGGUG------GU ...------..---((..((((((((((.......)))))..((((((((((((((((...(.....)..)))))))))))))))).......)))))..)).------.. ( -46.20) >DroYak_CAF1 5773 90 - 1 GUG------GA---GCUGGCUGCUCGGCGUAGAUGGUGGAUUGCUGGGACUGCGGAUGUUGCAGGAAGAUUCGUUGC------AGUUGGAACUGCGGCUGGUG------GU ((.------((---((.....)))).)).........(((((.(((.(((.......))).)))...)))))(((((------(((....)))))))).....------.. ( -29.30) >DroAna_CAF1 1414 108 - 1 GGGAACUUGUG---GCCGGCUGCUCCGCGUAGAUAGUCGGCGACUGCGUCUGCGGCUGCUGCAGAAAGAUGCGUUGCAACUGCAACUGCGGUUGCUGCUGGUGCUGUGGGU ....(((..((---(((..((((.....)))).(((.(((((((((((..(((((.(((((((......))))..))).)))))..))))))))))))))).))))..))) ( -44.90) >consensus GUG______GA___GCUGGCUGCUCGGCGUAGAUGGUGGAUUGCUGAGGCUGCGGCGGUUGCAGGAAGAUCCGCUGCAGCUGCAGUUGGAUCUGCGGCUGGUG______GU ..............(((((((((....(((((.((((.....))))...)))))((((((((((.........))))))))))..........)))))))))......... (-23.90 = -24.57 + 0.67)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 12:53:45 2006