
| Sequence ID | X_DroMel_CAF1 |
|---|---|
| Location | 325,546 – 325,706 |
| Length | 160 |
| Max. P | 0.997040 |

| Location | 325,546 – 325,666 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 91.67 |
| Mean single sequence MFE | -44.54 |
| Consensus MFE | -34.86 |
| Energy contribution | -35.30 |
| Covariance contribution | 0.44 |
| Combinations/Pair | 1.13 |
| Mean z-score | -2.70 |
| Structure conservation index | 0.78 |
| SVM decision value | 1.98 |
| SVM RNA-class probability | 0.984614 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 325546 120 + 22224390 CACGAAGCUCUUCAAGAUGCUCUGGUGGAAGGUGGGGGCAAUCAUCUUACGAUGAUGACGCCAAUGAUGUCCAUUGCUGAUCAGUAGACCAUCACCGAACCAGGGCUUGAAGUAGCGAUA ......(((((((((...((((((((....(((((.(((.((((((....))))))...)))......(((.((((.....)))).)))..)))))..)))))))))))))).))).... ( -45.30) >DroVir_CAF1 112460 120 + 1 CACAAAGCUCUUCAAGAUGCUCUGAUGGAAUGUGGGCGCAAUCAUUUUGCGAUGAUGGCGCCAAUGGUGGCCAUUGCUGAUCAGCAGCCCAUCACCGAACCAGGGCUUAAAGUAGCGAUA ......((((((......((((((.(((......(((((.((((((....)))))).)))))..((((((...(((((....))))).)))))))))...))))))...))).))).... ( -41.30) >DroSim_CAF1 2539 120 + 1 CACGAAGCUCUUCAAGAUGCUCUGGUGGAAGGUGGGGGCAAUCAUCUUACGAUGAUGACGCCAAUGAUGUCCAUUGCUGAUCAGUAGACCAUCACCGAACCAGGGCUUGAAAUAGCGAUA ......(((.(((((...((((((((....(((((.(((.((((((....))))))...)))......(((.((((.....)))).)))..)))))..)))))))))))))..))).... ( -40.90) >DroYak_CAF1 2750 120 + 1 CACGAAGCUCUUCAAGAUACUCUGGUGGAAGGUGGGGGCAAUCAUCUUGCGAUGAUGGCGCCAAUGGUGUCCAUUGCUGAUCAGCAGACCAUCUCCGAACCAGGGCUUGAAGUAGCGAUA ......((((((((((...((((((((((.(((((..((.((((....((((((..((((((...))))))))))))))))..))...))))))))..)))))))))))))).))).... ( -49.50) >DroMoj_CAF1 99525 120 + 1 CACGAAGCUCUUCAGGAUGCUCUGAUGGAAGGUGGGUGCGAUCAUCUUGCGAUGAUGGCGCCAAUGGUGGCCGUUGCUGAUCAGCAGACCAUCCCCGAACCAGGGCUUAAAGUAUCUAUA .((.(((((((((.(((((.((((.(((..(((.(((((.((((((....)))))).)))))(((((...))))))))..))).)))).)))))..))...)))))))...))....... ( -45.70) >consensus CACGAAGCUCUUCAAGAUGCUCUGGUGGAAGGUGGGGGCAAUCAUCUUGCGAUGAUGGCGCCAAUGGUGUCCAUUGCUGAUCAGCAGACCAUCACCGAACCAGGGCUUGAAGUAGCGAUA ......((((((((((...(((((((....(((((..((.((((((....))))))((((.(((((.....))))).)).)).))...))))).....)))))))))))))).))).... (-34.86 = -35.30 + 0.44)

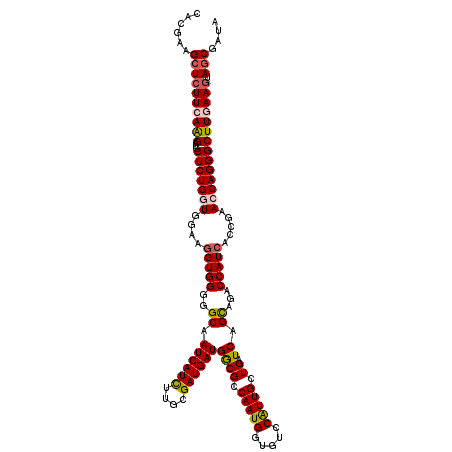
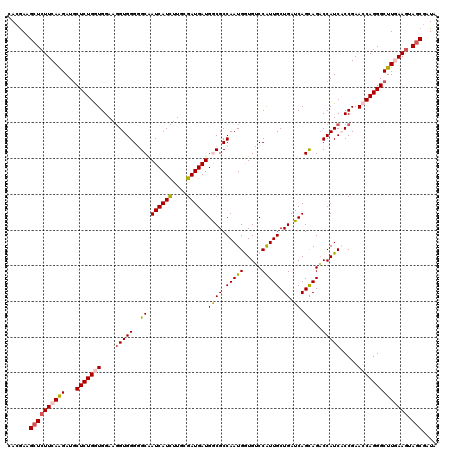
| Location | 325,546 – 325,666 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 91.67 |
| Mean single sequence MFE | -41.54 |
| Consensus MFE | -34.46 |
| Energy contribution | -34.18 |
| Covariance contribution | -0.28 |
| Combinations/Pair | 1.22 |
| Mean z-score | -2.62 |
| Structure conservation index | 0.83 |
| SVM decision value | 2.79 |
| SVM RNA-class probability | 0.997040 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 325546 120 - 22224390 UAUCGCUACUUCAAGCCCUGGUUCGGUGAUGGUCUACUGAUCAGCAAUGGACAUCAUUGGCGUCAUCAUCGUAAGAUGAUUGCCCCCACCUUCCACCAGAGCAUCUUGAAGAGCUUCGUG ....(((.((((((((.(((((..((((...(((((.((.....)).)))))......((((..((((((....))))))))))..))))....))))).))...)))))))))...... ( -42.10) >DroVir_CAF1 112460 120 - 1 UAUCGCUACUUUAAGCCCUGGUUCGGUGAUGGGCUGCUGAUCAGCAAUGGCCACCAUUGGCGCCAUCAUCGCAAAAUGAUUGCGCCCACAUUCCAUCAGAGCAUCUUGAAGAGCUUUGUG ....(((.(((((((.....((((..((((((((((.....))))(((((...)))))(((((.(((((......))))).)))))......))))))))))..))))))))))...... ( -41.00) >DroSim_CAF1 2539 120 - 1 UAUCGCUAUUUCAAGCCCUGGUUCGGUGAUGGUCUACUGAUCAGCAAUGGACAUCAUUGGCGUCAUCAUCGUAAGAUGAUUGCCCCCACCUUCCACCAGAGCAUCUUGAAGAGCUUCGUG ....(((.((((((((.(((((..((((...(((((.((.....)).)))))......((((..((((((....))))))))))..))))....))))).))...)))))))))...... ( -39.50) >DroYak_CAF1 2750 120 - 1 UAUCGCUACUUCAAGCCCUGGUUCGGAGAUGGUCUGCUGAUCAGCAAUGGACACCAUUGGCGCCAUCAUCGCAAGAUGAUUGCCCCCACCUUCCACCAGAGUAUCUUGAAGAGCUUCGUG ....(((.(((((((.((((((..((((.(((...(((....)))(((((...)))))((.((.((((((....)))))).))))))).)))).))))).)...))))))))))...... ( -44.60) >DroMoj_CAF1 99525 120 - 1 UAUAGAUACUUUAAGCCCUGGUUCGGGGAUGGUCUGCUGAUCAGCAACGGCCACCAUUGGCGCCAUCAUCGCAAGAUGAUCGCACCCACCUUCCAUCAGAGCAUCCUGAAGAGCUUCGUG ..............((...(((((.((((((.((((.((..........((((....))))((.((((((....)))))).))..........)).)))).))))))...)))))..)). ( -40.50) >consensus UAUCGCUACUUCAAGCCCUGGUUCGGUGAUGGUCUGCUGAUCAGCAAUGGACACCAUUGGCGCCAUCAUCGCAAGAUGAUUGCCCCCACCUUCCACCAGAGCAUCUUGAAGAGCUUCGUG ....(((.((((((((.(((((..((((...(((((.((.....)).)))))......((.((.((((((....)))))).)).))))))....))))).))...)))))))))...... (-34.46 = -34.18 + -0.28)

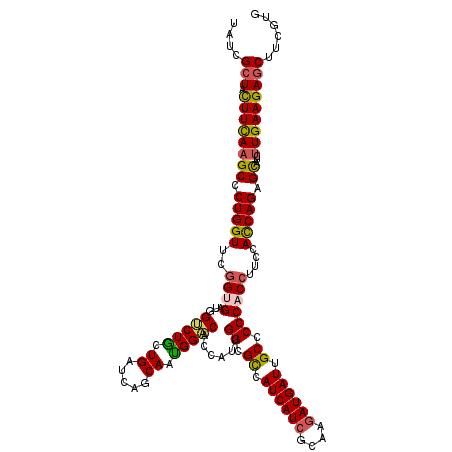
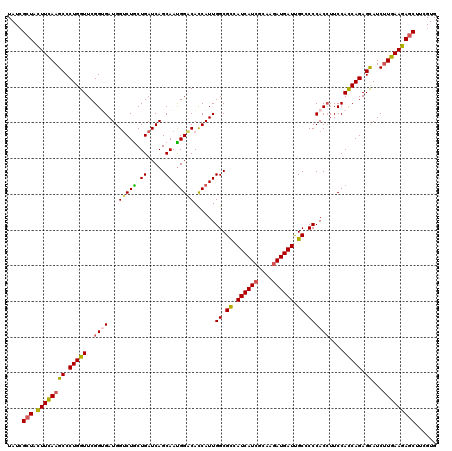
| Location | 325,586 – 325,706 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 90.17 |
| Mean single sequence MFE | -42.42 |
| Consensus MFE | -38.72 |
| Energy contribution | -38.96 |
| Covariance contribution | 0.24 |
| Combinations/Pair | 1.16 |
| Mean z-score | -1.82 |
| Structure conservation index | 0.91 |
| SVM decision value | 2.06 |
| SVM RNA-class probability | 0.986948 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 325586 120 - 22224390 GAUCCUGAGUGGGCACCAGCACUUGACCAAGGCGGAGGAGUAUCGCUACUUCAAGCCCUGGUUCGGUGAUGGUCUACUGAUCAGCAAUGGACAUCAUUGGCGUCAUCAUCGUAAGAUGAU ((((...(((((..((((.((((.(((((.(((...((((((....))))))..))).))))).)))).)))))))))))))..(((((.....))))).....((((((....)))))) ( -46.40) >DroVir_CAF1 112500 120 - 1 CAUAUUGAGCGGACACCAGCAUCUGACCAAGGCCGAGGAAUAUCGCUACUUUAAGCCCUGGUUCGGUGAUGGGCUGCUGAUCAGCAAUGGCCACCAUUGGCGCCAUCAUCGCAAAAUGAU (((.(((.((........))....(((((.(((.(((...........)))...))).)))))(((((((((((((.....))))....((((....)))).)))))))))))).))).. ( -36.90) >DroSim_CAF1 2579 120 - 1 GAUUCUGAGUGGGCACCAGCAUUUGACCAAGGCGGAGGAGUAUCGCUAUUUCAAGCCCUGGUUCGGUGAUGGUCUACUGAUCAGCAAUGGACAUCAUUGGCGUCAUCAUCGUAAGAUGAU ((((...(((((..((((.(((..(((((.(((...((((((....))))))..))).)))))..))).)))))))))))))..(((((.....))))).....((((((....)))))) ( -40.60) >DroYak_CAF1 2790 120 - 1 GAUCCUGAGCGGGCACCAGCACUUGACCAAGGCGGAGGAGUAUCGCUACUUCAAGCCCUGGUUCGGAGAUGGUCUGCUGAUCAGCAAUGGACACCAUUGGCGCCAUCAUCGCAAGAUGAU ((((...(((((..((((.(.((.(((((.(((...((((((....))))))..))).))))).)).).)))))))))))))..((((((...)))))).....((((((....)))))) ( -46.30) >DroMoj_CAF1 99565 120 - 1 GAUCUUGAGCGGACACCAGCACUUGACCAAGGCCGAGGAAUAUAGAUACUUUAAGCCCUGGUUCGGGGAUGGUCUGCUGAUCAGCAACGGCCACCAUUGGCGCCAUCAUCGCAAGAUGAU ((((...((((((..(((...((((((((.(((.((((.((....)).))))..))).))).)))))..))))))))))))).......((((....))))...((((((....)))))) ( -41.90) >consensus GAUCCUGAGCGGGCACCAGCACUUGACCAAGGCGGAGGAGUAUCGCUACUUCAAGCCCUGGUUCGGUGAUGGUCUGCUGAUCAGCAAUGGACACCAUUGGCGCCAUCAUCGCAAGAUGAU ((((...(((((..((((.((((.(((((.(((...((((((....))))))..))).))))).)))).)))))))))))))..((((((...)))))).....((((((....)))))) (-38.72 = -38.96 + 0.24)

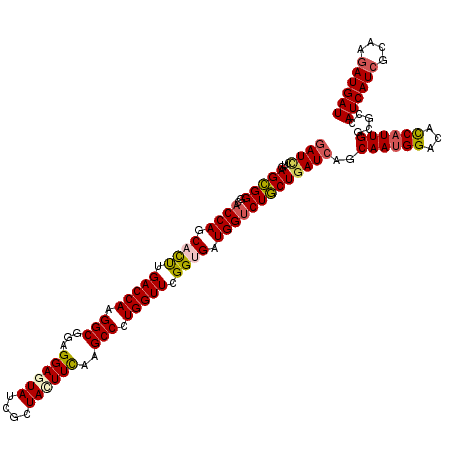
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 09:33:22 2006