
| Sequence ID | X_DroMel_CAF1 |
|---|---|
| Location | 19,571,633 – 19,571,748 |
| Length | 115 |
| Max. P | 0.797862 |

| Location | 19,571,633 – 19,571,748 |
|---|---|
| Length | 115 |
| Sequences | 6 |
| Columns | 118 |
| Reading direction | forward |
| Mean pairwise identity | 80.70 |
| Mean single sequence MFE | -39.42 |
| Consensus MFE | -25.95 |
| Energy contribution | -25.82 |
| Covariance contribution | -0.14 |
| Combinations/Pair | 1.28 |
| Mean z-score | -1.92 |
| Structure conservation index | 0.66 |
| SVM decision value | 0.61 |
| SVM RNA-class probability | 0.797862 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
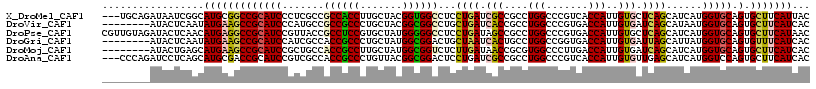
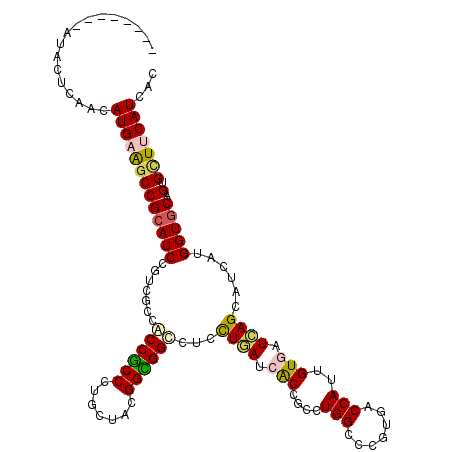
>X_DroMel_CAF1 19571633 115 + 22224390 ---UGCAGAUAAUCGGCAUGCGGCCGCAUCCCUCGCCGCCACCUUGCUACGGUGGCCUCCUGAUCGCCGCCUGGCCCGUCACCAUUGUGCUCAGCAUCAUGGUGCAGUGCUUCAUUAC ---.(((.......(((..(((((.((.......)).((((((.......)))))).........)))))...))).(.((((((.((((...)))).)))))))..)))........ ( -41.10) >DroVir_CAF1 9652 110 + 1 --------AUACUCAAUAUGAAGCCGCAUCCCAUGCCGCCGCCCUGCUACGGCGGCCUGCUGAUCACCGCCUGGCCCGUGACCAUUGUGAUCAGCAUAAUGGUGCAGUGCUUCAUCAC --------.........(((((((((((((....(((((((........))))))).((((((((((....(((.......)))..))))))))))....))))).).)))))))... ( -46.00) >DroPse_CAF1 4977 118 + 1 CGUUGUAGAUACUCAACAUGAGGCCGCAUCCGUUACCGCCUCCGUGCUAUGGGGGCCUCCUGAUAGCCGCCUGGCCCGUGACCAUUGUGCUCAGCAUCAUGGUGCAGUGCUUCAUAAC .((((........))))((((((((((((..(((((.(((..((.(((((.(((....))).)))))))...)))..)))))....))))...((((....)))).).)))))))... ( -42.20) >DroGri_CAF1 8622 110 + 1 --------AUACUCAAUAUGAAGCCGCAUCCAUCGCCACCGCCCUGCUAUGGCGGACUGCUAAUCACUGCCUGGCCGGUGACCAUUGUGAUUAGCAUUAUGGUGCAGUGUUUCAUCAC --------.........(((((((((((.((((.....(((((.......)))))..((((((((((....(((.......)))..))))))))))..))))))).).)))))))... ( -37.40) >DroMoj_CAF1 9137 110 + 1 --------AUACUGAGCAUGAAGCCGCAUCCGCUGCCACCGCCUUGCUAUGGCGGUCUCUUGAUAACCGCGUGGCCCUUGACCAUUGUGAUCAGCAUCAUGGUGCAGUGCUUCAUCAC --------.......(.(((((((((((((.(.(((.((((((.......))))))....((((...(((((((.......))).))))))))))).)..))))).).)))))))).. ( -34.10) >DroAna_CAF1 1675 115 + 1 ---CCCAGAUCCUCAGCAUGCGACCGCAUCCGUCGCCACCGCCCUGUUACGGCGGACUCCUGAUCGCCGCCUGGCCCGUCACCAUUGUGUUGAGCAUCAUGGUCCAGUGCUUCAUCAC ---.((((((.(((((((((((((.......)))))..(((((.......))))).....((((.(((....)))..)))).....)))))))).))).)))................ ( -35.70) >consensus ________AUACUCAACAUGAAGCCGCAUCCGUCGCCACCGCCCUGCUACGGCGGCCUCCUGAUCACCGCCUGGCCCGUGACCAUUGUGAUCAGCAUCAUGGUGCAGUGCUUCAUCAC .................(((((((((((((.......((((((.......))))))...((((.(((....(((.......)))..))).))))......))))).).)))))))... (-25.95 = -25.82 + -0.14)
| Location | 19,571,633 – 19,571,748 |
|---|---|
| Length | 115 |
| Sequences | 6 |
| Columns | 118 |
| Reading direction | reverse |
| Mean pairwise identity | 80.70 |
| Mean single sequence MFE | -44.35 |
| Consensus MFE | -25.97 |
| Energy contribution | -26.47 |
| Covariance contribution | 0.50 |
| Combinations/Pair | 1.17 |
| Mean z-score | -1.87 |
| Structure conservation index | 0.59 |
| SVM decision value | 0.17 |
| SVM RNA-class probability | 0.616592 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 19571633 115 - 22224390 GUAAUGAAGCACUGCACCAUGAUGCUGAGCACAAUGGUGACGGGCCAGGCGGCGAUCAGGAGGCCACCGUAGCAAGGUGGCGGCGAGGGAUGCGGCCGCAUGCCGAUUAUCUGCA--- ........((.(((((((((..(((...)))..)))))).)))))...((((.((((..(..((((((.......))))))..)..((.((((....)))).))))))..)))).--- ( -45.60) >DroVir_CAF1 9652 110 - 1 GUGAUGAAGCACUGCACCAUUAUGCUGAUCACAAUGGUCACGGGCCAGGCGGUGAUCAGCAGGCCGCCGUAGCAGGGCGGCGGCAUGGGAUGCGGCUUCAUAUUGAGUAU-------- ...(((((((...(((((....((((((((((..((((.....))))....)))))))))).((((((((......))))))))...)).))).))))))).........-------- ( -55.90) >DroPse_CAF1 4977 118 - 1 GUUAUGAAGCACUGCACCAUGAUGCUGAGCACAAUGGUCACGGGCCAGGCGGCUAUCAGGAGGCCCCCAUAGCACGGAGGCGGUAACGGAUGCGGCCUCAUGUUGAGUAUCUACAACG ((((((..((.(((.(((((..(((...)))..)))))..)))))..((.((((.......))))))))))))...(((((.(((.....))).)))))..((((........)))). ( -39.60) >DroGri_CAF1 8622 110 - 1 GUGAUGAAACACUGCACCAUAAUGCUAAUCACAAUGGUCACCGGCCAGGCAGUGAUUAGCAGUCCGCCAUAGCAGGGCGGUGGCGAUGGAUGCGGCUUCAUAUUGAGUAU-------- ...(((((...(((((((((..((((((((((..((((.....))))....))))))))))...((((((.((...)).)))))))))).))))).))))).........-------- ( -43.80) >DroMoj_CAF1 9137 110 - 1 GUGAUGAAGCACUGCACCAUGAUGCUGAUCACAAUGGUCAAGGGCCACGCGGUUAUCAAGAGACCGCCAUAGCAAGGCGGUGGCAGCGGAUGCGGCUUCAUGCUCAGUAU-------- ..........((((...(((((.(((((((....((((.....))))(((((((.......))))(((((.((...)).))))).)))))).)))).)))))..))))..-------- ( -38.70) >DroAna_CAF1 1675 115 - 1 GUGAUGAAGCACUGGACCAUGAUGCUCAACACAAUGGUGACGGGCCAGGCGGCGAUCAGGAGUCCGCCGUAACAGGGCGGUGGCGACGGAUGCGGUCGCAUGCUGAGGAUCUGGG--- (((......)))....(((.(((.((((.(((..((....)).(((..((((((..(....)..)))))).....))).)))(((((.......)))))....)))).)))))).--- ( -42.50) >consensus GUGAUGAAGCACUGCACCAUGAUGCUGAUCACAAUGGUCACGGGCCAGGCGGCGAUCAGGAGGCCGCCAUAGCAGGGCGGCGGCAACGGAUGCGGCCUCAUGCUGAGUAU________ ...((((....(((((((.((((((((.......((((.....))))..)))).))))....((((((.......))))))......)).)))))..))))................. (-25.97 = -26.47 + 0.50)


Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 12:49:16 2006