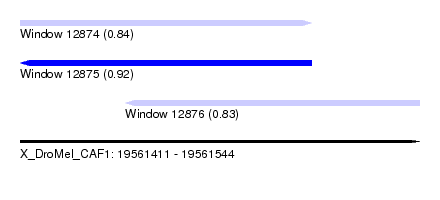
| Sequence ID | X_DroMel_CAF1 |
|---|---|
| Location | 19,561,411 – 19,561,544 |
| Length | 133 |
| Max. P | 0.920256 |

| Location | 19,561,411 – 19,561,508 |
|---|---|
| Length | 97 |
| Sequences | 6 |
| Columns | 109 |
| Reading direction | forward |
| Mean pairwise identity | 74.62 |
| Mean single sequence MFE | -30.77 |
| Consensus MFE | -16.43 |
| Energy contribution | -17.69 |
| Covariance contribution | 1.25 |
| Combinations/Pair | 1.22 |
| Mean z-score | -1.84 |
| Structure conservation index | 0.53 |
| SVM decision value | 0.76 |
| SVM RNA-class probability | 0.843721 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
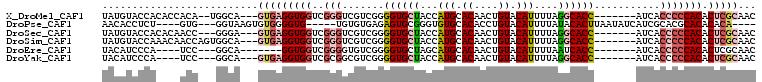
>X_DroMel_CAF1 19561411 97 + 22224390 UAUGUACCACACCACA--UGGCA---GUGAGGUGGUCGGGUCGUCGGGGUGCUACCAUGCACAACUGUACAUUUUAGGCACC-------AUCACCCCCACACUCGCAAC (((((........)))--))...---(((((((((..((((.(....((((((...(((.((....)).)))....))))))-------..))))))))).)))))... ( -31.40) >DroPse_CAF1 28189 93 + 1 AACACCUCU----GUG---GGUAAGUGUGGGGUG-----UGUGUGAGAGUGCGGGUGUGCACACCUGUACAUUUUAUACACUUAAUAUCAUCGCACGCACACACA---- .........----(((---.((..((((((((((-----((((((((((((((((((....))))))))).)))))))))).....))).))))))..)).))).---- ( -37.70) >DroSec_CAF1 25262 96 + 1 UAUGUACCACACAACC---GGGA---GUGAGGUGGUCGGGUCGUCGGGGUGCUACCAUGCACAACUGUACAUUUUAGGCACC-------AUCACCCCCACACUCGCAAC ......((........---))..---(((((((((..((((.(....((((((...(((.((....)).)))....))))))-------..))))))))).)))))... ( -31.00) >DroSim_CAF1 21873 99 + 1 UAUGUACCAAACAACCAGUGGCA---GUGAGGUGGUCGGGUCGUCGGGGUGCUACCAUGCACAACUGUACAUUUUAGGCACC-------AUCACCCCCACACUCGCAAC ......(((.........)))..---(((((((((..((((.(....((((((...(((.((....)).)))....))))))-------..))))))))).)))))... ( -30.20) >DroEre_CAF1 25010 88 + 1 UACAUCCCA----UCC---GGCA-------GGUGGUCGGGGUGUCGGGGUGCUAGCAUGCACAACUGUACAUUUUAAUCACC-------AUCACCCCCACACUCGCAAC .........----...---.((.-------.(((...((((((....((((.(((.(((.((....)).))).)))..))))-------..))))))..)))..))... ( -27.60) >DroYak_CAF1 24328 92 + 1 UACAUCCCA----UCC---GGCA---GUGAGGUGGUCGCGGCGUCGGGGUGCUACCAUGCACAACUGUACAUUUUAGGCACC-------AUCACCCCCACACUCGCAAC .........----...---....---(((((((((....((.((...((((((...(((.((....)).)))....))))))-------...)))))))).)))))... ( -26.70) >consensus UACAUACCA____ACC___GGCA___GUGAGGUGGUCGGGGCGUCGGGGUGCUACCAUGCACAACUGUACAUUUUAGGCACC_______AUCACCCCCACACUCGCAAC ..........................(((((((((..(((.......((((((...(((.((....)).)))....))))))...........))))))).)))))... (-16.43 = -17.69 + 1.25)


| Location | 19,561,411 – 19,561,508 |
|---|---|
| Length | 97 |
| Sequences | 6 |
| Columns | 109 |
| Reading direction | reverse |
| Mean pairwise identity | 74.62 |
| Mean single sequence MFE | -35.34 |
| Consensus MFE | -18.87 |
| Energy contribution | -19.82 |
| Covariance contribution | 0.95 |
| Combinations/Pair | 1.10 |
| Mean z-score | -1.99 |
| Structure conservation index | 0.53 |
| SVM decision value | 1.13 |
| SVM RNA-class probability | 0.920256 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 19561411 97 - 22224390 GUUGCGAGUGUGGGGGUGAU-------GGUGCCUAAAAUGUACAGUUGUGCAUGGUAGCACCCCGACGACCCGACCACCUCAC---UGCCA--UGUGGUGUGGUACAUA ....((.((((.((((((..-------..((((....((((((....)))))))))).)))))).)).)).))(((((.((((---.....--.)))).)))))..... ( -37.90) >DroPse_CAF1 28189 93 - 1 ----UGUGUGUGCGUGCGAUGAUAUUAAGUGUAUAAAAUGUACAGGUGUGCACACCCGCACUCUCACACA-----CACCCCACACUUACC---CAC----AGAGGUGUU ----((((((((.(((((...........(((((.....)))))((((....)))))))))...))))))-----))....((((((...---...----..)))))). ( -30.60) >DroSec_CAF1 25262 96 - 1 GUUGCGAGUGUGGGGGUGAU-------GGUGCCUAAAAUGUACAGUUGUGCAUGGUAGCACCCCGACGACCCGACCACCUCAC---UCCC---GGUUGUGUGGUACAUA ((((.(..(((.((((((..-------..((((....((((((....)))))))))).)))))).)))..))))).(((.(((---....---....))).)))..... ( -35.30) >DroSim_CAF1 21873 99 - 1 GUUGCGAGUGUGGGGGUGAU-------GGUGCCUAAAAUGUACAGUUGUGCAUGGUAGCACCCCGACGACCCGACCACCUCAC---UGCCACUGGUUGUUUGGUACAUA ((((.(..(((.((((((..-------..((((....((((((....)))))))))).)))))).)))..))))).(((..((---.(((...))).))..)))..... ( -34.30) >DroEre_CAF1 25010 88 - 1 GUUGCGAGUGUGGGGGUGAU-------GGUGAUUAAAAUGUACAGUUGUGCAUGCUAGCACCCCGACACCCCGACCACC-------UGCC---GGA----UGGGAUGUA ..((((.((((.((((((.(-------(((.......((((((....)))))))))).)))))).))))((((.((...-------....---)).----)))).)))) ( -34.51) >DroYak_CAF1 24328 92 - 1 GUUGCGAGUGUGGGGGUGAU-------GGUGCCUAAAAUGUACAGUUGUGCAUGGUAGCACCCCGACGCCGCGACCACCUCAC---UGCC---GGA----UGGGAUGUA ((((((.((((.((((((..-------..((((....((((((....)))))))))).)))))).))))))))))((.(((((---....---.).----)))).)).. ( -39.40) >consensus GUUGCGAGUGUGGGGGUGAU_______GGUGCCUAAAAUGUACAGUUGUGCAUGGUAGCACCCCGACGACCCGACCACCUCAC___UGCC___GGU____UGGGACAUA .....(((.((((..............((((.(((..((((((....))))))..)))))))............)))))))............................ (-18.87 = -19.82 + 0.95)

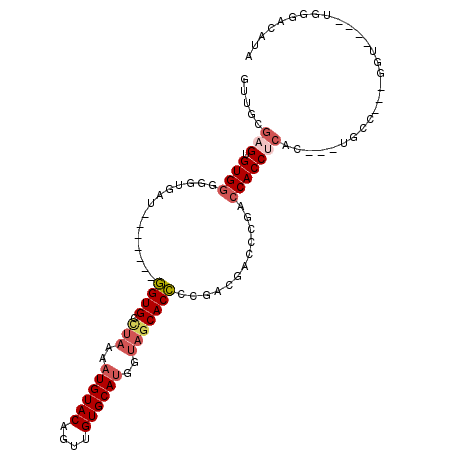
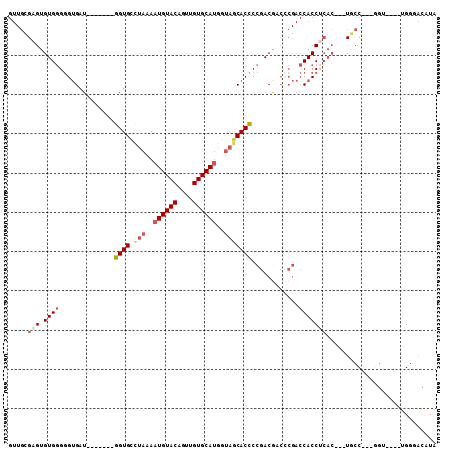
| Location | 19,561,446 – 19,561,544 |
|---|---|
| Length | 98 |
| Sequences | 5 |
| Columns | 98 |
| Reading direction | reverse |
| Mean pairwise identity | 93.03 |
| Mean single sequence MFE | -33.72 |
| Consensus MFE | -28.28 |
| Energy contribution | -28.04 |
| Covariance contribution | -0.24 |
| Combinations/Pair | 1.13 |
| Mean z-score | -1.53 |
| Structure conservation index | 0.84 |
| SVM decision value | 0.70 |
| SVM RNA-class probability | 0.827794 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 19561446 98 - 22224390 GUCUCUGGUAUUGGAGCGUGUCAGUGAGUGAGUGCGGUUGCGAGUGUGGGGGUGAUGGUGCCUAAAAUGUACAGUUGUGCAUGGUAGCACCCCGACGA (((((...((((((......))))))...))).)).........(((.((((((....((((....((((((....)))))))))).)))))).))). ( -31.20) >DroSec_CAF1 25296 98 - 1 GUCUCUGGCAUUGGAGCGUGUCGGUGAGUGAGUGCGGUUGCGAGUGUGGGGGUGAUGGUGCCUAAAAUGUACAGUUGUGCAUGGUAGCACCCCGACGA .((.(((((((......))))))).)).....(((....)))..(((.((((((....((((....((((((....)))))))))).)))))).))). ( -33.90) >DroSim_CAF1 21910 98 - 1 GUCGCUGGCAUUGGAGCGUGUCGGUGAGUGAGUGCGGUUGCGAGUGUGGGGGUGAUGGUGCCUAAAAUGUACAGUUGUGCAUGGUAGCACCCCGACGA .((((((((((......)))))))))).....(((....)))..(((.((((((....((((....((((((....)))))))))).)))))).))). ( -37.50) >DroEre_CAF1 25036 94 - 1 GUCUCUGGCAUUGGAGCGUGU----GAGUGAGUGUGGUUGCGAGUGUGGGGGUGAUGGUGAUUAAAAUGUACAGUUGUGCAUGCUAGCACCCCGACAC .((.((.((((......))))----.)).))............((((.((((((.((((.......((((((....)))))))))).)))))).)))) ( -32.31) >DroYak_CAF1 24358 94 - 1 AUCUCUGGCAUUGGAGCGUGU----GGGUGAGUGCGGUUGCGAGUGUGGGGGUGAUGGUGCCUAAAAUGUACAGUUGUGCAUGGUAGCACCCCGACGC .((.((.((((......))))----.)).)).(((....))).((((.((((((....((((....((((((....)))))))))).)))))).)))) ( -33.70) >consensus GUCUCUGGCAUUGGAGCGUGUC_GUGAGUGAGUGCGGUUGCGAGUGUGGGGGUGAUGGUGCCUAAAAUGUACAGUUGUGCAUGGUAGCACCCCGACGA ((..(((.((((.(..(........)..).)))))))..))...(((.((((((....((((....((((((....)))))))))).)))))).))). (-28.28 = -28.04 + -0.24)



Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 12:49:03 2006