
| Sequence ID | X_DroMel_CAF1 |
|---|---|
| Location | 19,549,109 – 19,549,200 |
| Length | 91 |
| Max. P | 0.999889 |

| Location | 19,549,109 – 19,549,200 |
|---|---|
| Length | 91 |
| Sequences | 5 |
| Columns | 92 |
| Reading direction | forward |
| Mean pairwise identity | 98.47 |
| Mean single sequence MFE | -26.40 |
| Consensus MFE | -26.44 |
| Energy contribution | -26.44 |
| Covariance contribution | -0.00 |
| Combinations/Pair | 1.00 |
| Mean z-score | -2.80 |
| Structure conservation index | 1.00 |
| SVM decision value | 4.40 |
| SVM RNA-class probability | 0.999889 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
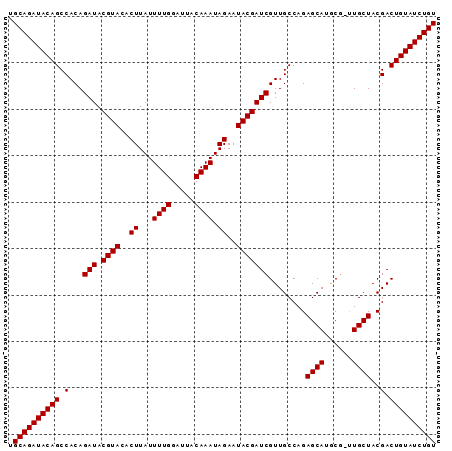
>X_DroMel_CAF1 19549109 91 + 22224390 UGCAGAUACAGCCACAGAUACGUACACUUAUUUUGGAUUACAAAUAGAAUACGAUCGUUGCCAGAGCAUGCG-UUGCUACGACUGUAUCUGU .((((((((((.(...(((.((((..((...((((.....)))).))..)))))))........((((....-.))))..).)))))))))) ( -26.40) >DroSec_CAF1 12702 91 + 1 UGCAGAUACAGCCACAGAUACGUACACUUAUUUUGGAUUACAAAUAGAAUACGAUCGUUGCCAGAGCAUGCG-UUGCUACGACUGUAUCUGU .((((((((((.(...(((.((((..((...((((.....)))).))..)))))))........((((....-.))))..).)))))))))) ( -26.40) >DroSim_CAF1 13638 91 + 1 UGCAGAUACAGCCACAGAUACGUACACUUAUUUUGGAUUACAAAUAGAAUACGAUCGUUGCCAGAGCAUGCG-UUGCUACGACUGUAUCUGU .((((((((((.(...(((.((((..((...((((.....)))).))..)))))))........((((....-.))))..).)))))))))) ( -26.40) >DroEre_CAF1 12654 91 + 1 UGCAGAUACAGCCACAGAUACGUACACUUAUUUUGGAUUACAAAUAGAAUACGAUCGUUGCCAGAGCAUACG-UUGCUACGACUGUAUCUGU .((((((((((.(...(((.((((..((...((((.....)))).))..)))))))........((((....-.))))..).)))))))))) ( -26.40) >DroYak_CAF1 12339 92 + 1 UGCAGAUACAGCCACAGAUACGUACACUUAUUUUGGAUUACAAAUAGAAUACGAUCGUUGCCAGAGCAUACGACUGCUACGACUGUAUCUGU .(((((((((((..(((...((((..((...((((.....)))).))..)))).(((((((....))).)))))))....).)))))))))) ( -26.40) >consensus UGCAGAUACAGCCACAGAUACGUACACUUAUUUUGGAUUACAAAUAGAAUACGAUCGUUGCCAGAGCAUGCG_UUGCUACGACUGUAUCUGU .((((((((((.(...(((.((((..((...((((.....)))).))..)))))))........((((......))))..).)))))))))) (-26.44 = -26.44 + -0.00)


| Location | 19,549,109 – 19,549,200 |
|---|---|
| Length | 91 |
| Sequences | 5 |
| Columns | 92 |
| Reading direction | reverse |
| Mean pairwise identity | 98.47 |
| Mean single sequence MFE | -29.84 |
| Consensus MFE | -28.86 |
| Energy contribution | -28.86 |
| Covariance contribution | -0.00 |
| Combinations/Pair | 1.00 |
| Mean z-score | -3.67 |
| Structure conservation index | 0.97 |
| SVM decision value | 4.36 |
| SVM RNA-class probability | 0.999881 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
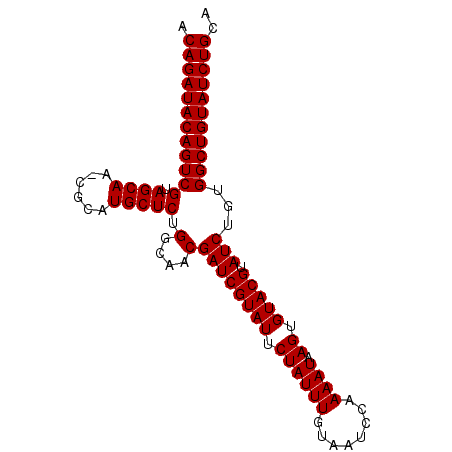
>X_DroMel_CAF1 19549109 91 - 22224390 ACAGAUACAGUCGUAGCAA-CGCAUGCUCUGGCAACGAUCGUAUUCUAUUUGUAAUCCAAAAUAAGUGUACGUAUCUGUGGCUGUAUCUGCA .((((((((((((.((((.-....))))).(....)((((((((.((((((........)))).)).))))).)))...))))))))))).. ( -28.50) >DroSec_CAF1 12702 91 - 1 ACAGAUACAGUCGUAGCAA-CGCAUGCUCUGGCAACGAUCGUAUUCUAUUUGUAAUCCAAAAUAAGUGUACGUAUCUGUGGCUGUAUCUGCA .((((((((((((.((((.-....))))).(....)((((((((.((((((........)))).)).))))).)))...))))))))))).. ( -28.50) >DroSim_CAF1 13638 91 - 1 ACAGAUACAGUCGUAGCAA-CGCAUGCUCUGGCAACGAUCGUAUUCUAUUUGUAAUCCAAAAUAAGUGUACGUAUCUGUGGCUGUAUCUGCA .((((((((((((.((((.-....))))).(....)((((((((.((((((........)))).)).))))).)))...))))))))))).. ( -28.50) >DroEre_CAF1 12654 91 - 1 ACAGAUACAGUCGUAGCAA-CGUAUGCUCUGGCAACGAUCGUAUUCUAUUUGUAAUCCAAAAUAAGUGUACGUAUCUGUGGCUGUAUCUGCA .(((((((((((((((..(-((((((((..(....)(((..((.......))..))).......)))))))))..))))))))))))))).. ( -32.00) >DroYak_CAF1 12339 92 - 1 ACAGAUACAGUCGUAGCAGUCGUAUGCUCUGGCAACGAUCGUAUUCUAUUUGUAAUCCAAAAUAAGUGUACGUAUCUGUGGCUGUAUCUGCA .(((((((((((((((..(((((.(((....))))))))(((((.((((((........)))).)).)))))...))))))))))))))).. ( -31.70) >consensus ACAGAUACAGUCGUAGCAA_CGCAUGCUCUGGCAACGAUCGUAUUCUAUUUGUAAUCCAAAAUAAGUGUACGUAUCUGUGGCUGUAUCUGCA .((((((((((((.((((......))))).(....)((((((((.((((((........)))).)).))))).)))...))))))))))).. (-28.86 = -28.86 + -0.00)


Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 12:48:50 2006