
| Sequence ID | X_DroMel_CAF1 |
|---|---|
| Location | 19,483,685 – 19,483,885 |
| Length | 200 |
| Max. P | 0.999221 |

| Location | 19,483,685 – 19,483,805 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 92.92 |
| Mean single sequence MFE | -49.50 |
| Consensus MFE | -46.78 |
| Energy contribution | -46.22 |
| Covariance contribution | -0.56 |
| Combinations/Pair | 1.19 |
| Mean z-score | -2.04 |
| Structure conservation index | 0.95 |
| SVM decision value | 3.44 |
| SVM RNA-class probability | 0.999221 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 19483685 120 - 22224390 AACGACUGCGAAACGCGCAGAUUGAAGCGGACGCGGAGGUCAAGAGCGGCAACGUGUCGUGCAGCGUUCUGGAUCCAGUAUACCUGGAAUGGGAUGCGCUGGUCAGUGUGUUGGACGGCG ..((((((((.....))))).)))..(((((((((...(((......)))..)))))).))).((((((((..(((((.....))))).))))))))((((..(((....)))..)))). ( -47.70) >DroSec_CAF1 5670 120 - 1 AACGCCUGCGAAAUGCGCAGGCUGAAGCGGAUGCGGAGGCCAAGAGCGGCAACGUGUCGUGCAGCGUGCAGGAUCCAGUAUACCUGGAAUGGGAUGCACUGGUCAGUGUGUUGGACGGAG ...(((((((.....))))))).......((((((...(((......)))..))))))((.(((((..(..((.(((((.(((((.....))).)).))))))).)..))))).)).... ( -49.10) >DroSim_CAF1 6620 120 - 1 AACGCCUGCGAAAUGCGCAGGCUGAAGCGGACGCGGAGGUCAAGAGCGGCAACGUGUCGUGCAGCGUGCUGGAUCCAGUAUACCUGGAAUGGGAUGCACUGGUCAGUGUGUUGGACGGGG ...(((((((.....))))))).......((((((...(((......)))..))))))((.(((((..((((..(((((.(((((.....))).)).)))))))))..))))).)).... ( -52.10) >DroEre_CAF1 5351 120 - 1 AACGCCUGCGAAACGCGCAGGCCGAAGCGGAUGCGGAGGUCAAAAGCGGUAACGUGUCGUGCAGCGUGCUGGAUCCGGUUUACCUUGAAUGGGAUGCACUGGUGAGUGUGCUGGACGGCG ...(((((((.....)))))))....(((..(((...........)))....)))(((((.(((((..((.(..(((((.(((((.....))).)).)))))).))..))))).))))). ( -48.10) >DroYak_CAF1 4906 120 - 1 AACGCCUGCGAAACGCGCAGGCUGAAGCGGAUGCGGAGGUCAAGAGCGGCAACGUGUCGUGCAGCGUGCUGGAUCCGGUAUACCUGGAAUGGGAUGCACUGGUGAGUGUGCUGGACGGCG ...(((((((.....)))))))....((.((((((...(((......)))..))))))((.(((((..((.(..(((((.(((((.....))).)).)))))).))..))))).)).)). ( -50.50) >consensus AACGCCUGCGAAACGCGCAGGCUGAAGCGGAUGCGGAGGUCAAGAGCGGCAACGUGUCGUGCAGCGUGCUGGAUCCAGUAUACCUGGAAUGGGAUGCACUGGUCAGUGUGUUGGACGGCG ...(((((((.....))))))).......((((((...(((......)))..))))))((.(((((..(((...(((((.(((((.....))).)).))))).)))..))))).)).... (-46.78 = -46.22 + -0.56)


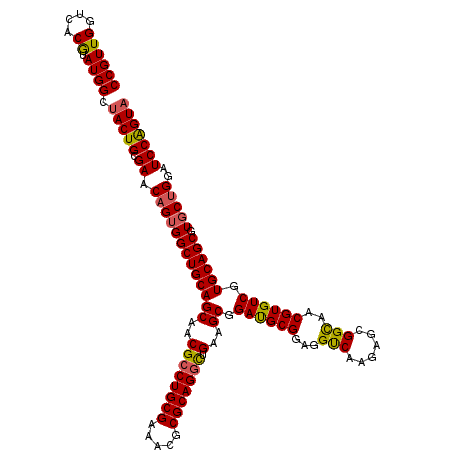
| Location | 19,483,725 – 19,483,845 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 94.33 |
| Mean single sequence MFE | -49.90 |
| Consensus MFE | -46.32 |
| Energy contribution | -46.00 |
| Covariance contribution | -0.32 |
| Combinations/Pair | 1.14 |
| Mean z-score | -1.66 |
| Structure conservation index | 0.93 |
| SVM decision value | 2.09 |
| SVM RNA-class probability | 0.987727 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 19483725 120 - 22224390 CCGUUGGUCACGUAUGGCUACUGCGAACAGUGGCUGCAGCAACGACUGCGAAACGCGCAGAUUGAAGCGGACGCGGAGGUCAAGAGCGGCAACGUGUCGUGCAGCGUUCUGGAUCCAGUA .....(((((....)))))((((.((.(((..((((((((..((((((((.....))))).)))..)).((((((...(((......)))..)))))).))))))...)))..)))))). ( -45.80) >DroSec_CAF1 5710 120 - 1 CCGUUGAUCACGUAUGGCUACUGCGAACAGUGGCUGCAGCAACGCCUGCGAAAUGCGCAGGCUGAAGCGGAUGCGGAGGCCAAGAGCGGCAACGUGUCGUGCAGCGUGCAGGAUCCAGUA .....((((..((((((((((((....)))))))((((((..((((((((.....))))))).)..)).((((((...(((......)))..)))))).)))).)))))..))))..... ( -51.40) >DroSim_CAF1 6660 120 - 1 CCGUUGAUCACGUAUGGCUACUGCGAACAGUGGCUGCAGCAACGCCUGCGAAAUGCGCAGGCUGAAGCGGACGCGGAGGUCAAGAGCGGCAACGUGUCGUGCAGCGUGCUGGAUCCAGUA .....((((......((((((((....)))))))).(((((..(((((((.....)))))))....(((((((((...(((......)))..)))))).)))....)))))))))..... ( -51.60) >DroEre_CAF1 5391 120 - 1 CCGUUGGUCACGUAUGGCUACUGCGAACAGUGGCUGCAGCAACGCCUGCGAAACGCGCAGGCCGAAGCGGAUGCGGAGGUCAAAAGCGGUAACGUGUCGUGCAGCGUGCUGGAUCCGGUU (((..((((......((((((((....)))))))).(((((.(((.((((..(((((...((((.....(((......))).....))))..)))))..))))))))))))))))))).. ( -49.50) >DroYak_CAF1 4946 120 - 1 CCGUUGGUCACAUAUGGCUACUGCGAACAGUGGCUGCAGCAACGCCUGCGAAACGCGCAGGCUGAAGCGGAUGCGGAGGUCAAGAGCGGCAACGUGUCGUGCAGCGUGCUGGAUCCGGUA (((..((((......((((((((....)))))))).(((((..(((((((.....)))))))....(((((((((...(((......)))..)))))).)))....)))))))))))).. ( -51.20) >consensus CCGUUGGUCACGUAUGGCUACUGCGAACAGUGGCUGCAGCAACGCCUGCGAAACGCGCAGGCUGAAGCGGAUGCGGAGGUCAAGAGCGGCAACGUGUCGUGCAGCGUGCUGGAUCCAGUA ((((((....)).)))).(((((.((.(((((((((((((..((((((((.....))))))).)..)).((((((...(((......)))..)))))).)))))).)))))..))))))) (-46.32 = -46.00 + -0.32)


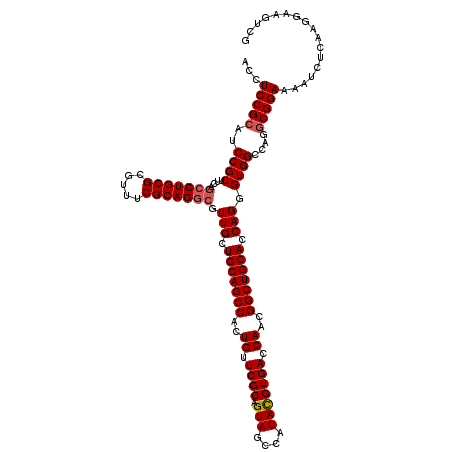
| Location | 19,483,765 – 19,483,885 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 94.92 |
| Mean single sequence MFE | -47.74 |
| Consensus MFE | -44.32 |
| Energy contribution | -44.76 |
| Covariance contribution | 0.44 |
| Combinations/Pair | 1.03 |
| Mean z-score | -1.55 |
| Structure conservation index | 0.93 |
| SVM decision value | 1.70 |
| SVM RNA-class probability | 0.972705 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 19483765 120 + 22224390 ACCUCCGCGUCCGCUUCAAUCUGCGCGUUUCGCAGUCGUUGCUGCAGCCACUGUUCGCAGUAGCCAUACGUGACCAACGGCUGCACCAGGGUGGCCAGGCGGAAAAUCUCAAGGAAGUCG ...(((((.((((((.....(((((.....)))))...(((.(((((((..((.((((.(((....))))))).))..))))))).))))))))..).)))))................. ( -41.10) >DroSec_CAF1 5750 120 + 1 GCCUCCGCAUCCGCUUCAGCCUGCGCAUUUCGCAGGCGUUGCUGCAGCCACUGUUCGCAGUAGCCAUACGUGAUCAACGGCUGCACCAGGGUGGCCAGGCGGAAAAUCUCAAGGAAGUCG ...(((((..(((((...(((((((.....))))))).(((.(((((((..((.((((.(((....))))))).))..))))))).))))))))....)))))................. ( -49.50) >DroSim_CAF1 6700 120 + 1 ACCUCCGCGUCCGCUUCAGCCUGCGCAUUUCGCAGGCGUUGCUGCAGCCACUGUUCGCAGUAGCCAUACGUGAUCAACGGCUGCACCAGGGUGGCCAGGCGGAAAAUCUCAAGGAAGUCG ...(((((.((((((...(((((((.....))))))).(((.(((((((..((.((((.(((....))))))).))..))))))).))))))))..).)))))................. ( -49.10) >DroEre_CAF1 5431 120 + 1 ACCUCCGCAUCCGCUUCGGCCUGCGCGUUUCGCAGGCGUUGCUGCAGCCACUGUUCGCAGUAGCCAUACGUGACCAACGGCUGCACCAGCGUGGCCAGACGGAAAAUUUCAAGGAAGUCG ...(((((..((((....(((((((.....)))))))((((.(((((((..((.((((.(((....))))))).))..))))))).))))))))...).))))................. ( -48.40) >DroYak_CAF1 4986 120 + 1 ACCUCCGCAUCCGCUUCAGCCUGCGCGUUUCGCAGGCGUUGCUGCAGCCACUGUUCGCAGUAGCCAUAUGUGACCAACGGCUGCACCAGUGUGGCCAGGCGGAAAAUCUCGAGGAAGUCG .((((....((((((((((((((((.....)))))))(((((((((((....))).))))))))......))).....(((..((....))..))).)))))).......))))...... ( -50.60) >consensus ACCUCCGCAUCCGCUUCAGCCUGCGCGUUUCGCAGGCGUUGCUGCAGCCACUGUUCGCAGUAGCCAUACGUGACCAACGGCUGCACCAGGGUGGCCAGGCGGAAAAUCUCAAGGAAGUCG ...(((((..((((....(((((((.....))))))).(((.(((((((..((.((((.(((....))))))).))..))))))).))).))))....)))))................. (-44.32 = -44.76 + 0.44)


Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 12:48:20 2006