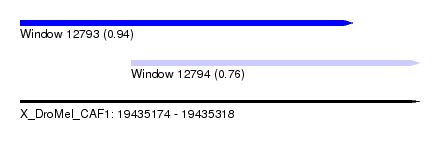
| Sequence ID | X_DroMel_CAF1 |
|---|---|
| Location | 19,435,174 – 19,435,318 |
| Length | 144 |
| Max. P | 0.939470 |

| Location | 19,435,174 – 19,435,294 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 80.56 |
| Mean single sequence MFE | -39.59 |
| Consensus MFE | -27.72 |
| Energy contribution | -27.95 |
| Covariance contribution | 0.23 |
| Combinations/Pair | 1.24 |
| Mean z-score | -2.63 |
| Structure conservation index | 0.70 |
| SVM decision value | 1.28 |
| SVM RNA-class probability | 0.939470 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 19435174 120 + 22224390 UUAGCUGGCUGUCCAGCUUCUUCACGCGCUUCAAGUACUCCGGAUGCACCAGCUGGCCCAGUUCCUCCAUCUGCUUCUGCAGGUUGAACAGCUUGUGCUGGUACAUUCUACGGCAGUGAA ..((((((....))))))...((((..(((....(((....(((((.((((((.(((...((((....((((((....)))))).)))).)))...)))))).))))))))))).)))). ( -42.30) >DroVir_CAF1 12870 120 + 1 UUAGCUGAUUGUCCAGCUUGCGUACACGCUUCAUAUAUUCCGGAUGUAUCAGCUGAUUCAGCUCCUCCAGCUGCUUCUGCAGAUUAAUUAGCUUGUGCUGGUACAUUCUGCAAAACAAAA ..(((((......))))).(((....)))............((((((((((((......((((.......((((....)))).......))))...))))))))))))............ ( -35.64) >DroPse_CAF1 22057 120 + 1 UCAGCUGGUUGUCCAGCUUCUUAACUCGUUUUAUAUACUCUGGAUGGAUGAGCUGAUUCAGUUCCUCCAUUUGCUUUUGCAGAUUGAGAAGCUUGUGCUGGUACAUUCUGUGGAAAAGGG ..((((((....))))))...................(((((((((((.((((((...)))))).)))))))..(((..((((.((...(((....)))....)).))))..))).)))) ( -38.50) >DroGri_CAF1 7203 120 + 1 UCAGCUGGUUGUCCAGCUUGCGCAAACGCUUCAUAUACUCCGGGUGCAUUAGCUGAUUCAGCUCCUCCAGCUGCUUCUGCAGAUUGAUCAGUUUGUGCUGGUACAUUCUGCAAAAACGCA ..((((((....))))))(((((...((............)).)))))...((.((..(((((.....)))))..))((((((.((((((((....)))))).)).)))))).....)). ( -40.40) >DroEre_CAF1 6207 120 + 1 UCAGCUGGUUGUCCAGCUUCUUCACGCGCUUUAUGUACUCCGGGUGCACCAGCUGGCUUAGUUCUUCCAUCUGCUUCUGCAGGUUCAGCAGCUUGUGCUGAUACAUUCUGCGGGAGUUAA ((((((((((..((.((........))((.....)).....))..).)))))))))................(((((((((((.((((((.....)))))).....)))))))))))... ( -42.20) >DroPer_CAF1 24125 120 + 1 UCAGCUGGUUGUCCAGCUUCUUAACGCGUUUUAUAUACUCUGGAUGGAUGAGCUGAUUCAGUUCCUCCAUUUGCUUUUGCAGAUUGAGAAGCUUGUGCUGGUACAUUCUGUGGAAAAGGG ..((((((....))))))...................(((((((((((.((((((...)))))).)))))))..(((..((((.((...(((....)))....)).))))..))).)))) ( -38.50) >consensus UCAGCUGGUUGUCCAGCUUCUUCACGCGCUUCAUAUACUCCGGAUGCAUCAGCUGAUUCAGUUCCUCCAUCUGCUUCUGCAGAUUGAGCAGCUUGUGCUGGUACAUUCUGCGGAAAAGAA ..((((((....)))))).......................(((((.((((((......((((..((.((((((....)))))).))..))))...)))))).)))))............ (-27.72 = -27.95 + 0.23)
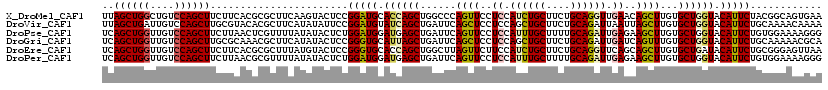
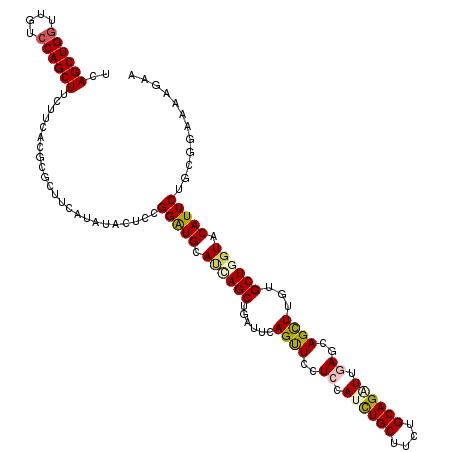
| Location | 19,435,214 – 19,435,318 |
|---|---|
| Length | 104 |
| Sequences | 6 |
| Columns | 108 |
| Reading direction | forward |
| Mean pairwise identity | 77.83 |
| Mean single sequence MFE | -33.35 |
| Consensus MFE | -25.83 |
| Energy contribution | -25.45 |
| Covariance contribution | -0.38 |
| Combinations/Pair | 1.31 |
| Mean z-score | -1.14 |
| Structure conservation index | 0.77 |
| SVM decision value | 0.51 |
| SVM RNA-class probability | 0.763470 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 19435214 104 + 22224390 CGGAUGCACCAGCUGGCCCAGUUCCUCCAUCUGCUUCUGCAGGUUGAACAGCUUGUGCUGGUACAUUCUACGGCAGUGAAAUGAUUAUGGAUCGA--AGAUAGG--AG .(((((.((((((.(((...((((....((((((....)))))).)))).)))...)))))).)))))..((((.((((.....)))).).))).--.......--.. ( -30.20) >DroSec_CAF1 2842 104 + 1 CGGGUGCACCAGCUGGCCCAGUUCCUCAAUCUGCUUCUGCAGAUUGAGCAGCUUGUGCUGGUACAUUCUGCGGCAGAGAAAUGAUGAUGGAUCGA--AGAUAGG--AG .(((((.((((((....(.((((.((((((((((....)))))))))).)))).).)))))).))))).............((((.....)))).--.......--.. ( -35.00) >DroSim_CAF1 3194 104 + 1 CGGGUGCACCAGCUGGCCCAGUUCCUCAAUCUGCUUCUGCAGGUUGAGCAGCUUGUGCUGGUACAUUCUGCGGCAGAGAAAUGAUGAUGGAUCGA--AGAUGGG--AG .((((((....))..))))..((((((..((((((...((((..(((.((((....)))).).))..))))))))))....((((.....)))).--.)).)))--). ( -34.50) >DroEre_CAF1 6247 106 + 1 CGGGUGCACCAGCUGGCUUAGUUCUUCCAUCUGCUUCUGCAGGUUCAGCAGCUUGUGCUGAUACAUUCUGCGGGAGUUAAAUGACAAUGAGAGGCAGAAGUAAG--AA (((.((...)).))).((((.((((.((.((((((((((((((.((((((.....)))))).....)))))))))))............))))).)))).))))--.. ( -35.20) >DroYak_CAF1 3970 101 + 1 CGGGUGCACCAGUUGGCCCAGUUCCUCCAUCUGCUUCUGCAGGUUCAGCAGCUUGUGCUGGUACAUUCUGCGGGAGUGAAAUUAUUUUGAUA-----AAGUAGG--AA .((((((....))..))))..(((((.(.((.(((((((((((.((((((.....)))))).....)))))))))))))..((((....)))-----).).)))--)) ( -31.50) >DroPer_CAF1 24165 105 + 1 UGGAUGGAUGAGCUGAUUCAGUUCCUCCAUUUGCUUUUGCAGAUUGAGAAGCUUGUGCUGGUACAUUCUGUGGAAAAGGGAA---CACAGAAGGCGUAAGCAAUUUCA .(((((((.((((((...)))))).)))))))((....))....(((((.(((((((((......(((((((..........---))))))))))))))))..))))) ( -33.70) >consensus CGGGUGCACCAGCUGGCCCAGUUCCUCCAUCUGCUUCUGCAGGUUGAGCAGCUUGUGCUGGUACAUUCUGCGGCAGAGAAAUGAUGAUGGAACGA__AAAUAGG__AA .(((((.((((((..((..((((.(((.((((((....)))))).))).)))).)))))))).)))))........................................ (-25.83 = -25.45 + -0.38)


Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 12:47:48 2006