
| Sequence ID | X_DroMel_CAF1 |
|---|---|
| Location | 19,409,755 – 19,409,860 |
| Length | 105 |
| Max. P | 0.866810 |

| Location | 19,409,755 – 19,409,860 |
|---|---|
| Length | 105 |
| Sequences | 6 |
| Columns | 110 |
| Reading direction | forward |
| Mean pairwise identity | 82.25 |
| Mean single sequence MFE | -54.37 |
| Consensus MFE | -33.26 |
| Energy contribution | -33.73 |
| Covariance contribution | 0.48 |
| Combinations/Pair | 1.31 |
| Mean z-score | -2.74 |
| Structure conservation index | 0.61 |
| SVM decision value | 0.64 |
| SVM RNA-class probability | 0.809415 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
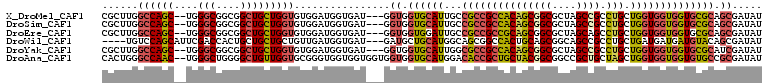
>X_DroMel_CAF1 19409755 105 + 22224390 CGCUUGGCCAGC--UGGGCGGCGGCUGCUGGUGUGGAUGGUGAU---GGUGGUGCAUUGCCGCCGCCACAGCGGCGCUAGCCGCCUGCUGGUGGUGGUGCGCAGCGAUAU ((((..((((((--.(((((((.(((((((..((((.(((..((---(......)))..)))))))..)))))))....))))))))))))).(((...))))))).... ( -59.30) >DroSim_CAF1 9847 105 + 1 CGCUUGGCCAGC--UGGGCGGCGGCUGCUGGUGUGGAUGGUGAU---GGUGGUGCAUUGCCGCCGCCACAGCGGCGCUAGCCGCCUGCUGGUGGUGGUGCGCAGCGAUAU ((((..((((((--.(((((((.(((((((..((((.(((..((---(......)))..)))))))..)))))))....))))))))))))).(((...))))))).... ( -59.30) >DroEre_CAF1 9602 105 + 1 CGCUUGGCCAGC--UGGGCGGCGGCUGCUGGUGUGGAUGGUGAU---GGUGGUGGAUUGCCGCCGCCGCAGCGGCGCUAGCAGCCUGCUGGUGGUGGUGCGCAGCGAUAU ((((..(....)--..))))..((((((((((((...((.((.(---((((((((....))))))))))).))))))))))))))(((((((......)).))))).... ( -57.40) >DroWil_CAF1 4044 103 + 1 ----UGUCCAGCAUUCGACCACUGCUGCUGCUGUUGAUGGUGAU---GAUGCUGCAUGGCAGCGGCCACUGCAGCGGCAGCCGCCUGCUGAUGAUGAUGUACAGCGAUAU ----....(((((..((......(((((((((((.(.((((...---..((((((...))))))))))).)))))))))))))..))))).................... ( -39.00) >DroYak_CAF1 9784 105 + 1 CGCUUGGCCAGC--UGGGCGGCGGCUGCUGGUGUGGAUGGUGAU---GGUGGUGCAUUGGCGCCGCCACAGCGGCGCUAGCCGCCUGCUGGUGGUGGUGCGCAUCGAUAU (((.(.((((((--.((((((((((.((((.(((((.(((((..---((((...))))..)))))))))).))))))).))))))))))))).)....)))......... ( -56.80) >DroAna_CAF1 9093 108 + 1 CACUGGGCCAAC--UGGGCUGGGGCUGUUGGUGCGGGUGGUGGUGGUGGUGGUGCAUGGACACCGCUGCUACGGCGGCCGCUGCUAGCUGGUGGUGGUGUGCCGCGAUAU ((((.(((((((--..(((....))))))))).).))))((((..((((((.........))))))..)))).((((((((..(((.....)))..))).)))))..... ( -54.40) >consensus CGCUUGGCCAGC__UGGGCGGCGGCUGCUGGUGUGGAUGGUGAU___GGUGGUGCAUUGCCGCCGCCACAGCGGCGCUAGCCGCCUGCUGGUGGUGGUGCGCAGCGAUAU ......((((((....(((....)))))))))................((.((((((...(((((((((((((((....)))).))).)))))))))))))).))..... (-33.26 = -33.73 + 0.48)
| Location | 19,409,755 – 19,409,860 |
|---|---|
| Length | 105 |
| Sequences | 6 |
| Columns | 110 |
| Reading direction | reverse |
| Mean pairwise identity | 82.25 |
| Mean single sequence MFE | -40.13 |
| Consensus MFE | -29.50 |
| Energy contribution | -30.06 |
| Covariance contribution | 0.56 |
| Combinations/Pair | 1.35 |
| Mean z-score | -2.46 |
| Structure conservation index | 0.74 |
| SVM decision value | 0.85 |
| SVM RNA-class probability | 0.866810 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
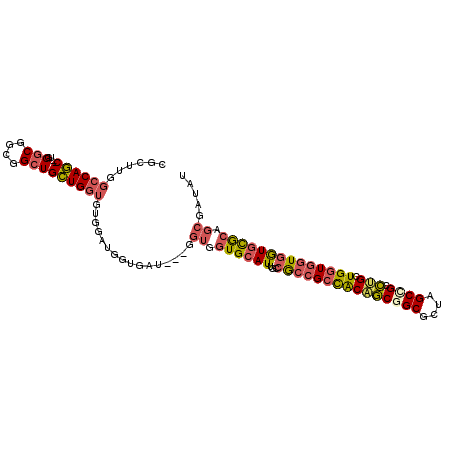
>X_DroMel_CAF1 19409755 105 - 22224390 AUAUCGCUGCGCACCACCACCAGCAGGCGGCUAGCGCCGCUGUGGCGGCGGCAAUGCACCACC---AUCACCAUCCACACCAGCAGCCGCCGCCCA--GCUGGCCAAGCG ....((((((............))).)))(((.(.(((((((.(((((((((..(((......---................))))))))))))))--)).)))).))). ( -45.75) >DroSim_CAF1 9847 105 - 1 AUAUCGCUGCGCACCACCACCAGCAGGCGGCUAGCGCCGCUGUGGCGGCGGCAAUGCACCACC---AUCACCAUCCACACCAGCAGCCGCCGCCCA--GCUGGCCAAGCG ....((((((............))).)))(((.(.(((((((.(((((((((..(((......---................))))))))))))))--)).)))).))). ( -45.75) >DroEre_CAF1 9602 105 - 1 AUAUCGCUGCGCACCACCACCAGCAGGCUGCUAGCGCCGCUGCGGCGGCGGCAAUCCACCACC---AUCACCAUCCACACCAGCAGCCGCCGCCCA--GCUGGCCAAGCG .....((((.(........))))).....(((.(.(((((((.(((((((((...........---...................)))))))))))--)).)))).))). ( -41.31) >DroWil_CAF1 4044 103 - 1 AUAUCGCUGUACAUCAUCAUCAGCAGGCGGCUGCCGCUGCAGUGGCCGCUGCCAUGCAGCAUC---AUCACCAUCAACAGCAGCAGCAGUGGUCGAAUGCUGGACA---- .....((((...........)))).((((((.(((((....))))).))))))...((((((.---.(((((((.....((....)).))))).))))))))....---- ( -39.10) >DroYak_CAF1 9784 105 - 1 AUAUCGAUGCGCACCACCACCAGCAGGCGGCUAGCGCCGCUGUGGCGGCGCCAAUGCACCACC---AUCACCAUCCACACCAGCAGCCGCCGCCCA--GCUGGCCAAGCG .........(((.......(((((.((((((..(((((((....)))))))...(((......---................)))...))))))..--)))))....))) ( -39.15) >DroAna_CAF1 9093 108 - 1 AUAUCGCGGCACACCACCACCAGCUAGCAGCGGCCGCCGUAGCAGCGGUGUCCAUGCACCACCACCACCACCACCCGCACCAACAGCCCCAGCCCA--GUUGGCCCAGUG .....((((.............(((.((.((((...)))).)))))(((((....)))))..............)))).(((((.((....))...--)))))....... ( -29.70) >consensus AUAUCGCUGCGCACCACCACCAGCAGGCGGCUAGCGCCGCUGUGGCGGCGGCAAUGCACCACC___AUCACCAUCCACACCAGCAGCCGCCGCCCA__GCUGGCCAAGCG ...................(((((.((((((....))))))..(((((((((..(((.........................))))))))))))....)))))....... (-29.50 = -30.06 + 0.56)


Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 12:47:37 2006