
| Sequence ID | X_DroMel_CAF1 |
|---|---|
| Location | 19,392,957 – 19,393,057 |
| Length | 100 |
| Max. P | 0.997411 |

| Location | 19,392,957 – 19,393,057 |
|---|---|
| Length | 100 |
| Sequences | 6 |
| Columns | 110 |
| Reading direction | forward |
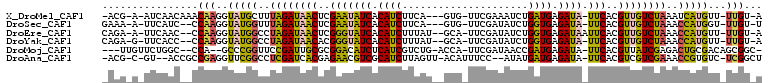
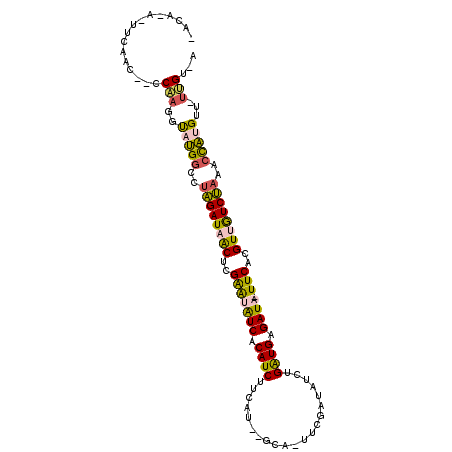
| Mean pairwise identity | 67.78 |
| Mean single sequence MFE | -32.35 |
| Consensus MFE | -18.44 |
| Energy contribution | -17.58 |
| Covariance contribution | -0.86 |
| Combinations/Pair | 1.41 |
| Mean z-score | -3.51 |
| Structure conservation index | 0.57 |
| SVM decision value | 2.85 |
| SVM RNA-class probability | 0.997411 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 19392957 100 + 22224390 -ACG-A-AUCAACAAACAAGGUAUGCUUUAGAUAACUCGAAUAUCACAUCUUCA---GUG-UUCGAAAUCUGAUGAGAUA-UUCACGUUGUCUAAAUCAUGUU-UUGU-A -...-.-........((((..((((.((((((((((..(((((((.((((....---...-..........)))).))))-)))..)))))))))).))))..-))))-. ( -30.63) >DroSec_CAF1 26110 99 + 1 GAAA-A-UUCAUC--CCAAGGUAUGGUUUAGAUAACUCGAAUAUCACAUCUUCA---GUG-UUCGAUAUCUGGUGAGAUA-UUCACGUUGUCUAAACCAUGGU-UUGU-U ....-.-......--.((((.(((((((((((((((..(((((((.((((....---...-..........)))).))))-)))..))))))))))))))).)-))).-. ( -34.73) >DroEre_CAF1 24689 101 + 1 CAGA-A-UUCAAC--CCAAGGUAUGGCCUAGAUAACUCGGGUAUCACAUCUUUAU--GCA-UUCGAUAUCUGGUGAGAUAAUUCACGUUGUCUAAACCAUGUU-UUGU-A ....-.-......--.(((..(((((..((((((((.((((((((..........--...-...))))))))(((((....)))))))))))))..)))))..-))).-. ( -29.29) >DroYak_CAF1 24036 100 + 1 CAGA-G-UUCACC--CCAAGGUAUGGCCUAGAUAACACGGGUAUCACAUCUUUAU--GCA-UUCGAUAUCUGGUGAGAUA-UUCACGUUGUCUAAACCAUGUU-UUGU-A ....-.-......--.(((..(((((..((((((((.((((((((..........--...-...))))))))(((((...-)))))))))))))..)))))..-))).-. ( -29.79) >DroMoj_CAF1 32524 99 + 1 ---UUGUUCUGGC--CCA--GCCCGGUUCCGAUUGCGCGGACAUCUCAUCGUCUG-ACCA-UUCGAUAACCGAUGAGAUA-UUCACGUUAUCGAGACUGCGACAGCGGC- ---........((--((.--.(.((((..((((.(((.(((.(((((((((..((-.(..-...).))..))))))))).-))).))).))))..)))).)...).)))- ( -32.30) >DroAna_CAF1 37408 100 + 1 -ACG-C-GU--ACCGCCGAGGUUCGGCCUCGAUCACGAGAACGUCGCAUCUUAGUU-ACAUUUCC--AUAUGAUGAGAUA-UUCACGUCGUCGAAACCGUGUC-UCGGCU -...-.-..--...(((((((..(((..(((((.(((.(((..((.((((......-........--....)))).))..-))).))).)))))..)))..))-))))). ( -37.37) >consensus _ACA_A_UUCAAC__CCAAGGUAUGGCCUAGAUAACUCGAAUAUCACAUCUUCAU__GCA_UUCGAUAUCUGAUGAGAUA_UUCACGUUGUCUAAACCAUGUU_UUGU_A ................(((..(((((..((((((((..(((((((.((((.....................)))).)))).)))..))))))))..)))))...)))... (-18.44 = -17.58 + -0.86)

| Location | 19,392,957 – 19,393,057 |
|---|---|
| Length | 100 |
| Sequences | 6 |
| Columns | 110 |
| Reading direction | reverse |
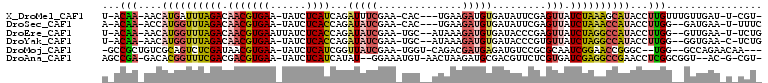
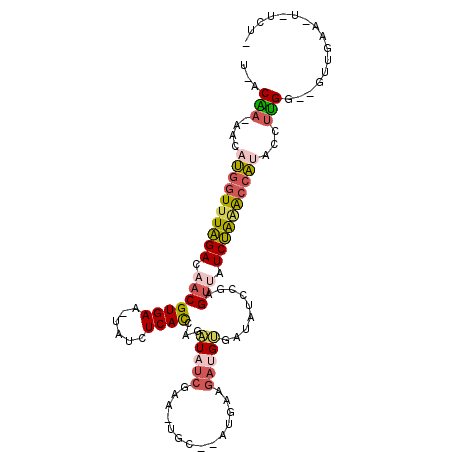
| Mean pairwise identity | 67.78 |
| Mean single sequence MFE | -28.88 |
| Consensus MFE | -14.78 |
| Energy contribution | -14.84 |
| Covariance contribution | 0.06 |
| Combinations/Pair | 1.36 |
| Mean z-score | -2.29 |
| Structure conservation index | 0.51 |
| SVM decision value | 2.20 |
| SVM RNA-class probability | 0.990122 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 19392957 100 - 22224390 U-ACAA-AACAUGAUUUAGACAACGUGAA-UAUCUCAUCAGAUUUCGAA-CAC---UGAAGAUGUGAUAUUCGAGUUAUCUAAAGCAUACCUUGUUUGUUGAU-U-CGU- .-((((-...(((.((((((.(((.((((-((((.((((....((((..-...---)))))))).)))))))).))).)))))).)))...))))........-.-...- ( -23.60) >DroSec_CAF1 26110 99 - 1 A-ACAA-ACCAUGGUUUAGACAACGUGAA-UAUCUCACCAGAUAUCGAA-CAC---UGAAGAUGUGAUAUUCGAGUUAUCUAAACCAUACCUUGG--GAUGAA-U-UUUC .-.((.-.((((((((((((.(((.((((-((((.((.(((........-..)---))....)).)))))))).))).))))))))).....)))--..))..-.-.... ( -27.30) >DroEre_CAF1 24689 101 - 1 U-ACAA-AACAUGGUUUAGACAACGUGAAUUAUCUCACCAGAUAUCGAA-UGC--AUAAAGAUGUGAUACCCGAGUUAUCUAGGCCAUACCUUGG--GUUGAA-U-UCUG .-.(((-..(((((((((((.(((((((......))))......(((..-..(--(((....)))).....)))))).))))))))).....)).--.)))..-.-.... ( -22.00) >DroYak_CAF1 24036 100 - 1 U-ACAA-AACAUGGUUUAGACAACGUGAA-UAUCUCACCAGAUAUCGAA-UGC--AUAAAGAUGUGAUACCCGUGUUAUCUAGGCCAUACCUUGG--GGUGAA-C-UCUG .-....-...((((((((((.(((((((.-....))))...(((((...-...--.....))))).........))).)))))))))).....((--((....-)-))). ( -23.60) >DroMoj_CAF1 32524 99 - 1 -GCCGCUGUCGCAGUCUCGAUAACGUGAA-UAUCUCAUCGGUUAUCGAA-UGGU-CAGACGAUGAGAUGUCCGCGCAAUCGGAACCGGGC--UGG--GCCAGAACAA--- -(((.(.(((.(.((..((((..((((.(-((((((((((...(((...-.)))-....))))))))))).))))..))))..)).))))--.))--))........--- ( -36.70) >DroAna_CAF1 37408 100 - 1 AGCCGA-GACACGGUUUCGACGACGUGAA-UAUCUCAUCAUAU--GGAAAUGU-AACUAAGAUGCGACGUUCUCGUGAUCGAGGCCGAACCUCGGCGGU--AC-G-CGU- .(((((-(...(((((((((..(((.(((-..((.((((((((--....))))-......)))).))..))).)))..)))))))))...))))))...--..-.-...- ( -40.10) >consensus U_ACAA_AACAUGGUUUAGACAACGUGAA_UAUCUCACCAGAUAUCGAA_UGC__AUGAAGAUGUGAUAUCCGAGUUAUCUAAACCAUACCUUGG__GUUGAA_U_UCU_ ...(((....((((((((((.(((((((......))))...(((((..............))))).........))).))))))))))...)))................ (-14.78 = -14.84 + 0.06)
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 12:47:35 2006