
| Sequence ID | X_DroMel_CAF1 |
|---|---|
| Location | 19,392,684 – 19,392,786 |
| Length | 102 |
| Max. P | 0.995668 |

| Location | 19,392,684 – 19,392,786 |
|---|---|
| Length | 102 |
| Sequences | 6 |
| Columns | 118 |
| Reading direction | forward |
| Mean pairwise identity | 71.78 |
| Mean single sequence MFE | -30.04 |
| Consensus MFE | -19.02 |
| Energy contribution | -19.00 |
| Covariance contribution | -0.02 |
| Combinations/Pair | 1.35 |
| Mean z-score | -2.54 |
| Structure conservation index | 0.63 |
| SVM decision value | 2.60 |
| SVM RNA-class probability | 0.995668 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
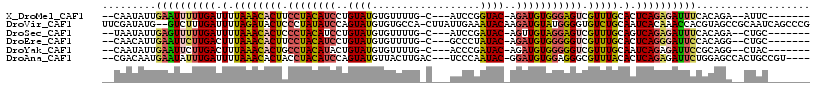
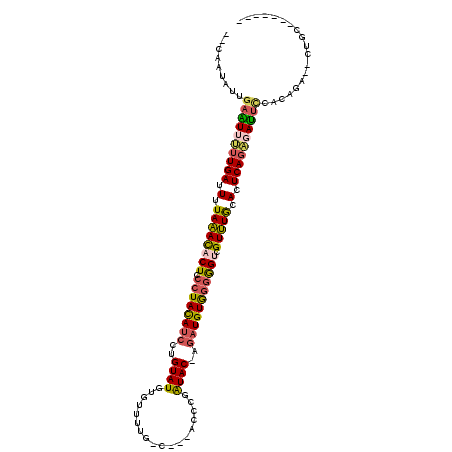
>X_DroMel_CAF1 19392684 102 + 22224390 --CAAUAUUGAAUUUUUGAUUUUAAACACUUCCUACAUCCUGUAUGUGUUUUG-C---AUCCGGUAC-AGAUGUGGGAGUCGUUUGCACUCAGAGAUUUCACAGA--AUUC------- --.......((((((((((.(.((((((((.((((((((.((((((((.....-)---))...))))-)))))))))))).))))).).))))))))))......--....------- ( -29.60) >DroVir_CAF1 40909 115 + 1 UUCGAUAUG--GUCUUUGAUUUUAGAUACUCCCUAUAUCCAGUAUGUGUGCCA-CUUAUUGAAAUACAAGAUGUAUGGGGUGUCUGCAAUCACAAACCACGUAGCCGCAAUCAGCCCG ...(((.((--((.(.(((((.((((((((((.(((((((((((.(((...))-).)))))........)))))).)))))))))).))))).).)))).((....)).)))...... ( -31.20) >DroSec_CAF1 25857 102 + 1 --UAAUAUUGAGUUUUUGAUUUUAAACACUCCCUACAUCCUGUAUGUGUUUUG-C---AUCCGAUAC-AGUUGUAGGAGUCGUUUGCAGUCAGAGAUUUCACAGA--CUGC------- --.......((((((((((((.((((((((((.((((..(((((((((.....-)---))...))))-)).))))))))).))))).))))))))))))......--....------- ( -31.10) >DroEre_CAF1 24386 102 + 1 --CAACAUUGAAUUCUUGACUUUAAACACUUCCUACAUCCUGUAUGUGUUUUG-C---GCCCUAUAC-AGAUGUGGGGGUCGUUUGCACUCAGGGAUUCCACAGG--CUGC------- --.......((((((((((.(.((((((((.((((((((.((((((((.....-)---))...))))-)))))))))))).))))).).))))))))))......--....------- ( -33.10) >DroYak_CAF1 23718 102 + 1 --CAAUAUUGAAUUCUUGACUUUAAACACUGCCUACAUACUGUAUGUGUUUUG-C---ACCCGAUAC-AGAUGUGGGGGUCGUUUGCAAUCAGAGAUUCCGCAGG--CUAC------- --.......((((..((((.(.((((((((.(((((((.(((((((((.....-)---))...))))-)))))))))))).))))).).))))..))))......--....------- ( -28.80) >DroAna_CAF1 37162 108 + 1 --CGACAAUGAAUAUUUGAUUUUAAACACUACCUACAUCCAGUAUGUUACUUGAC---UCCCAAUAC-GGAUGUGGAGGGCGUUUACACUCAGAGAUUCUGGAGCCACUGCCGU---- --.......((((.(((((.(.(((((.((..((((((((.((((..........---.....))))-))))))))..)).))))).).))))).)))).((........))..---- ( -26.46) >consensus __CAAUAUUGAAUUUUUGAUUUUAAACACUCCCUACAUCCUGUAUGUGUUUUG_C___ACCCGAUAC_AGAUGUGGGGGUCGUUUGCACUCAGAGAUUCCACAGA__CUGC_______ .........((((((((((.(.((((((((.((((((((..((((..................))))..))))))))))).))))).).))))))))))................... (-19.02 = -19.00 + -0.02)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 12:47:32 2006