| Sequence ID | X_DroMel_CAF1 |
|---|---|
| Location | 19,151,712 – 19,151,822 |
| Length | 110 |
| Max. P | 0.935709 |

| Location | 19,151,712 – 19,151,822 |
|---|---|
| Length | 110 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 77.71 |
| Mean single sequence MFE | -25.82 |
| Consensus MFE | -8.81 |
| Energy contribution | -11.01 |
| Covariance contribution | 2.20 |
| Combinations/Pair | 1.00 |
| Mean z-score | -2.31 |
| Structure conservation index | 0.34 |
| SVM decision value | 1.25 |
| SVM RNA-class probability | 0.935709 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
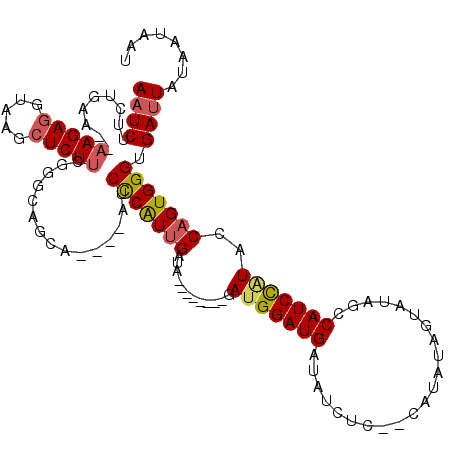
>X_DroMel_CAF1 19151712 110 + 22224390 AUUAUUAUAAUCACCCACUGGUAUGGAUGGCUAUACUAUAUG-GGAGAUAUCAUCCAUC--------UAUCAAUGAGAUACAUAUUUGCCCGAAGAGCUUACCUCUU-UUUCAGAAGAUU .........(((.((((.((((((((....))))))))..))-)).)))(((((((((.--------.....))).))).....((((...((((((.....)))))-)..)))).))). ( -22.40) >DroSec_CAF1 8293 108 + 1 AUUAUUAUAAUCACCCACUGGUAUAGAUGGAUAUACUAUAUA--GAGAUAUCAUCCAUCCAUC----UCUCAAUGGGAU----UGCUGCCCGAAGAGCUUAGCUCUU--UUCAGAAGAUU ........((((.((((.(((...((((((((.....((((.--...)))).....)))))))----).))).))))..----..(((...(((((((...))))))--).)))..)))) ( -27.20) >DroSim_CAF1 6261 114 + 1 AUUAUUAUAAUCACCCACUGGUAUGGAUGGAUAUACUAUAUG--GAGCUAUCAUCCAUCCAUCUAUCUAUCAAUGGGAU----UGCUGCCCGAAGAGCUUAGCUCUUUUUUCAGAAGAUU ........((((.((((.(((((((((((((((((((.....--.)).))).)))))))))......))))).))))..----..(((...(((((((...)))))))...)))..)))) ( -33.60) >DroEre_CAF1 11426 99 + 1 ACUAUCAUAAUCACCCACUGGUACGGAUGGCUAU-----AUG--GAGAUAGCAUCCAUC--------CAUCAAUGGGAA----AGCGCCACGAAGAGCUUACGUCUU--UUCAGAAGAUU ........((((.((((.((((..((((((((((-----...--...)))))...))))--------))))).))))..----........(((((((....).)))--)))....)))) ( -25.20) >DroYak_CAF1 8533 91 + 1 ACUAUUAUAUUCACCCACUGGUAGGGAUGUCUAU-----AUGAAGAGAUAUCAUCCAUC--------UAUCCACGAGAU--------------AGAGCUUACCUCUU--UUCAGAAGAUU ...................(((((((((((((..-----......))))))).....((--------((((.....)))--------------))).))))))((((--(...))))).. ( -20.70) >consensus AUUAUUAUAAUCACCCACUGGUAUGGAUGGCUAUACUAUAUG__GAGAUAUCAUCCAUC________UAUCAAUGGGAU____AGCUGCCCGAAGAGCUUACCUCUU__UUCAGAAGAUU ........((((.((((.(((((((((((......................)))))))..........)))).))))...............(((((.....))))).........)))) ( -8.81 = -11.01 + 2.20)



| Location | 19,151,712 – 19,151,822 |
|---|---|
| Length | 110 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 77.71 |
| Mean single sequence MFE | -27.66 |
| Consensus MFE | -13.39 |
| Energy contribution | -13.71 |
| Covariance contribution | 0.32 |
| Combinations/Pair | 1.17 |
| Mean z-score | -2.22 |
| Structure conservation index | 0.48 |
| SVM decision value | 1.10 |
| SVM RNA-class probability | 0.914761 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 19151712 110 - 22224390 AAUCUUCUGAAA-AAGAGGUAAGCUCUUCGGGCAAAUAUGUAUCUCAUUGAUA--------GAUGGAUGAUAUCUCC-CAUAUAGUAUAGCCAUCCAUACCAGUGGGUGAUUAUAAUAAU ...(((((....-.)))))...((.(....)))...((((...(((((((...--------.(((((((.(((((..-.....)).)))..)))))))..)))))))....))))..... ( -24.80) >DroSec_CAF1 8293 108 - 1 AAUCUUCUGAA--AAGAGCUAAGCUCUUCGGGCAGCA----AUCCCAUUGAGA----GAUGGAUGGAUGAUAUCUC--UAUAUAGUAUAUCCAUCUAUACCAGUGGGUGAUUAUAAUAAU ((((.(((...--.)))(((..((.(....)))))).----..(((((((..(----((((((((....((((...--.))))....)))))))))....))))))).))))........ ( -32.10) >DroSim_CAF1 6261 114 - 1 AAUCUUCUGAAAAAAGAGCUAAGCUCUUCGGGCAGCA----AUCCCAUUGAUAGAUAGAUGGAUGGAUGAUAGCUC--CAUAUAGUAUAUCCAUCCAUACCAGUGGGUGAUUAUAAUAAU ((((..(((....((((((...))))))....)))..----..(((((((........(((((((((((...(((.--.....))).)))))))))))..))))))).))))........ ( -32.60) >DroEre_CAF1 11426 99 - 1 AAUCUUCUGAA--AAGACGUAAGCUCUUCGUGGCGCU----UUCCCAUUGAUG--------GAUGGAUGCUAUCUC--CAU-----AUAGCCAUCCGUACCAGUGGGUGAUUAUGAUAGU .....(((...--.)))(((((((.((....)).)).----..(((((((...--------.(((((((((((...--...-----)))).)))))))..)))))))...)))))..... ( -28.00) >DroYak_CAF1 8533 91 - 1 AAUCUUCUGAA--AAGAGGUAAGCUCU--------------AUCUCGUGGAUA--------GAUGGAUGAUAUCUCUUCAU-----AUAGACAUCCCUACCAGUGGGUGAAUAUAAUAGU .......(((.--.(((((((...(((--------------((((.......)--------))))))...))))))))))(-----(((..((((((.....).)))))..))))..... ( -20.80) >consensus AAUCUUCUGAA__AAGAGGUAAGCUCUUCGGGCAGCA____AUCCCAUUGAUA________GAUGGAUGAUAUCUC__CAUAUAGUAUAGCCAUCCAUACCAGUGGGUGAUUAUAAUAAU ((((.........(((((.....)))))...............(((((((............(((((((......................)))))))..))))))).))))........ (-13.39 = -13.71 + 0.32)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 12:45:04 2006