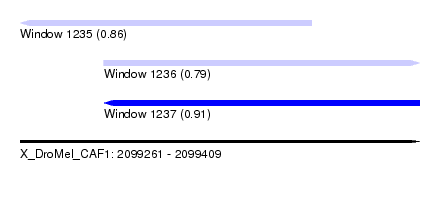
| Sequence ID | X_DroMel_CAF1 |
|---|---|
| Location | 2,099,261 – 2,099,409 |
| Length | 148 |
| Max. P | 0.907528 |

| Location | 2,099,261 – 2,099,369 |
|---|---|
| Length | 108 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 70.44 |
| Mean single sequence MFE | -53.94 |
| Consensus MFE | -25.58 |
| Energy contribution | -28.46 |
| Covariance contribution | 2.88 |
| Combinations/Pair | 1.11 |
| Mean z-score | -1.87 |
| Structure conservation index | 0.47 |
| SVM decision value | 0.84 |
| SVM RNA-class probability | 0.863800 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 2099261 108 - 22224390 GGACCUGG---ACCGGGUCCGUUGCGACGCAUAAAGAGCGGUGGCGGCGGCGGUGGUGGUCCACCCAUGGGGCCCAUGGGUCCGGGACCGCGCAUACC------AGG---CACCGGUGGC ((.(((((---((((...(((((((.((((.......)).)).))))))))))).(((((((((((((((...))))))))...))))))).....))------)))---..))...... ( -55.10) >DroPse_CAF1 27587 108 - 1 UGGAUUGGUGCACCGCGACCGUUGUGGCGCAUGAAGUGAGGCGG------UGGCGGUGGACCACCCAUCGGUGGCAUGGGG------CAACGCAUGGCCAUGGGCGGCACCAUCUGUGGU .....((((.((((((.((((((....(((.....))).)))))------).)))))).)))).((((.(((((((((.(.------...).))))(((......))).))))).)))). ( -55.60) >DroSim_CAF1 24070 102 - 1 GGACCUGG---ACCGGGUCCGUUGCGACGCAUAAAAAGUGGUGG------CGGUGGUGGUCCACCCAUGGGGCCCAUGGGUCCGGGACCGCGCAUACC------AGG---CAUCGGUGGC ((.(((((---(((((((((((..(.((.........)).)..)------)(((((....)))))....))))))...)))))))).))..((.((((------...---....)))))) ( -49.40) >DroYak_CAF1 25424 108 - 1 GGACCUGG---ACCGGGUCCGUUGCGCCGCUAAAAAUGCGGUGGUGGCGGCGGCGGUGGUCCACCCAUGGGGCCCAUGGGCCCGGGACCGCGCAUACC------GGG---CAUCGGUGGU ..(((...---(((((((((((..((((((.......))))).)..))((..(((.((((((.(((((((...)))))))....)))))))))...))------)))---).)))))))) ( -56.30) >DroPer_CAF1 27667 108 - 1 UGGAUUGGUGCACCGCGACCGUUGUGGCGCAUGAAGUGAGGCGG------UGGCGGUGGACCACCCAUCGGUGGCAUGGGG------CAACGCAUGGCCAUGGGCGGCACUAUCUGUGGU .....((((.((((((.((((((....(((.....))).)))))------).)))))).)))).((((.(((((((((.(.------...).))))(((......))).))))).)))). ( -53.30) >consensus GGACCUGG___ACCGGGUCCGUUGCGACGCAUAAAGUGAGGUGG______CGGCGGUGGUCCACCCAUGGGGCCCAUGGGGCCGGGACCGCGCAUACC______AGG___CAUCGGUGGU ((((((((....))))))))..(((..(((..........)))............(((((((.((((((.....))))))....)))))))))).......................... (-25.58 = -28.46 + 2.88)

| Location | 2,099,292 – 2,099,409 |
|---|---|
| Length | 117 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 78.29 |
| Mean single sequence MFE | -45.86 |
| Consensus MFE | -26.68 |
| Energy contribution | -30.68 |
| Covariance contribution | 4.00 |
| Combinations/Pair | 1.07 |
| Mean z-score | -1.92 |
| Structure conservation index | 0.58 |
| SVM decision value | 0.58 |
| SVM RNA-class probability | 0.788588 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 2099292 117 + 22224390 CCCAUGGGCCCCAUGGGUGGACCACCACCGCCGCCGCCACCGCUCUUUAUGCGUCGCAACGGACCCGGU---CCAGGUCCUAUGAUGGGUGUCCCACCGCCCAUGCACAUGAUGGGUCCA ....(((((((((((.(((((((...(((((((..((...(((.......)))..))..)))...))))---...)))))....(((((((......))))))))).))))..))))))) ( -47.70) >DroPse_CAF1 27621 111 + 1 CCCAUGCCACCGAUGGGUGGUCCACCGCCA------CCGCCUCACUUCAUGCGCCACAACGGUCGCGGUGCACCAAUCCACAUGAUGGGCGGUCCAC---CUAUGCACUUGAUGGGUCCU (((((.......((((((((.(((((((.(------(((....................)))).)))))((.(((.((.....))))))))).))))---)))).......))))).... ( -39.99) >DroSim_CAF1 24101 111 + 1 CCCAUGGGCCCCAUGGGUGGACCACCACCG------CCACCACUUUUUAUGCGUCGCAACGGACCCGGU---CCAGGUCCUAUGAUGGGUGUUCCGCCGCCCAUGCACAUGAUGGGUCCA ....(((((((((((((((((.((((...(------(.((((.......)).)).))...(((((....---...))))).......)))).))))))((....)).))))..))))))) ( -44.40) >DroYak_CAF1 25455 117 + 1 CCCAUGGGCCCCAUGGGUGGACCACCGCCGCCGCCACCACCGCAUUUUUAGCGGCGCAACGGACCCGGU---CCAGGUCCCAUGAUGGGUGGCCCACCGCCCAUGCAUAUGAUGGGUCCA ....(((((((((((((((((((.(((.((((((................))))))...)))....)))---)))...))))))((((((((....)))))))).........))))))) ( -56.59) >DroPer_CAF1 27701 111 + 1 CCCAUGCCACCGAUGGGUGGUCCACCGCCA------CCGCCUCACUUCAUGCGCCACAACGGUCGCGGUGCACCAAUCCACAUGAUGGGGGGUCCAC---CCAUGCACUUGAUGGGUCCU (((((((.((((.(((((((....))))).------.(((..........)))...)).)))).))((((((((.(((.....))).))(((....)---)).))))))..))))).... ( -40.60) >consensus CCCAUGGGCCCCAUGGGUGGACCACCGCCG______CCACCGCACUUUAUGCGCCGCAACGGACCCGGU___CCAGGUCCCAUGAUGGGUGGUCCACCGCCCAUGCACAUGAUGGGUCCA .....((((((....(((((....))))).....................((........(((((.((....)).)))))....((((((((....)))))))))).......)))))). (-26.68 = -30.68 + 4.00)

| Location | 2,099,292 – 2,099,409 |
|---|---|
| Length | 117 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 78.29 |
| Mean single sequence MFE | -54.42 |
| Consensus MFE | -31.30 |
| Energy contribution | -33.82 |
| Covariance contribution | 2.52 |
| Combinations/Pair | 1.06 |
| Mean z-score | -2.13 |
| Structure conservation index | 0.58 |
| SVM decision value | 1.05 |
| SVM RNA-class probability | 0.907528 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
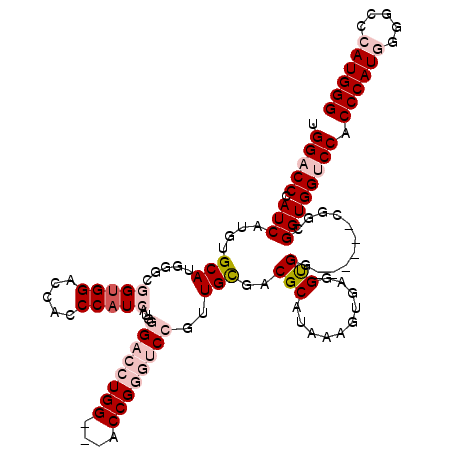
>X_DroMel_CAF1 2099292 117 - 22224390 UGGACCCAUCAUGUGCAUGGGCGGUGGGACACCCAUCAUAGGACCUGG---ACCGGGUCCGUUGCGACGCAUAAAGAGCGGUGGCGGCGGCGGUGGUGGUCCACCCAUGGGGCCCAUGGG ((((((.(((((.(((......((((((...))))))...(((((((.---..)))))))(((((.((((.......)).)).))))).))))))))))))))(((((((...))))))) ( -55.60) >DroPse_CAF1 27621 111 - 1 AGGACCCAUCAAGUGCAUAG---GUGGACCGCCCAUCAUGUGGAUUGGUGCACCGCGACCGUUGUGGCGCAUGAAGUGAGGCGG------UGGCGGUGGACCACCCAUCGGUGGCAUGGG ....(((((...((.(((.(---((((.((((.......))))..((((.((((((.((((((....(((.....))).)))))------).)))))).)))).))))).)))))))))) ( -50.10) >DroSim_CAF1 24101 111 - 1 UGGACCCAUCAUGUGCAUGGGCGGCGGAACACCCAUCAUAGGACCUGG---ACCGGGUCCGUUGCGACGCAUAAAAAGUGGUGG------CGGUGGUGGUCCACCCAUGGGGCCCAUGGG ((((((((((.((((.(((((.(......).)))))))))(((((((.---..)))))))((..(.((.........)).)..)------)))))..))))))(((((((...))))))) ( -51.70) >DroYak_CAF1 25455 117 - 1 UGGACCCAUCAUAUGCAUGGGCGGUGGGCCACCCAUCAUGGGACCUGG---ACCGGGUCCGUUGCGCCGCUAAAAAUGCGGUGGUGGCGGCGGCGGUGGUCCACCCAUGGGGCCCAUGGG ((((((.(((.((((.(((((.((....)).)))))))))(((((((.---..)))))))((((((((((((.........))))))).))))))))))))))(((((((...))))))) ( -61.70) >DroPer_CAF1 27701 111 - 1 AGGACCCAUCAAGUGCAUGG---GUGGACCCCCCAUCAUGUGGAUUGGUGCACCGCGACCGUUGUGGCGCAUGAAGUGAGGCGG------UGGCGGUGGACCACCCAUCGGUGGCAUGGG ...(((((((....).))))---))...(((.((((((((.((..((((.((((((.((((((....(((.....))).)))))------).)))))).))))))))).)))))...))) ( -53.00) >consensus UGGACCCAUCAUGUGCAUGGGCGGUGGACCACCCAUCAUGGGACCUGG___ACCGGGUCCGUUGCGACGCAUAAAGUGAGGUGG______CGGCGGUGGUCCACCCAUGGGGCCCAUGGG .(((((.(((....(((.....(((((.....)))))...((((((((....))))))))..)))..(((..........)))...........)))))))).((((((.....)))))) (-31.30 = -33.82 + 2.52)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 09:53:13 2006