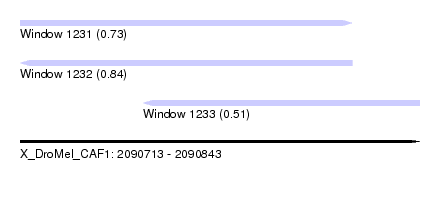
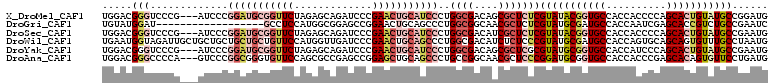
| Sequence ID | X_DroMel_CAF1 |
|---|---|
| Location | 2,090,713 – 2,090,843 |
| Length | 130 |
| Max. P | 0.840831 |

| Location | 2,090,713 – 2,090,821 |
|---|---|
| Length | 108 |
| Sequences | 6 |
| Columns | 111 |
| Reading direction | forward |
| Mean pairwise identity | 74.31 |
| Mean single sequence MFE | -45.64 |
| Consensus MFE | -27.21 |
| Energy contribution | -29.60 |
| Covariance contribution | 2.39 |
| Combinations/Pair | 1.19 |
| Mean z-score | -1.47 |
| Structure conservation index | 0.60 |
| SVM decision value | 0.41 |
| SVM RNA-class probability | 0.725628 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
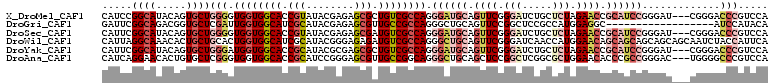
>X_DroMel_CAF1 2090713 108 + 22224390 CAUCCGGCAUACAGUGCUGGGGUGGUGGCACCGUAUACGAGAGCGCUGUCGCCAGGGAUGCAGUUCGGGAUCUGCUCUAGAACCGCAUCCGGGAU---CGGGACCCGUCCA ..((((((((...)))))))).((((((((.(((........))).)))))))).((((((.((((.(((.....))).)))).)))))).(((.---(((...)))))). ( -47.20) >DroGri_CAF1 11176 93 + 1 GAUUCGGCAGACGGUGCUCGAUUGGUGGCAUCGCAUACGAGAGCGUUGCCGCCAGGGCUGCAGUUCCGGCUCCGCCAUGGAGGC------------------AUCCAUACA .....(((.((((((((....(((((((((.(((........))).)))))))))....)))...)))..)).)))(((((...------------------.)))))... ( -38.00) >DroSec_CAF1 14253 108 + 1 CAUUCGGCAUACAGUGCUGGGGUGGUGGCACCGUAUACGAGAGCGAUGUCGCCAGGGAUGCAGUUCGGGAUCUGCUCUAGAACCGCAUCCGGGAU---CGGGACCCGUCCA ..((((((((...)))))))).((((((((.(((........))).)))))))).((((((.((((.(((.....))).)))).)))))).(((.---(((...)))))). ( -44.40) >DroWil_CAF1 10579 111 + 1 CAUUAGGCAAACACUGCUGCACUGGUGGCAUCGCAUACGGGAGAGAUGUCGCCAGGGCUGCAGUUCGGGAUCAACCAUGGAACAGCAGCAGCAGCAGCAAUCUACCAUUCA .....((......(((((((.(((((((((((.(........).))))))))))).(((((.((((.((.....))...)))).))))).))))))).......))..... ( -47.52) >DroYak_CAF1 16852 108 + 1 CAUUCGGCAUACAGUGCUGGGAUGGUGGCACCGCAUACGCGAGCGCUGUCGCCAGGGAUGCAGUUCGGGAUCUGCUCUAGAACCGCAUCCGGGAU---CGGGACCCGUCCA ....((((((...))))))(((((((((((.(((........))).)))))))..((((((.((((.(((.....))).)))).))))))(((..---.....))))))). ( -46.50) >DroAna_CAF1 14638 108 + 1 CAUCAGGAACACUGUGCUCGGGUGGUGGCACCGCAUCCGGGAGCGUUGCCGGCAGGGCUGCAGCUCCGGCUCGGCGCUGGAACACCCGCCGGGAC---UGGGGCCCGUCCA .....(((((.((...((((((((.((....)))))))))))).))).))((..(((((.(((..(((((..((.(......).)).)))))..)---)).)))))..)). ( -50.20) >consensus CAUUCGGCAUACAGUGCUGGGGUGGUGGCACCGCAUACGAGAGCGAUGUCGCCAGGGAUGCAGUUCGGGAUCUGCUCUAGAACCGCAUCCGGGAU___CGGGACCCGUCCA .....((((.....))))(((.((((((((.(((........))).)))))))).((((((.((((.(((.....))).)))).)))))).............)))..... (-27.21 = -29.60 + 2.39)
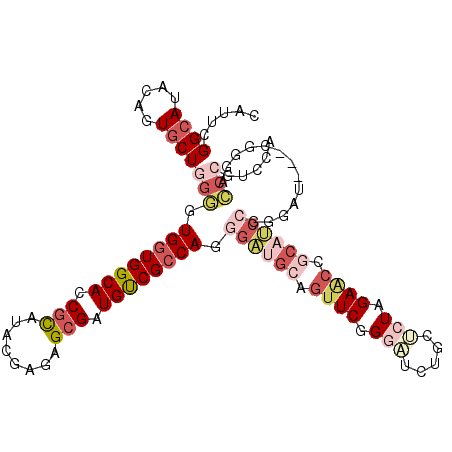
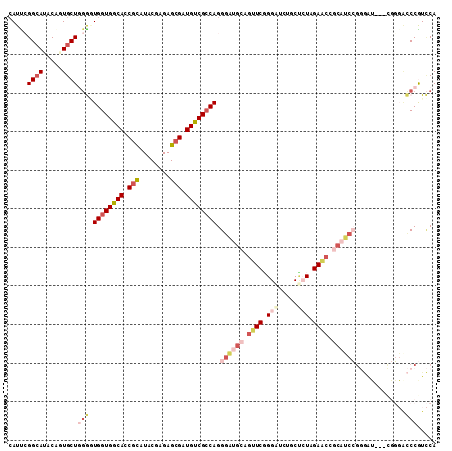
| Location | 2,090,713 – 2,090,821 |
|---|---|
| Length | 108 |
| Sequences | 6 |
| Columns | 111 |
| Reading direction | reverse |
| Mean pairwise identity | 74.31 |
| Mean single sequence MFE | -44.12 |
| Consensus MFE | -24.62 |
| Energy contribution | -27.15 |
| Covariance contribution | 2.53 |
| Combinations/Pair | 1.24 |
| Mean z-score | -1.74 |
| Structure conservation index | 0.56 |
| SVM decision value | 0.75 |
| SVM RNA-class probability | 0.840831 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 2090713 108 - 22224390 UGGACGGGUCCCG---AUCCCGGAUGCGGUUCUAGAGCAGAUCCCGAACUGCAUCCCUGGCGACAGCGCUCUCGUAUACGGUGCCACCACCCCAGCACUGUAUGCCGGAUG .((((((...)))---.))).(((((((((((..((.....))..)))))))))))(((....)))...(((.(((((((((((..........))))))))))).))).. ( -44.60) >DroGri_CAF1 11176 93 - 1 UGUAUGGAU------------------GCCUCCAUGGCGGAGCCGGAACUGCAGCCCUGGCGGCAACGCUCUCGUAUGCGAUGCCACCAAUCGAGCACCGUCUGCCGAAUC ...(((((.------------------...)))))((((((..(((...(((.....(((.((((.(((........))).)))).))).....))))))))))))..... ( -35.00) >DroSec_CAF1 14253 108 - 1 UGGACGGGUCCCG---AUCCCGGAUGCGGUUCUAGAGCAGAUCCCGAACUGCAUCCCUGGCGACAUCGCUCUCGUAUACGGUGCCACCACCCCAGCACUGUAUGCCGAAUG .((((((...)))---.))).(((((((((((..((.....))..)))))))))))..((((....)))).(((((((((((((..........)))))))))).)))... ( -43.40) >DroWil_CAF1 10579 111 - 1 UGAAUGGUAGAUUGCUGCUGCUGCUGCUGUUCCAUGGUUGAUCCCGAACUGCAGCCCUGGCGACAUCUCUCCCGUAUGCGAUGCCACCAGUGCAGCAGUGUUUGCCUAAUG .....(((((((.((((((((.(((((.((((...((.....)).)))).))))).((((.(.((((.(........).)))).).)))).)))))))))))))))..... ( -45.30) >DroYak_CAF1 16852 108 - 1 UGGACGGGUCCCG---AUCCCGGAUGCGGUUCUAGAGCAGAUCCCGAACUGCAUCCCUGGCGACAGCGCUCGCGUAUGCGGUGCCACCAUCCCAGCACUGUAUGCCGAAUG .((((((...)))---.))).(((((((((((..((.....))..)))))))))))(((....)))...(((.(((((((((((..........))))))))))))))... ( -45.70) >DroAna_CAF1 14638 108 - 1 UGGACGGGCCCCA---GUCCCGGCGGGUGUUCCAGCGCCGAGCCGGAGCUGCAGCCCUGCCGGCAACGCUCCCGGAUGCGGUGCCACCACCCGAGCACAGUGUUCCUGAUG .((..((((..((---(..(((((.(((((....)))))..)))))..)))..))))..))((((.(((........))).)))).......((((.....))))...... ( -50.70) >consensus UGGACGGGUCCCG___AUCCCGGAUGCGGUUCCAGAGCAGAUCCCGAACUGCAGCCCUGGCGACAACGCUCUCGUAUGCGGUGCCACCACCCCAGCACUGUAUGCCGAAUG .....(((.............(((((((((((.............)))))))))))..((((....)))))))(((((((((((..........)))))))))))...... (-24.62 = -27.15 + 2.53)
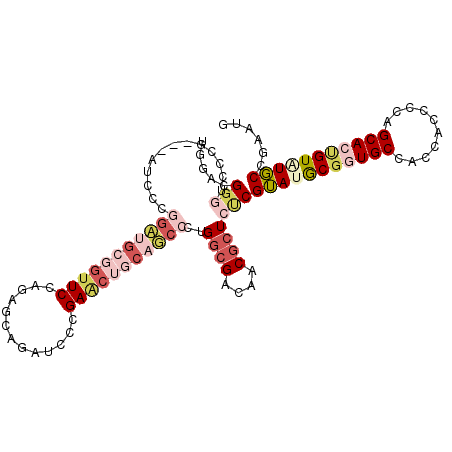
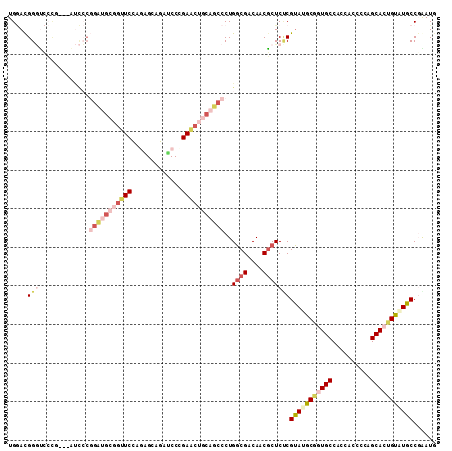
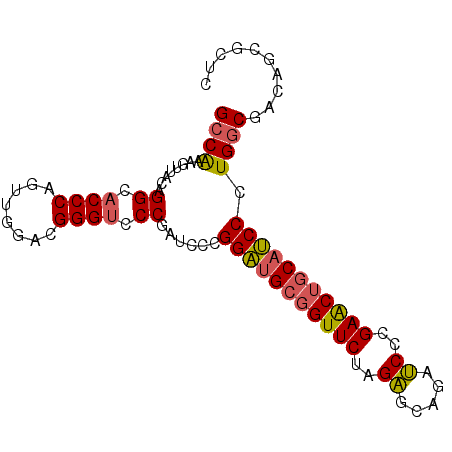
| Location | 2,090,753 – 2,090,843 |
|---|---|
| Length | 90 |
| Sequences | 6 |
| Columns | 90 |
| Reading direction | reverse |
| Mean pairwise identity | 92.00 |
| Mean single sequence MFE | -36.30 |
| Consensus MFE | -31.30 |
| Energy contribution | -31.30 |
| Covariance contribution | 0.00 |
| Combinations/Pair | 1.17 |
| Mean z-score | -1.39 |
| Structure conservation index | 0.86 |
| SVM decision value | -0.05 |
| SVM RNA-class probability | 0.510242 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 2090753 90 - 22224390 GCCAAAAUUGCAGGCACCCAGUUGGACGGGUCCCGAUCCCGGAUGCGGUUCUAGAGCAGAUCCCGAACUGCAUCCCUGGCGACAGCGCUC ((((........((.((((........)))).))......(((((((((((..((.....))..))))))))))).)))).......... ( -34.20) >DroSec_CAF1 14293 90 - 1 GCCAAAGUUGCAGGCACCCAGUUGGACGGGUCCCGAUCCCGGAUGCGGUUCUAGAGCAGAUCCCGAACUGCAUCCCUGGCGACAUCGCUC ......(((((.((.((((........)))).))......(((((((((((..((.....))..)))))))))))...)))))....... ( -35.20) >DroSim_CAF1 14943 90 - 1 GCCAAAGUUGCAGGCACCCAGUUGGACGGGUCCCGAUCCCGGAUGCGGUUCUAGAGCAGAUCCCGAACUGCAUCCCUGGCGACAGCGCUC ......(((((.((.((((........)))).))......(((((((((((..((.....))..)))))))))))...)))))....... ( -35.20) >DroEre_CAF1 14301 90 - 1 GCCAAAGUUACAGGCACCCAGUUGGACGGGUCCCGAUCCCGGAUGCGGUUCUAGAGCAGAUCCCGAACUGCAUCCCUGGCGACAGCGCUC ((((........((.((((........)))).))......(((((((((((..((.....))..))))))))))).)))).......... ( -34.20) >DroYak_CAF1 16892 90 - 1 GCCAAAGUUACAGGCACCCAGUUGGACGGGUCCCGAUCCCGGAUGCGGUUCUAGAGCAGAUCCCGAACUGCAUCCCUGGCGACAGCGCUC ((((........((.((((........)))).))......(((((((((((..((.....))..))))))))))).)))).......... ( -34.20) >DroAna_CAF1 14678 90 - 1 GCCGAAGUUACAGGCACCCAGUUGGACGGGCCCCAGUCCCGGCGGGUGUUCCAGCGCCGAGCCGGAGCUGCAGCCCUGCCGGCAACGCUC (((.........))).....(((((..((((..(((..(((((.(((((....)))))..)))))..)))..))))..)))))....... ( -44.80) >consensus GCCAAAGUUACAGGCACCCAGUUGGACGGGUCCCGAUCCCGGAUGCGGUUCUAGAGCAGAUCCCGAACUGCAUCCCUGGCGACAGCGCUC ((((........((.((((........)))).))......(((((((((((..((.....))..))))))))))).)))).......... (-31.30 = -31.30 + 0.00)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 09:53:09 2006