
| Sequence ID | X_DroMel_CAF1 |
|---|---|
| Location | 18,612,086 – 18,612,256 |
| Length | 170 |
| Max. P | 0.998189 |

| Location | 18,612,086 – 18,612,198 |
|---|---|
| Length | 112 |
| Sequences | 5 |
| Columns | 114 |
| Reading direction | forward |
| Mean pairwise identity | 85.85 |
| Mean single sequence MFE | -40.38 |
| Consensus MFE | -30.33 |
| Energy contribution | -32.22 |
| Covariance contribution | 1.89 |
| Combinations/Pair | 1.16 |
| Mean z-score | -4.01 |
| Structure conservation index | 0.75 |
| SVM decision value | 3.03 |
| SVM RNA-class probability | 0.998189 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
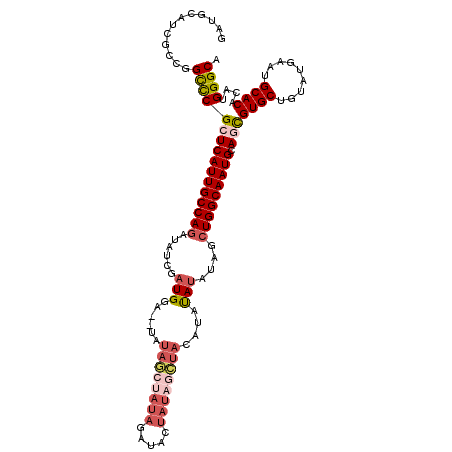
>X_DroMel_CAF1 18612086 112 + 22224390 GAUGCAUCGCCGGCCCGCUCAUUGCCAGAUAUCGAUGGA--UAUAGCUAUAGAUACUAUAGCUACAUAUAUCUAGCUGGCAAUGCAGCGUGCUGUAUGAAUGCACACAUGGGCA ............((((((((((((((((.....((((.(--(.(((((((((...))))))))).)).))))...))))))))).)))((((.........))))....)))). ( -46.30) >DroSec_CAF1 73199 112 + 1 GAUGCAUCGCCGGCCCGCUCAUUGCCAGAUAUCGAUGGA--UAUAGCUAUAGAUACUAUAGCUACAUACACAUAGCUGGCAAUGCAGCGUGCUGUAUGAAUGCACACAUGGGCA ............((((((((((((((((......(((.(--(.(((((((((...))))))))).))...)))..))))))))).)))((((.........))))....)))). ( -44.90) >DroSim_CAF1 71547 112 + 1 GAUGCAUCGCCGGCCCGCUCAUUGCCAGAUAUCGAUGGA--UAUAGCUAUAGAUACUAUAGCUACAUACAUAUAGCUGGCAAUGCAGCGUGCUGUAUGAAUGCACACAUGGGCA ............((((((((((((((((......(((.(--(.(((((((((...))))))))).)).)))....))))))))).)))((((.........))))....)))). ( -45.80) >DroEre_CAF1 59562 98 + 1 GAUGCAUCGCCGGCUCGCUCAUUGCCAGAUAUCGAUGGA------------UAU---ACACCUAUGUAUAUAAA-GUGGCAAUGCAACGUGCUGUAUGAAUGCACAUAUGGGCA ........((((((.((..(((((((((....).....(------------(((---(((....)))))))...-.))))))))...)).)))(((((......))))).))). ( -27.70) >DroYak_CAF1 64199 110 + 1 GAUGCAUCGCCGGUCCGCUCAUUGCCAGAUAUCGAUGGAGCUAUAUCCAUCUAU---AUACAUAUAGAUAUAUA-CUGGCAAUGCAAUGUGCUGUAUGAAUGCACACAUGGGCA ............((((...((((((((((((((((((((......))))))(((---(....)))))))))...-)))))))))..(((((.((((....))))))))))))). ( -37.20) >consensus GAUGCAUCGCCGGCCCGCUCAUUGCCAGAUAUCGAUGGA__UAUAGCUAUAGAUACUAUAGCUACAUAUAUAUAGCUGGCAAUGCAGCGUGCUGUAUGAAUGCACACAUGGGCA ............((((((((((((((((......(((......((((((((.....))))))))....)))....))))))))).)))((((.........))))....)))). (-30.33 = -32.22 + 1.89)


| Location | 18,612,086 – 18,612,198 |
|---|---|
| Length | 112 |
| Sequences | 5 |
| Columns | 114 |
| Reading direction | reverse |
| Mean pairwise identity | 85.85 |
| Mean single sequence MFE | -42.30 |
| Consensus MFE | -30.75 |
| Energy contribution | -33.48 |
| Covariance contribution | 2.73 |
| Combinations/Pair | 1.11 |
| Mean z-score | -4.61 |
| Structure conservation index | 0.73 |
| SVM decision value | 2.94 |
| SVM RNA-class probability | 0.997826 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 18612086 112 - 22224390 UGCCCAUGUGUGCAUUCAUACAGCACGCUGCAUUGCCAGCUAGAUAUAUGUAGCUAUAGUAUCUAUAGCUAUA--UCCAUCGAUAUCUGGCAAUGAGCGGGCCGGCGAUGCAUC .(((..((((((....))))))((.((((.(((((((((...(((..((((((((((((...)))))))))))--)..))).....))))))))))))).)).)))........ ( -48.10) >DroSec_CAF1 73199 112 - 1 UGCCCAUGUGUGCAUUCAUACAGCACGCUGCAUUGCCAGCUAUGUGUAUGUAGCUAUAGUAUCUAUAGCUAUA--UCCAUCGAUAUCUGGCAAUGAGCGGGCCGGCGAUGCAUC .(((..((((((....))))))((.((((.(((((((((.((((((.((((((((((((...)))))))))))--).)))..))).))))))))))))).)).)))........ ( -49.20) >DroSim_CAF1 71547 112 - 1 UGCCCAUGUGUGCAUUCAUACAGCACGCUGCAUUGCCAGCUAUAUGUAUGUAGCUAUAGUAUCUAUAGCUAUA--UCCAUCGAUAUCUGGCAAUGAGCGGGCCGGCGAUGCAUC .(((..((((((....))))))((.((((.(((((((((.((((((.((((((((((((...)))))))))))--).)))..))).))))))))))))).)).)))........ ( -48.90) >DroEre_CAF1 59562 98 - 1 UGCCCAUAUGUGCAUUCAUACAGCACGUUGCAUUGCCAC-UUUAUAUACAUAGGUGU---AUA------------UCCAUCGAUAUCUGGCAAUGAGCGAGCCGGCGAUGCAUC .......((((((.........))))))(((((((((..-..(((((((....))))---)))------------.............(((.........)))))))))))).. ( -29.10) >DroYak_CAF1 64199 110 - 1 UGCCCAUGUGUGCAUUCAUACAGCACAUUGCAUUGCCAG-UAUAUAUCUAUAUGUAU---AUAGAUGGAUAUAGCUCCAUCGAUAUCUGGCAAUGAGCGGACCGGCGAUGCAUC .(((...((((((.........))))))..(((((((((-(((((((......))))---)))((((((......)))))).....)))))))))........)))........ ( -36.20) >consensus UGCCCAUGUGUGCAUUCAUACAGCACGCUGCAUUGCCAGCUAUAUAUAUGUAGCUAUAGUAUCUAUAGCUAUA__UCCAUCGAUAUCUGGCAAUGAGCGGGCCGGCGAUGCAUC .(((..((((((....))))))((.((((.(((((((((.(((.....((((((((((.....)))))))))).........))).))))))))))))).)).)))........ (-30.75 = -33.48 + 2.73)



| Location | 18,612,125 – 18,612,234 |
|---|---|
| Length | 109 |
| Sequences | 4 |
| Columns | 114 |
| Reading direction | forward |
| Mean pairwise identity | 85.91 |
| Mean single sequence MFE | -28.00 |
| Consensus MFE | -18.98 |
| Energy contribution | -20.10 |
| Covariance contribution | 1.12 |
| Combinations/Pair | 1.04 |
| Mean z-score | -2.34 |
| Structure conservation index | 0.68 |
| SVM decision value | 0.86 |
| SVM RNA-class probability | 0.869752 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 18612125 109 + 22224390 -UAUAGCUAUAGAUACUAUAGCUACAUAUAUCUAGCUGGCAAUGCAGCGUGCUGUAUGAAUGCACACAUGGGCA----UAUAUGGCUCAUUACGUACGAAUCCACAUAUAUAGA -..(((((((((...))))))))).((((((...((((......))))(((.((((((((((..(.((((....----..)))))..)))).))))))....)))))))))... ( -27.40) >DroSec_CAF1 73238 107 + 1 -UAUAGCUAUAGAUACUAUAGCUACAUACACAUAGCUGGCAAUGCAGCGUGCUGUAUGAAUGCACACAUGGGCA----UAUAUGGCUCAUUACGUACGAAUCCACAUAGAUA-- -..(((((((((...)))))))))(((((((((.((((......))))))).))))))..(((....((((((.----......))))))...)))................-- ( -28.90) >DroSim_CAF1 71586 109 + 1 -UAUAGCUAUAGAUACUAUAGCUACAUACAUAUAGCUGGCAAUGCAGCGUGCUGUAUGAAUGCACACAUGGGCA----UAUAUGGCUCACUACGUACGAAUCCGCAUAGAUAGA -..(((((((((...)))))))))....((((((((..((......))..)))))))).((((.....(((((.----......)))))...(....).....))))....... ( -29.00) >DroYak_CAF1 64239 108 + 1 CUAUAUCCAUCUAU---AUACAUAUAGAUAUAUA-CUGGCAAUGCAAUGUGCUGUAUGAAUGCACACAUGGGCAUACAUAUAUGGCUCAUUACGUACGAGUCCGCAUUGAAA-- .(((((..((((((---(....))))))))))))-....((((((.(((((.((((((.(((....)))...)))))))))))(((((.........))))).))))))...-- ( -26.70) >consensus _UAUAGCUAUAGAUACUAUAGCUACAUACAUAUAGCUGGCAAUGCAGCGUGCUGUAUGAAUGCACACAUGGGCA____UAUAUGGCUCAUUACGUACGAAUCCACAUAGAUA__ ...(((((((((...)))))))))..........((((......))))(.((((((((..(((........)))....)))))))).).......................... (-18.98 = -20.10 + 1.12)


| Location | 18,612,125 – 18,612,234 |
|---|---|
| Length | 109 |
| Sequences | 4 |
| Columns | 114 |
| Reading direction | reverse |
| Mean pairwise identity | 85.91 |
| Mean single sequence MFE | -31.78 |
| Consensus MFE | -25.33 |
| Energy contribution | -25.08 |
| Covariance contribution | -0.25 |
| Combinations/Pair | 1.23 |
| Mean z-score | -3.06 |
| Structure conservation index | 0.80 |
| SVM decision value | 2.30 |
| SVM RNA-class probability | 0.992059 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 18612125 109 - 22224390 UCUAUAUAUGUGGAUUCGUACGUAAUGAGCCAUAUA----UGCCCAUGUGUGCAUUCAUACAGCACGCUGCAUUGCCAGCUAGAUAUAUGUAGCUAUAGUAUCUAUAGCUAUA- (((((((((((((.(((((.....))))))))))))----)).......((((.........))))((((......))))))))....(((((((((((...)))))))))))- ( -33.20) >DroSec_CAF1 73238 107 - 1 --UAUCUAUGUGGAUUCGUACGUAAUGAGCCAUAUA----UGCCCAUGUGUGCAUUCAUACAGCACGCUGCAUUGCCAGCUAUGUGUAUGUAGCUAUAGUAUCUAUAGCUAUA- --....(((((((.(((((.....))))))))))))----(((.((((.((((.........))))((((......)))))))).)))(((((((((((...)))))))))))- ( -33.10) >DroSim_CAF1 71586 109 - 1 UCUAUCUAUGCGGAUUCGUACGUAGUGAGCCAUAUA----UGCCCAUGUGUGCAUUCAUACAGCACGCUGCAUUGCCAGCUAUAUGUAUGUAGCUAUAGUAUCUAUAGCUAUA- ...((((....)))).((((((((...(((.....(----(((....((((((.........)))))).)))).....))).))))))))(((((((((...)))))))))..- ( -32.30) >DroYak_CAF1 64239 108 - 1 --UUUCAAUGCGGACUCGUACGUAAUGAGCCAUAUAUGUAUGCCCAUGUGUGCAUUCAUACAGCACAUUGCAUUGCCAG-UAUAUAUCUAUAUGUAU---AUAGAUGGAUAUAG --...((((((((...(((((((((((...))).)))))))).))..((((((.........)))))).))))))...(-(((.((((((((....)---))))))).)))).. ( -28.50) >consensus __UAUCUAUGCGGAUUCGUACGUAAUGAGCCAUAUA____UGCCCAUGUGUGCAUUCAUACAGCACGCUGCAUUGCCAGCUAUAUAUAUGUAGCUAUAGUAUCUAUAGCUAUA_ .......((((((.(((((.....))))))))))).....(((....((((((.........)))))).)))................(((((((((((...))))))))))). (-25.33 = -25.08 + -0.25)



| Location | 18,612,164 – 18,612,256 |
|---|---|
| Length | 92 |
| Sequences | 4 |
| Columns | 98 |
| Reading direction | reverse |
| Mean pairwise identity | 82.94 |
| Mean single sequence MFE | -22.55 |
| Consensus MFE | -16.70 |
| Energy contribution | -16.45 |
| Covariance contribution | -0.25 |
| Combinations/Pair | 1.18 |
| Mean z-score | -2.28 |
| Structure conservation index | 0.74 |
| SVM decision value | 1.41 |
| SVM RNA-class probability | 0.951159 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 18612164 92 - 22224390 AUGUAC--AUAUGUAUUAUAUCUAUCUAUAUAUGUGGAUUCGUACGUAAUGAGCCAUAUA----UGCCCAUGUGUGCAUUCAUACAGCACGCUGCAUU ((((((--(((((...((((......))))(((((((.(((((.....))))))))))))----....))))))))))).....(((....))).... ( -23.30) >DroSec_CAF1 73277 83 - 1 ----AC--GUAUGUAU-CUAUC----UAUCUAUGUGGAUUCGUACGUAAUGAGCCAUAUA----UGCCCAUGUGUGCAUUCAUACAGCACGCUGCAUU ----((--(((((.((-((((.----.......)))))).)))))))............(----(((....((((((.........)))))).)))). ( -22.90) >DroSim_CAF1 71625 87 - 1 ----AC--GUAUGUAU-CUAUCUAUCUAUCUAUGCGGAUUCGUACGUAGUGAGCCAUAUA----UGCCCAUGUGUGCAUUCAUACAGCACGCUGCAUU ----..--(((((.((-(..(.((......)).)..))).)))))((((((.((..((((----(((.(....).))))..)))..)).))))))... ( -19.80) >DroYak_CAF1 64275 89 - 1 ----AUAGAUAUGUAC-ACAUG----UUUCAAUGCGGACUCGUACGUAAUGAGCCAUAUAUGUAUGCCCAUGUGUGCAUUCAUACAGCACAUUGCAUU ----...((.((((((-(((((----.((((.((((........)))).)))).((((....))))..))))))))))))).....((.....))... ( -24.20) >consensus ____AC__AUAUGUAU_AUAUC____UAUCUAUGCGGAUUCGUACGUAAUGAGCCAUAUA____UGCCCAUGUGUGCAUUCAUACAGCACGCUGCAUU ..........(((((................((((((.(((((.....)))))))))))............((((((.........))))))))))). (-16.70 = -16.45 + -0.25)



Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 12:39:22 2006