
| Sequence ID | X_DroMel_CAF1 |
|---|---|
| Location | 18,545,350 – 18,545,467 |
| Length | 117 |
| Max. P | 0.941904 |

| Location | 18,545,350 – 18,545,467 |
|---|---|
| Length | 117 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 82.43 |
| Mean single sequence MFE | -52.98 |
| Consensus MFE | -37.27 |
| Energy contribution | -37.50 |
| Covariance contribution | 0.23 |
| Combinations/Pair | 1.23 |
| Mean z-score | -2.68 |
| Structure conservation index | 0.70 |
| SVM decision value | 0.84 |
| SVM RNA-class probability | 0.862795 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
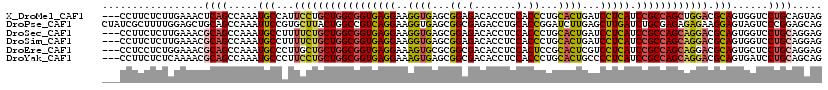
>X_DroMel_CAF1 18545350 117 + 22224390 CUACUGCAGGACCACUGCGUCCAGCUGGCGGAUGAGGAUCAGUGCAGGGUGGAGGUGUCUCCGCUCACCUUCCUCACCGCCAGCAGGAAUGGCAUUUGGCUGAGUUUCAAGAGAAGG--- ..(((.(((..(((.(((.(((.((((((((.((((((...(((...(((((((....))))))))))..)))))))))))))).)))...)))..)))))))))............--- ( -52.00) >DroPse_CAF1 16702 120 + 1 CUGCUCCGGGACUACUCCUUCUCUCUCGCAGAUCAAGCUCAAGAUCCGGUGCAGGUCUCGCCGCUCACCUUCCUGACGGCCAGUAAGCACGGAAUUUGGCUGCAGCUCCAAAAGCGAUAG ((((...((((........))))....))))(((.(((.((((.((((.(((....((.((((.(((......))))))).))...))))))).)))))))...(((.....)))))).. ( -35.30) >DroSec_CAF1 10166 117 + 1 CUCCUGCAGGACCACUGCGUCCUGCUGGCGGAUGAGGAUCAGUGCAGGGUGGAGGUGUCUCCGCUCACCUUCCUCACCGCCAGCAGAAAAGGCAUUUGGCUGCGUUUCAAGAGAAGG--- (((..((((.....))))...((((((((((.((((((...(((...(((((((....))))))))))..)))))))))))))))).(((.(((......))).)))...)))....--- ( -54.80) >DroSim_CAF1 10226 117 + 1 CUCCUGCAGGACCACUGCGUCCUGCUGGCGGAUGAGGAUCAGUGCAGGGUGGAGGUGUCUCCGCUCACCUUCCUCACCGCCAGCAGAAAAGGCAUUUGGCUGCGUUUCAAGAGAAGG--- (((..((((.....))))...((((((((((.((((((...(((...(((((((....))))))))))..)))))))))))))))).(((.(((......))).)))...)))....--- ( -54.80) >DroEre_CAF1 2628 117 + 1 CUCCUGCAGGAGCACUGCGUCCUGCUGGCGGAUGAGGACGAGUGCGGAGUGGAGGUGUCGCCGCGCACUUUCCUCACCGCCAGCAGCAAGGGCAUUUGGCUGCGUUUCCAGAGGAGG--- ((((.((((.....))))(((((((((((((.((((((.(((((((..((((.....))))..)))))))))))))))))))))....))))).(((((........))))))))).--- ( -61.90) >DroYak_CAF1 2695 117 + 1 CUGCUGCAGGAUCACUGCGUCCUGCUGGCGGAUGAGGGGCAGUGCAGGGUGGAGGUGUCGCCGCUCACUUUCCUCACCGCCAGCAGGAAGGGCAUUUGGCUGCGUUUUGAGAGAAGG--- .((((((((.....)))).((((((((((((.(((((((.((((.(((((((.....))))).)))))))))))))))))))))))))..))))..........((((....)))).--- ( -59.10) >consensus CUCCUGCAGGACCACUGCGUCCUGCUGGCGGAUGAGGAUCAGUGCAGGGUGGAGGUGUCGCCGCUCACCUUCCUCACCGCCAGCAGGAAAGGCAUUUGGCUGCGUUUCAAGAGAAGG___ .....((((.....))))(((((((((((((.((((((........(((((..((.....))...)))))))))))))))))))))....)))........................... (-37.27 = -37.50 + 0.23)


| Location | 18,545,350 – 18,545,467 |
|---|---|
| Length | 117 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 82.43 |
| Mean single sequence MFE | -52.73 |
| Consensus MFE | -38.56 |
| Energy contribution | -38.35 |
| Covariance contribution | -0.21 |
| Combinations/Pair | 1.32 |
| Mean z-score | -3.60 |
| Structure conservation index | 0.73 |
| SVM decision value | 1.32 |
| SVM RNA-class probability | 0.941904 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 18545350 117 - 22224390 ---CCUUCUCUUGAAACUCAGCCAAAUGCCAUUCCUGCUGGCGGUGAGGAAGGUGAGCGGAGACACCUCCACCCUGCACUGAUCCUCAUCCGCCAGCUGGACGCAGUGGUCCUGCAGUAG ---..(((....)))(((((((((..(((...(((.((((((((((((((.((((.(.((((....)))).)....))))..)))))).)))))))).))).))).)))..))).))).. ( -51.00) >DroPse_CAF1 16702 120 - 1 CUAUCGCUUUUGGAGCUGCAGCCAAAUUCCGUGCUUACUGGCCGUCAGGAAGGUGAGCGGCGAGACCUGCACCGGAUCUUGAGCUUGAUCUGCGAGAGAGAAGGAGUAGUCCCGGAGCAG .....((((..(((.((((((((((..((((((((((((..((....))..))))))))(((.....)))..))))..))).)))...(((.(....))))....)))))))..)))).. ( -44.20) >DroSec_CAF1 10166 117 - 1 ---CCUUCUCUUGAAACGCAGCCAAAUGCCUUUUCUGCUGGCGGUGAGGAAGGUGAGCGGAGACACCUCCACCCUGCACUGAUCCUCAUCCGCCAGCAGGACGCAGUGGUCCUGCAGGAG ---....((((((.......((((..(((...((((((((((((((((((.((((.(.((((....)))).)....))))..)))))).)))))))))))).))).))))....)))))) ( -56.00) >DroSim_CAF1 10226 117 - 1 ---CCUUCUCUUGAAACGCAGCCAAAUGCCUUUUCUGCUGGCGGUGAGGAAGGUGAGCGGAGACACCUCCACCCUGCACUGAUCCUCAUCCGCCAGCAGGACGCAGUGGUCCUGCAGGAG ---....((((((.......((((..(((...((((((((((((((((((.((((.(.((((....)))).)....))))..)))))).)))))))))))).))).))))....)))))) ( -56.00) >DroEre_CAF1 2628 117 - 1 ---CCUCCUCUGGAAACGCAGCCAAAUGCCCUUGCUGCUGGCGGUGAGGAAAGUGCGCGGCGACACCUCCACUCCGCACUCGUCCUCAUCCGCCAGCAGGACGCAGUGCUCCUGCAGGAG ---.(((((..(....)((((.....(((...(.((((((((((((((((.((((((.((.(.......).)).))))))..)))))).)))))))))).).)))......))))))))) ( -58.00) >DroYak_CAF1 2695 117 - 1 ---CCUUCUCUCAAAACGCAGCCAAAUGCCCUUCCUGCUGGCGGUGAGGAAAGUGAGCGGCGACACCUCCACCCUGCACUGCCCCUCAUCCGCCAGCAGGACGCAGUGAUCCUGCAGCAG ---..............((((.....(((...(((((((((((((((((..((((((.((.(.......).)))).))))...))))).)))))))))))).)))......))))..... ( -51.20) >consensus ___CCUUCUCUUGAAACGCAGCCAAAUGCCCUUCCUGCUGGCGGUGAGGAAGGUGAGCGGAGACACCUCCACCCUGCACUGAUCCUCAUCCGCCAGCAGGACGCAGUGGUCCUGCAGGAG .................((((.....(((...(((((((((((((((((..((((...((.(.......).))...))))...))))).)))))))))))).)))......))))..... (-38.56 = -38.35 + -0.21)
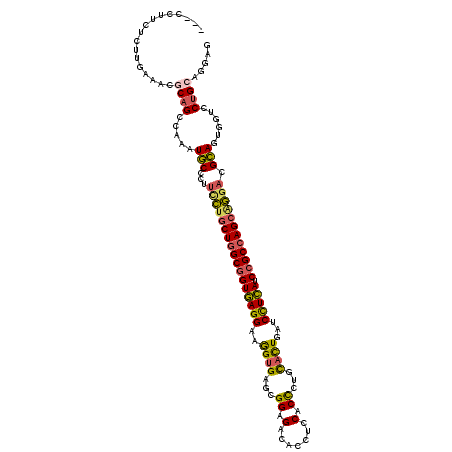

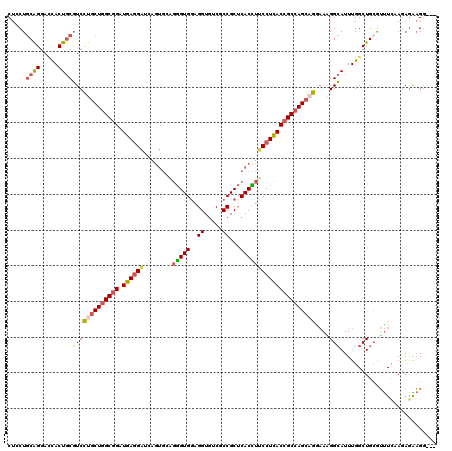
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 12:38:50 2006