
| Sequence ID | X_DroMel_CAF1 |
|---|---|
| Location | 18,321,696 – 18,321,813 |
| Length | 117 |
| Max. P | 0.693739 |

| Location | 18,321,696 – 18,321,813 |
|---|---|
| Length | 117 |
| Sequences | 6 |
| Columns | 117 |
| Reading direction | forward |
| Mean pairwise identity | 81.14 |
| Mean single sequence MFE | -49.45 |
| Consensus MFE | -35.07 |
| Energy contribution | -36.93 |
| Covariance contribution | 1.87 |
| Combinations/Pair | 1.25 |
| Mean z-score | -1.20 |
| Structure conservation index | 0.71 |
| SVM decision value | -0.05 |
| SVM RNA-class probability | 0.509925 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
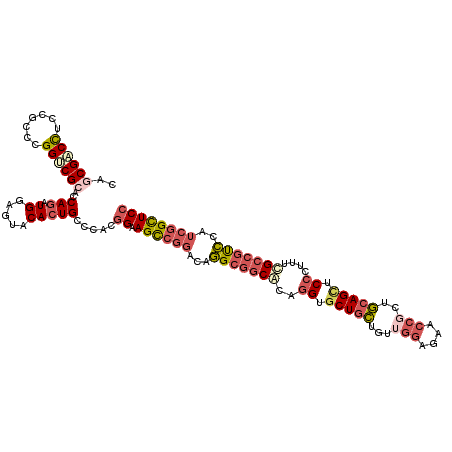
>X_DroMel_CAF1 18321696 117 + 22224390 CAGCGACCUCCGCCCGGUCGCACCAGAUGGAGUACACUGCCCACGGAAGCCGGACAGGCGGCGCAGGUGCUGCUGUUGGAGAACCGCUGCAGCUCCCUUUCGCCGUCCAUCGGCUCC ..((((((.......))))))......(((.((.....))))).(((.(((((...(((((((.(((.(((((.((.((....)))).)))))..)))..)))))))..)))))))) ( -51.30) >DroSec_CAF1 113 117 + 1 CAGCGCCCUCCGCCCGGUCGCACCAGAUGGAGUACACUGCCCACGGAAGCCGGACAGGCGGCACAGGUGCUGCUGUUGGAGAACCGCUGCAGCUCCCUUUCGCCGUCCAUCGGCUCC (((.(..(((((.(.((.....)).).)))))..).))).....(((.(((((...((((((..(((.(((((.((.((....)))).)))))..)))...))))))..)))))))) ( -47.50) >DroSim_CAF1 115 117 + 1 CAGCGACCUCCGCCCGGUCGCACCAGAUGGAGUACACUGCCCACGGUAGCCGGACAGGCGGCACAGGUGCUGCUGUUGGAGAACCGCUGCAGCUCCCUUUCGCCGUCCAUCGGUUCC ..((((((.......))))))(((.((((((((...(((((...)))))....)).(((((....((.(((((.((.((....)))).))))).))...))))).)))))))))... ( -49.50) >DroEre_CAF1 113 117 + 1 CAACGACCUCCACCCGGGCGCACCAGAUGGAGUACACUGCUCACUGAAGCCGGGCAGGCGGCGCAGGUGCUGCUGUUGGAGAACCUCUGCAGUUCCCUGGCGCCGCCCAUCGGCUCC ....(..(((((.(.((.....)).).)))))..)............((((((...(((((((((((.(((((....((....))...)))))..))).))))))))..)))))).. ( -50.80) >DroYak_CAF1 158 117 + 1 CAGCGACCACCACCUGGUCGCACCAGAUGGAGCACACUGCUCACGGAAGCCGGACAGGCGGCGCAGGUGCUGCUGUUGGAGAACCGCUGCAGCUCCCUGUCGCCGCCCAGCGGCUCC ..(((((((.....)))))))........((((.....))))..(((.((((....(((((((((((.(((((.((.((....)))).)))))..)))).)))))))...))))))) ( -61.90) >DroMoj_CAF1 22557 111 + 1 CAACGCCUGCCGCUGGGGCGAAGCAAAUGAAGCACACUGGCCACGGCAGUGGGACACGGAGCACACGAGCUAGAAUUGCUGGCAA---UCAGAGC---AUUGGCAUGAAUGGGCUCC ....(((..(.((((.(((.(.((.......))....).))).)))).)..)).)..(((((.((...(((((.....)))))..---(((..((---....)).))).)).))))) ( -35.70) >consensus CAGCGACCUCCGCCCGGUCGCACCAGAUGGAGUACACUGCCCACGGAAGCCGGACAGGCGGCACAGGUGCUGCUGUUGGAGAACCGCUGCAGCUCCCUUUCGCCGUCCAUCGGCUCC ..((((((.......))))))..(((.((.....))))).....((.((((((...(((((((..((.(((((...(((....)))..))))).))....)))))))..)))))))) (-35.07 = -36.93 + 1.87)


| Location | 18,321,696 – 18,321,813 |
|---|---|
| Length | 117 |
| Sequences | 6 |
| Columns | 117 |
| Reading direction | reverse |
| Mean pairwise identity | 81.14 |
| Mean single sequence MFE | -55.55 |
| Consensus MFE | -39.47 |
| Energy contribution | -40.67 |
| Covariance contribution | 1.20 |
| Combinations/Pair | 1.26 |
| Mean z-score | -1.55 |
| Structure conservation index | 0.71 |
| SVM decision value | 0.33 |
| SVM RNA-class probability | 0.693739 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 18321696 117 - 22224390 GGAGCCGAUGGACGGCGAAAGGGAGCUGCAGCGGUUCUCCAACAGCAGCACCUGCGCCGCCUGUCCGGCUUCCGUGGGCAGUGUACUCCAUCUGGUGCGACCGGGCGGAGGUCGCUG (((((((((((.(((((...(((.(((((...((....))....))))).))).))))).)))).)))))))......(((((..((((..((((.....))))..))))..))))) ( -58.50) >DroSec_CAF1 113 117 - 1 GGAGCCGAUGGACGGCGAAAGGGAGCUGCAGCGGUUCUCCAACAGCAGCACCUGUGCCGCCUGUCCGGCUUCCGUGGGCAGUGUACUCCAUCUGGUGCGACCGGGCGGAGGGCGCUG (((((((((((.(((((...(((.(((((...((....))....))))).))).))))).)))).)))))))......(((((..((((..((((.....))))..))))..))))) ( -59.00) >DroSim_CAF1 115 117 - 1 GGAACCGAUGGACGGCGAAAGGGAGCUGCAGCGGUUCUCCAACAGCAGCACCUGUGCCGCCUGUCCGGCUACCGUGGGCAGUGUACUCCAUCUGGUGCGACCGGGCGGAGGUCGCUG ...(((((((((.((((...(((.(((((...((....))....))))).))).))))(((((((((.(....)))))))).))..)))))).)))((((((.......)))))).. ( -53.20) >DroEre_CAF1 113 117 - 1 GGAGCCGAUGGGCGGCGCCAGGGAACUGCAGAGGUUCUCCAACAGCAGCACCUGCGCCGCCUGCCCGGCUUCAGUGAGCAGUGUACUCCAUCUGGUGCGCCCGGGUGGAGGUCGUUG (((((((.((((((((((.(((...((((...((....))....))))..)))))))))))))..)))))))......(((((..((((((((((.....))))))))))..))))) ( -60.90) >DroYak_CAF1 158 117 - 1 GGAGCCGCUGGGCGGCGACAGGGAGCUGCAGCGGUUCUCCAACAGCAGCACCUGCGCCGCCUGUCCGGCUUCCGUGAGCAGUGUGCUCCAUCUGGUGCGACCAGGUGGUGGUCGCUG (((((((.(((((((((.((((..(((((...((....))....))))).)))))))))))))..)))))))(....)(((((.(((((((((((.....)))))))).)))))))) ( -66.60) >DroMoj_CAF1 22557 111 - 1 GGAGCCCAUUCAUGCCAAU---GCUCUGA---UUGCCAGCAAUUCUAGCUCGUGUGCUCCGUGUCCCACUGCCGUGGCCAGUGUGCUUCAUUUGCUUCGCCCCAGCGGCAGGCGUUG (((((.....((((.....---(((..((---(((....)))))..))).)))).)))))........(((((((((...(((.((.......))..))).))).))))))...... ( -35.10) >consensus GGAGCCGAUGGACGGCGAAAGGGAGCUGCAGCGGUUCUCCAACAGCAGCACCUGCGCCGCCUGUCCGGCUUCCGUGGGCAGUGUACUCCAUCUGGUGCGACCGGGCGGAGGUCGCUG (((((((..((.(((((...(((.(((((...((....))....))))).))).)))))))....))))).)).....(((((..((((.(((((.....))))).))))..))))) (-39.47 = -40.67 + 1.20)


Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 12:36:27 2006