
| Sequence ID | X_DroMel_CAF1 |
|---|---|
| Location | 18,202,026 – 18,202,126 |
| Length | 100 |
| Max. P | 0.766625 |

| Location | 18,202,026 – 18,202,126 |
|---|---|
| Length | 100 |
| Sequences | 6 |
| Columns | 118 |
| Reading direction | reverse |
| Mean pairwise identity | 77.30 |
| Mean single sequence MFE | -34.79 |
| Consensus MFE | -20.98 |
| Energy contribution | -23.07 |
| Covariance contribution | 2.09 |
| Combinations/Pair | 1.27 |
| Mean z-score | -1.65 |
| Structure conservation index | 0.60 |
| SVM decision value | 0.52 |
| SVM RNA-class probability | 0.766625 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
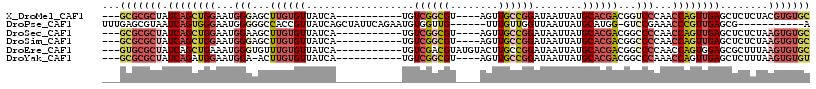
>X_DroMel_CAF1 18202026 100 - 22224390 ---GCGCGCUAUCAGCUGGAAUGGGAGCUUGUGUUAUCA-----------UGUCGGCGU----AGUUGCCGGAUAAUUAUGCACGACGGUCCCAACCAGUUGAGCUCUCUACGUGUGC ---((((((..((((((((..(((((.(((((((.....-----------..((((((.----...))))))........)))))).).))))).)))))))).........)))))) ( -42.26) >DroPse_CAF1 101473 100 - 1 UUUGAGCGUAAUCAGUGGGAAUGGGGGCCACCGUUAUCAGCUAUUCAGAAUGUGGUUG------UUUGUUGGUUAAUUAUGCAUGG-GUCCGAAACCCGUUGAGCG-----------A ......(((..((((((((..(.(((.((((((....(((((((.......)))))))------.....))))...........))-.))).)..)))))))))))-----------. ( -26.00) >DroSec_CAF1 62319 100 - 1 ---GCGCGCUAUCAGCUGGAAUGGAAGCUUGUGUUAUCA-----------UGUCGGCGU----AGUUGCCGGAUAAUUAUGCACGACGGCCCCAACCAGUUGAGCUCUCUAAGUGUGC ---(((((((.((((((((..(((..(((.(((....))-----------)((((((((----(((((.....))))))))).))))))).))).))))))))........))))))) ( -37.30) >DroSim_CAF1 61460 100 - 1 ---GCGCGCUAUCAGCUGGAAUGGGAGCUUGUGUUAUCA-----------UGUCGGCGU----AGUUGCCGGAUAAUUAUGCACGACGGCCCCAACCAGUUGAGCUCUCUAAGUGUGC ---(((((((.((((((((..((((.(((.(((....))-----------)((((((((----(((((.....))))))))).))))))))))).))))))))........))))))) ( -42.40) >DroEre_CAF1 56210 104 - 1 ---GUGCGCUAUCAGCUGAAAUGGGUGUUUGUGUUAUCA-----------UGUCGACGUAUGUACUUGCCGGAUAAUUAUGCACGACGGCCCCAACCAGUGGAGCGCUUUAAGUGUGC ---..(((((....((((....((((..((((((.....-----------((((...(((......)))..)))).....))))))..))))....))))..)))))........... ( -29.40) >DroYak_CAF1 56312 99 - 1 ---GCGCGCUAUCAGAUGGAAUGGA-ACUUGUGUUAUCA-----------UGUCGGCGU----AGUUGCCGGAUAAUUAUGCACGACGGCCCAAACCAGUUGAGCUCUUUAAGUGUGU ---(((((((.((((.(((..(((.-.(((((((.....-----------..((((((.----...))))))........)))))).)..)))..))).))))........))))))) ( -31.36) >consensus ___GCGCGCUAUCAGCUGGAAUGGGAGCUUGUGUUAUCA___________UGUCGGCGU____AGUUGCCGGAUAAUUAUGCACGACGGCCCCAACCAGUUGAGCUCUCUAAGUGUGC ...(((((((.((((((((...(((...((((((..................((((((........))))))........))))))...)))...))))))))........))))))) (-20.98 = -23.07 + 2.09)


Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 12:35:29 2006