
| Sequence ID | X_DroMel_CAF1 |
|---|---|
| Location | 17,962,153 – 17,962,254 |
| Length | 101 |
| Max. P | 0.996713 |

| Location | 17,962,153 – 17,962,254 |
|---|---|
| Length | 101 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 84.19 |
| Mean single sequence MFE | -37.88 |
| Consensus MFE | -35.95 |
| Energy contribution | -36.01 |
| Covariance contribution | 0.06 |
| Combinations/Pair | 1.04 |
| Mean z-score | -3.33 |
| Structure conservation index | 0.95 |
| SVM decision value | 2.74 |
| SVM RNA-class probability | 0.996713 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
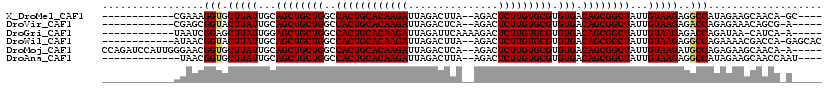
>X_DroMel_CAF1 17962153 101 + 22224390 ------------CGAAAGGUGCUUAUUGCAGCUGCUGGCCACUGCACAAGAUUAGACUUA--AGACUCUUGUGCGUGUGACAGCGGCUAUUGUAAGAGGCCAUAGAAGCAACA-GC---- ------------.....(((.(((((...((((((((..((((((((((((.........--....))))))))).))).))))))))...)))))..)))............-..---- ( -37.82) >DroVir_CAF1 36538 100 + 1 ------------CGAGCGGUACUUAUUGCAGCUGCUGGCCACUGCACAAGAUUAGACUCA--AGACUCUUGUGCGUGUGACAGCGGCUAUUGUAAGAGACCAGAGAAACAGCG-A----- ------------.....(((.(((((...((((((((..((((((((((((.........--....))))))))).))).))))))))...)))))..)))............-.----- ( -36.82) >DroGri_CAF1 30718 101 + 1 ------------UAAUCGGAGCUUAUUGGAGCUGCUGGCCACUGCACAAGAUUAGAUUCAAAAGACUCUUGUGCGUGUGACAGCGGCUAUUGUAAGAGACCAGAUAA-CAUCA-A----- ------------..(((((..(((((...((((((((..((((((((((((...............))))))))).))).))))))))...)))))...)).)))..-.....-.----- ( -37.36) >DroWil_CAF1 28322 105 + 1 ------------AUAACGGUACUUAUUGCAGCUGCUGGCCACUGCACAAGAUUAGACUUA--AGACUCUUGUGCGUGUGACAGCGGCUAUUGUAAGAGGCCAGAAAACGACCA-GAGCAC ------------.....(((.(((((...((((((((..((((((((((((.........--....))))))))).))).))))))))...)))))..)))............-...... ( -37.12) >DroMoj_CAF1 33027 112 + 1 CCAGAUCCAUUGGGAACGGUGCUUAUUGCAGCUGCUGGCCACUGCACAAGAUUAGACUCA--AGACUCUUGUGCGUGUGACAGCGGCUAUUGUAAGAUGCCAGAGAAGCAACA-A----- ...(.(((....))).)(((((((((...((((((((..((((((((((((.........--....))))))))).))).))))))))...))))).))))............-.----- ( -40.22) >DroAna_CAF1 21778 101 + 1 -------------UAACGGUGCUUAUUGCAGCUGCUGGCCACUGCACAAGAUUAGACUUA--AGACUCUUGUGCGUGUGACAGCGGCUAUUGUAAGAGGCCAUAGAAGCAACCAAU---- -------------....(((.(((((...((((((((..((((((((((((.........--....))))))))).))).))))))))...)))))..)))...............---- ( -37.92) >consensus ____________CGAACGGUGCUUAUUGCAGCUGCUGGCCACUGCACAAGAUUAGACUCA__AGACUCUUGUGCGUGUGACAGCGGCUAUUGUAAGAGGCCAGAGAAGCAACA_A_____ .................(((.(((((...((((((((..((((((((((((...............))))))))).))).))))))))...)))))..)))................... (-35.95 = -36.01 + 0.06)

| Location | 17,962,153 – 17,962,254 |
|---|---|
| Length | 101 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 84.19 |
| Mean single sequence MFE | -32.73 |
| Consensus MFE | -28.86 |
| Energy contribution | -29.03 |
| Covariance contribution | 0.17 |
| Combinations/Pair | 1.00 |
| Mean z-score | -2.43 |
| Structure conservation index | 0.88 |
| SVM decision value | 1.46 |
| SVM RNA-class probability | 0.956125 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
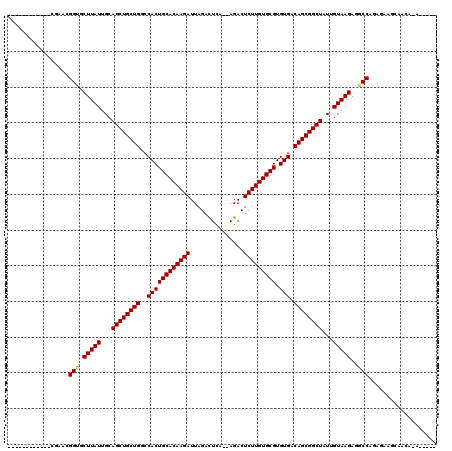
>X_DroMel_CAF1 17962153 101 - 22224390 ----GC-UGUUGCUUCUAUGGCCUCUUACAAUAGCCGCUGUCACACGCACAAGAGUCU--UAAGUCUAAUCUUGUGCAGUGGCCAGCAGCUGCAAUAAGCACCUUUCG------------ ----((-((((((..((..(((...........)))((((((((..((((((((....--.........)))))))).)))).))))))..))))).)))........------------ ( -34.12) >DroVir_CAF1 36538 100 - 1 -----U-CGCUGUUUCUCUGGUCUCUUACAAUAGCCGCUGUCACACGCACAAGAGUCU--UGAGUCUAAUCUUGUGCAGUGGCCAGCAGCUGCAAUAAGUACCGCUCG------------ -----.-............(((..((((...((((.((((((((..((((((((....--.........)))))))).)))).)))).))))...)))).))).....------------ ( -31.52) >DroGri_CAF1 30718 101 - 1 -----U-UGAUG-UUAUCUGGUCUCUUACAAUAGCCGCUGUCACACGCACAAGAGUCUUUUGAAUCUAAUCUUGUGCAGUGGCCAGCAGCUCCAAUAAGCUCCGAUUA------------ -----.-.....-..(((.((...((((....(((.((((((((..((((((((.((....))......)))))))).)))).)))).)))....))))..)))))..------------ ( -29.30) >DroWil_CAF1 28322 105 - 1 GUGCUC-UGGUCGUUUUCUGGCCUCUUACAAUAGCCGCUGUCACACGCACAAGAGUCU--UAAGUCUAAUCUUGUGCAGUGGCCAGCAGCUGCAAUAAGUACCGUUAU------------ (((((.-.(((((.....)))))........((((.((((((((..((((((((....--.........)))))))).)))).)))).)))).....)))))......------------ ( -34.32) >DroMoj_CAF1 33027 112 - 1 -----U-UGUUGCUUCUCUGGCAUCUUACAAUAGCCGCUGUCACACGCACAAGAGUCU--UGAGUCUAAUCUUGUGCAGUGGCCAGCAGCUGCAAUAAGCACCGUUCCCAAUGGAUCUGG -----(-((((((..((..(((...........)))((((((((..((((((((....--.........)))))))).)))).))))))..)))))))...(((.(((....)))..))) ( -34.72) >DroAna_CAF1 21778 101 - 1 ----AUUGGUUGCUUCUAUGGCCUCUUACAAUAGCCGCUGUCACACGCACAAGAGUCU--UAAGUCUAAUCUUGUGCAGUGGCCAGCAGCUGCAAUAAGCACCGUUA------------- ----...(((.((((...((........)).((((.((((((((..((((((((....--.........)))))))).)))).)))).))))....)))))))....------------- ( -32.42) >consensus _____U_UGUUGCUUCUCUGGCCUCUUACAAUAGCCGCUGUCACACGCACAAGAGUCU__UAAGUCUAAUCUUGUGCAGUGGCCAGCAGCUGCAAUAAGCACCGUUCG____________ ...................((...((((...((((.((((((((..((((((((...............)))))))).)))).)))).))))...))))..))................. (-28.86 = -29.03 + 0.17)


Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 12:31:30 2006