
| Sequence ID | X_DroMel_CAF1 |
|---|---|
| Location | 17,948,804 – 17,948,909 |
| Length | 105 |
| Max. P | 0.996442 |

| Location | 17,948,804 – 17,948,909 |
|---|---|
| Length | 105 |
| Sequences | 6 |
| Columns | 106 |
| Reading direction | forward |
| Mean pairwise identity | 76.91 |
| Mean single sequence MFE | -41.22 |
| Consensus MFE | -22.70 |
| Energy contribution | -23.20 |
| Covariance contribution | 0.50 |
| Combinations/Pair | 1.23 |
| Mean z-score | -3.45 |
| Structure conservation index | 0.55 |
| SVM decision value | 2.70 |
| SVM RNA-class probability | 0.996442 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
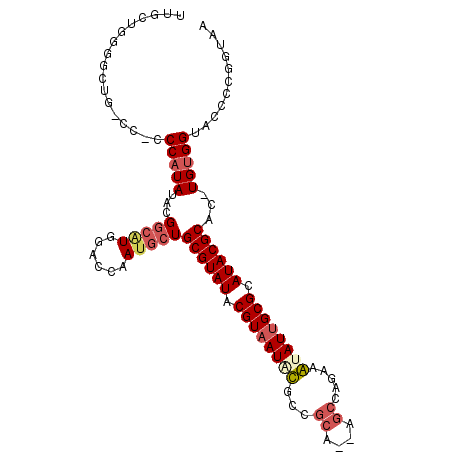
>X_DroMel_CAF1 17948804 105 + 22224390 UCACCGGGGUACCACA-GUGCGUAUGCGCCAUAUUGCUGGCAUAUGCCGCAUAUUACGUAUACGCAGCAUUGGUUCACGCCGUAUAUGGGAGGACAACCCCAGCAA ....(((.((((((..-(((((((((((..((((.((.(((....)))))))))..)))))))))).)..))))..)).)))....((((.(.....))))).... ( -39.20) >DroPse_CAF1 11932 102 + 1 CUAUCGGGG--CCACAUAUGCGUAUGCGCAAUGCAUAUAGCG--AGCGGCGUAUUACGUAUACGCAGCAUUUGCCCAUGCCGUAUAUGGGGGCACUGCUACUACAU ....(((.(--((.((((((((((((.(((((((.....)))--.((.((((((.....)))))).))..)))).)))).))))))))..))).)))......... ( -37.70) >DroEre_CAF1 8740 102 + 1 UCACCGGGGUACCACA-GUGCGUAUGCGCAAUAUUGCUGGCUUAUGCCGCACAUUACGUAUACGCAGCAUUGGUCCACGCCGUAUAAGGG-GG-CAACCCCAGCA- ....(((.((((((..-(((((((((((.(((..(((.(((....)))))).))).)))))))))).)..))))..)).))).....(((-(.-...))))....- ( -44.30) >DroYak_CAF1 8843 104 + 1 UUACCGGGGUACCACA-GUGCGUAUGCGCAAUAUUUCUGGCUUAUGCCGCACAUUACGUAUACGCAGCAUUGGUCCACUCCGUAUAUGGG-GGCCAACCCCAGCAA ....((((((((((..-(((((((((((.(((.....((((....))))...))).)))))))))).)..))))..))))))....((((-(.....))))).... ( -40.80) >DroAna_CAF1 5627 97 + 1 UCUACGGCGUACCACA-GUGCGUAUGCGCAAUACUUAUGGC------GGCGUAUUACGUAUACGCACCGUUGCCUCAUGCCGUAUAUGGG-GG-CAGCCCCAGACA ..((((((((.(.((.-(((((((((((.(((((.......------...))))).))))))))))).)).)....))))))))..((((-(.-...))))).... ( -40.60) >DroPer_CAF1 10257 102 + 1 CUAUCGGGG--CCACAUAUGCGUAUGCGCAAUGCAUAUAGCG--AGCGGCGUAUUACGUAUACGCAGCAUUUGCCCAUGCCGUAUAUGGGGGCCCUGCUACUACAU ....(((((--((.((((((((((((.(((((((.....)))--.((.((((((.....)))))).))..)))).)))).))))))))..)))))))......... ( -44.70) >consensus UCACCGGGGUACCACA_GUGCGUAUGCGCAAUACUUAUGGCG__UGCCGCAUAUUACGUAUACGCAGCAUUGGCCCACGCCGUAUAUGGG_GG_CAACCCCAGCAA .....(((((........((((((((((.(((((.....((....))...))))).)))))))))).......((((.........))))......)))))..... (-22.70 = -23.20 + 0.50)


| Location | 17,948,804 – 17,948,909 |
|---|---|
| Length | 105 |
| Sequences | 6 |
| Columns | 106 |
| Reading direction | reverse |
| Mean pairwise identity | 76.91 |
| Mean single sequence MFE | -41.72 |
| Consensus MFE | -21.34 |
| Energy contribution | -22.07 |
| Covariance contribution | 0.72 |
| Combinations/Pair | 1.12 |
| Mean z-score | -3.24 |
| Structure conservation index | 0.51 |
| SVM decision value | 2.55 |
| SVM RNA-class probability | 0.995147 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 17948804 105 - 22224390 UUGCUGGGGUUGUCCUCCCAUAUACGGCGUGAACCAAUGCUGCGUAUACGUAAUAUGCGGCAUAUGCCAGCAAUAUGGCGCAUACGCAC-UGUGGUACCCCGGUGA (..(((((((.......(((((....(((((.....(((((((((((.....)))))))))))..((((......))))...)))))..-))))).)))))))..) ( -44.20) >DroPse_CAF1 11932 102 - 1 AUGUAGUAGCAGUGCCCCCAUAUACGGCAUGGGCAAAUGCUGCGUAUACGUAAUACGCCGCU--CGCUAUAUGCAUUGCGCAUACGCAUAUGUGG--CCCCGAUAG .....((((((.(((((((......))...)))))..))))))((((.....))))(((((.--.((.....))..((((....))))...))))--)........ ( -33.40) >DroEre_CAF1 8740 102 - 1 -UGCUGGGGUUG-CC-CCCUUAUACGGCGUGGACCAAUGCUGCGUAUACGUAAUGUGCGGCAUAAGCCAGCAAUAUUGCGCAUACGCAC-UGUGGUACCCCGGUGA -.((((((((.(-((-.........(((((......)))))((((((.(((((((((((((....))).)).)))))))).))))))..-...))))))))))).. ( -46.70) >DroYak_CAF1 8843 104 - 1 UUGCUGGGGUUGGCC-CCCAUAUACGGAGUGGACCAAUGCUGCGUAUACGUAAUGUGCGGCAUAAGCCAGAAAUAUUGCGCAUACGCAC-UGUGGUACCCCGGUAA .(((((((((...((-(((......)).).))((((....(((((((.((((((((..(((....)))....)))))))).))))))).-..))))))))))))). ( -44.70) >DroAna_CAF1 5627 97 - 1 UGUCUGGGGCUG-CC-CCCAUAUACGGCAUGAGGCAACGGUGCGUAUACGUAAUACGCC------GCCAUAAGUAUUGCGCAUACGCAC-UGUGGUACGCCGUAGA ....(((((...-.)-))))..((((((.....((.(((((((((((.((((((((...------.......)))))))).))))))))-))).))..)))))).. ( -45.00) >DroPer_CAF1 10257 102 - 1 AUGUAGUAGCAGGGCCCCCAUAUACGGCAUGGGCAAAUGCUGCGUAUACGUAAUACGCCGCU--CGCUAUAUGCAUUGCGCAUACGCAUAUGUGG--CCCCGAUAG .(((....)))(((((..(((((.((..(((.((((..((.((((((.....)))))).)).--.((.....)).)))).))).)).))))).))--)))...... ( -36.30) >consensus UUGCUGGGGCUG_CC_CCCAUAUACGGCAUGGACCAAUGCUGCGUAUACGUAAUACGCCGCA__AGCCAGAAAUAUUGCGCAUACGCAC_UGUGGUACCCCGGUAA .................(((((...(((((......)))))((((((.((((((((...((....)).....)))))))).))))))...)))))........... (-21.34 = -22.07 + 0.72)


Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 12:31:22 2006