
| Sequence ID | X_DroMel_CAF1 |
|---|---|
| Location | 17,929,510 – 17,929,626 |
| Length | 116 |
| Max. P | 0.771291 |

| Location | 17,929,510 – 17,929,626 |
|---|---|
| Length | 116 |
| Sequences | 6 |
| Columns | 116 |
| Reading direction | forward |
| Mean pairwise identity | 90.63 |
| Mean single sequence MFE | -38.10 |
| Consensus MFE | -28.75 |
| Energy contribution | -29.33 |
| Covariance contribution | 0.59 |
| Combinations/Pair | 1.16 |
| Mean z-score | -2.97 |
| Structure conservation index | 0.75 |
| SVM decision value | 0.04 |
| SVM RNA-class probability | 0.551248 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 17929510 116 + 22224390 CUGAAAACCAGACCCAACAAGCUGGCUCUGCUCAAGAGCAGCAGCAGCAAGGAUCUGACCCGCCUGUUCCUGGACGAUUCAACCAACACAGCCGUGCUGGUGUCCAACACCAGUAU ........((((.((.....((((.(((((((....))))).))))))..)).))))...((.((((...(((.........)))..)))).))((((((((.....)))))))). ( -37.30) >DroSec_CAF1 6036 116 + 1 CUGAAAACCAGACCCAACAAGCUGGCGCUGCUCAAGAGCAGCAGCAGCAAGGAUCUGACCCGCCUGUUUCUGGACGAUUCAACCAACACAGCCGUGCUGGUGUCCAACACCAGCAU .((((..(((((..((....((((..((((((....))))))..))))..((.......))...))..)))))....))))............(((((((((.....))))))))) ( -41.70) >DroSim_CAF1 5192 116 + 1 CUGAAAACCAGACCCAACAAGCUGGCGCUGCUCAAGAGCAGCAGCAGCAAGGAUCUGACCCGCCUGUACCUGGACGAUUCAACCAACACAGCUGUGCUGGUGUCCAACACCAGCAU (((.....)))........(((((((((((((....)))))).)).....((...(((..((((.......)).))..))).))....)))))(((((((((.....))))))))) ( -41.90) >DroEre_CAF1 5971 116 + 1 CUGAAAACCAGACCCAACAAGCUGGCGCUGCUUAAGAGCAGCAGCACCAAAGAUCUGACCCGCCUGUUCCUGGACGAUUCAACCAACACAGCUGUGCUGGUGUCCAACACCAGUCU (((.....))).....(((.((((((((((((....)))))).))..........(((..((((.......)).))..))).......)))))))(((((((.....))))))).. ( -36.00) >DroYak_CAF1 5851 116 + 1 CUGAAAACCAGACCCAACAAGCUGGCGCUGCUCAAGAGCAGCAGCACCAAGGAUCUGACCCGCCUGUUCCUGGACGACUCAACCAACACAGCUGUACUGGUGUCCAACACCAGUCU (((.....))).....(((.((((((((((((....)))))).)).....((...(((..((((.......)).))..))).))....)))))))(((((((.....))))))).. ( -37.80) >DroAna_CAF1 5335 116 + 1 UUGAAAUCCAAACCCAAUAAGCUGGCCCUGCUCAAGAGCAGCAGCACCAAGGAUCUCACCAGACUCUUCAUCGACGAAGGCACCAACACGACUCUGGUGGUGUCCAACACCAGCCU ....................(((((..(((((....))))).........((((..(((((((.(((((......)))(....).....)).)))))))..))))....))))).. ( -33.90) >consensus CUGAAAACCAGACCCAACAAGCUGGCGCUGCUCAAGAGCAGCAGCACCAAGGAUCUGACCCGCCUGUUCCUGGACGAUUCAACCAACACAGCUGUGCUGGUGUCCAACACCAGCAU (((.....))).........((((((((((((....)))))).)).....((...(((..((((.......)).))..))).))....))))...(((((((.....))))))).. (-28.75 = -29.33 + 0.59)

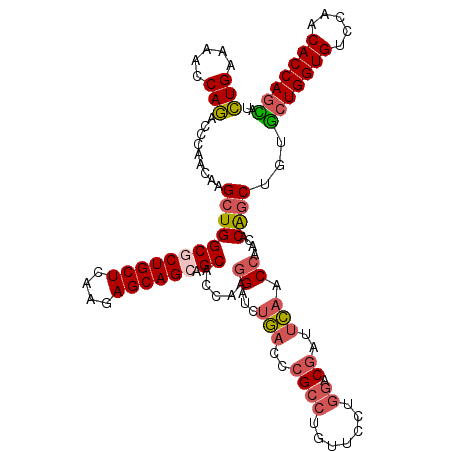

| Location | 17,929,510 – 17,929,626 |
|---|---|
| Length | 116 |
| Sequences | 6 |
| Columns | 116 |
| Reading direction | reverse |
| Mean pairwise identity | 90.63 |
| Mean single sequence MFE | -49.53 |
| Consensus MFE | -38.70 |
| Energy contribution | -39.45 |
| Covariance contribution | 0.75 |
| Combinations/Pair | 1.09 |
| Mean z-score | -2.95 |
| Structure conservation index | 0.78 |
| SVM decision value | 0.53 |
| SVM RNA-class probability | 0.771291 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 17929510 116 - 22224390 AUACUGGUGUUGGACACCAGCACGGCUGUGUUGGUUGAAUCGUCCAGGAACAGGCGGGUCAGAUCCUUGCUGCUGCUGCUCUUGAGCAGAGCCAGCUUGUUGGGUCUGGUUUUCAG ...(((.(.((((((((((((((....))))))))......)))))).).)))(..(..(((((((..((((((.(((((....))))))).)))).....)))))))..)..).. ( -48.90) >DroSec_CAF1 6036 116 - 1 AUGCUGGUGUUGGACACCAGCACGGCUGUGUUGGUUGAAUCGUCCAGAAACAGGCGGGUCAGAUCCUUGCUGCUGCUGCUCUUGAGCAGCGCCAGCUUGUUGGGUCUGGUUUUCAG .((((((((.....))))))))((.((((.((((.........))))..)))).))(..(((((((..((((.(((((((....))))))).)))).....)))))))..)..... ( -53.70) >DroSim_CAF1 5192 116 - 1 AUGCUGGUGUUGGACACCAGCACAGCUGUGUUGGUUGAAUCGUCCAGGUACAGGCGGGUCAGAUCCUUGCUGCUGCUGCUCUUGAGCAGCGCCAGCUUGUUGGGUCUGGUUUUCAG ...(((...((((((((((((((....))))))))......))))))...)))(..(..(((((((..((((.(((((((....))))))).)))).....)))))))..)..).. ( -51.80) >DroEre_CAF1 5971 116 - 1 AGACUGGUGUUGGACACCAGCACAGCUGUGUUGGUUGAAUCGUCCAGGAACAGGCGGGUCAGAUCUUUGGUGCUGCUGCUCUUAAGCAGCGCCAGCUUGUUGGGUCUGGUUUUCAG ...(((.(.((((((((((((((....))))))))......)))))).).)))(..(..((((((((((((((((((.......)))))))))))......)))))))..)..).. ( -50.60) >DroYak_CAF1 5851 116 - 1 AGACUGGUGUUGGACACCAGUACAGCUGUGUUGGUUGAGUCGUCCAGGAACAGGCGGGUCAGAUCCUUGGUGCUGCUGCUCUUGAGCAGCGCCAGCUUGUUGGGUCUGGUUUUCAG ..(((((((.....))))))).(((((.....))))).(....((((((((((((((((...)))).((((((((((.(....))))))))))))))))))...)))))....).. ( -48.70) >DroAna_CAF1 5335 116 - 1 AGGCUGGUGUUGGACACCACCAGAGUCGUGUUGGUGCCUUCGUCGAUGAAGAGUCUGGUGAGAUCCUUGGUGCUGCUGCUCUUGAGCAGGGCCAGCUUAUUGGGUUUGGAUUUCAA .....((((.....))))((((((.((..((((..(....)..))))...)).))))))(((((((.((((.(((((.(....)))))).))))(((.....)))..))))))).. ( -43.50) >consensus AGACUGGUGUUGGACACCAGCACAGCUGUGUUGGUUGAAUCGUCCAGGAACAGGCGGGUCAGAUCCUUGCUGCUGCUGCUCUUGAGCAGCGCCAGCUUGUUGGGUCUGGUUUUCAG ...(((...((((((((((((((....))))))))......))))))...)))(..(..(((((((.....(((((((((....)))))...)))).....)))))))..)..).. (-38.70 = -39.45 + 0.75)



Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 12:31:11 2006