
| Sequence ID | X_DroMel_CAF1 |
|---|---|
| Location | 17,348,580 – 17,348,727 |
| Length | 147 |
| Max. P | 0.988663 |

| Location | 17,348,580 – 17,348,700 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 82.40 |
| Mean single sequence MFE | -32.48 |
| Consensus MFE | -25.80 |
| Energy contribution | -26.16 |
| Covariance contribution | 0.36 |
| Combinations/Pair | 1.26 |
| Mean z-score | -2.40 |
| Structure conservation index | 0.79 |
| SVM decision value | 1.78 |
| SVM RNA-class probability | 0.977128 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 17348580 120 - 22224390 AAUGGCCACAGAUGCAGGUGGGAGACACUUGAAUAUUAUGCAUUUGUAAAAGCCAGUUGGAUUUCCAUUUGGCUAACUGGUUAUGUGGUUAUGUUAUUCUGAAAUCUAAUAAUAACAAAG .(((((((((((((((((((.....)))).........))))))).....((((((((((....((....))))))))))))..))))))))((((((.(........).)))))).... ( -34.20) >DroSec_CAF1 65603 120 - 1 AAUGGCCACAAAUGCAGGUGGGAGACACUUGAAUAUUAUGCAUUGGUAAAAGUCAGUUGGAUUUCCAUUUGGCUAACUGGUUAUGUGGUUAUGUUAUUCUAAAAGCUAAUAAUAACAAAG .(((((((((....((((((.....))))))..........((..((...((((((.(((....))).)))))).))..))..)))))))))((((((............)))))).... ( -31.80) >DroSim_CAF1 55575 120 - 1 AAUGGCCACAGAUGCAGGUGGGAGACACUUGAAUAUUAUGCAUUUGUGAAAGCCAGUUGGAUUUCCAUUUGGCUAACUGGUUAUGUGGUUAUGUUAUUCUAAAAGCUAAUAAUAACAAAG .(((((((((((((((((((.....)))).........))))))).....((((((((((....((....))))))))))))..))))))))((((((............)))))).... ( -33.40) >DroEre_CAF1 66168 100 - 1 AAUGGCCACAGUUGCAGGUGGGAGACACUUGACUAUUAUGCAUUUGUAAAAGCUAGUUGGUUUUCGGCUUUC-CAUUUGGCUAUCUGGCGAUGU-------------------GGCCAUG .(((((((((.((((((((((....)......(......))))))))))..(((((.(((((...((....)-)....))))).)))))..)))-------------------)))))). ( -34.70) >DroYak_CAF1 57186 119 - 1 AAUGGCCACAAAUGCAGGUGGGAGACACUUGACUAUUAUGCAUUUGUUAAAGCCAGUUGGAUUUCCAUUAGG-UAUUUGGUUAUGUGGUAAUGUUAUUCUGAACGCUAAUGAUAACAAUG .((((((((((((((((((((..((...))..))))).)))))))))....(((...(((....)))...))-)....)))))).......(((((((.(........).)))))))... ( -28.30) >consensus AAUGGCCACAGAUGCAGGUGGGAGACACUUGAAUAUUAUGCAUUUGUAAAAGCCAGUUGGAUUUCCAUUUGGCUAACUGGUUAUGUGGUUAUGUUAUUCUAAAAGCUAAUAAUAACAAAG .(((((((((....((((((.....))))))...................((((((((((....((....)))))))))))).)))))))))((((((.(........).)))))).... (-25.80 = -26.16 + 0.36)

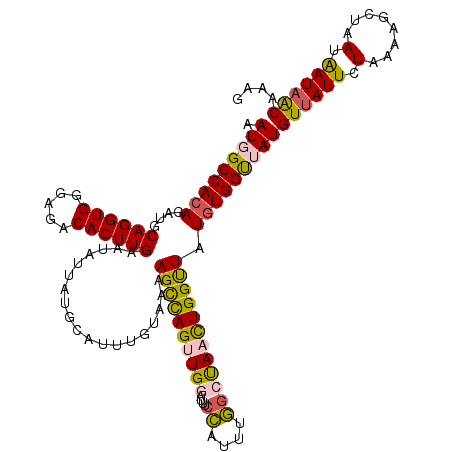
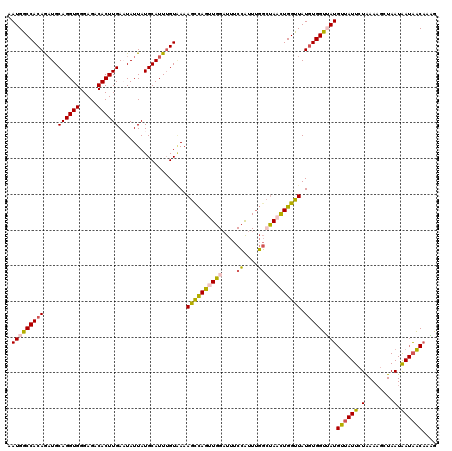
| Location | 17,348,620 – 17,348,727 |
|---|---|
| Length | 107 |
| Sequences | 5 |
| Columns | 107 |
| Reading direction | reverse |
| Mean pairwise identity | 90.65 |
| Mean single sequence MFE | -32.70 |
| Consensus MFE | -29.24 |
| Energy contribution | -28.68 |
| Covariance contribution | -0.56 |
| Combinations/Pair | 1.20 |
| Mean z-score | -1.88 |
| Structure conservation index | 0.89 |
| SVM decision value | 2.13 |
| SVM RNA-class probability | 0.988663 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 17348620 107 - 22224390 CUGCCAGCGGUAGUGGAGCAUCUAAUAAAUGGCCACAGAUGCAGGUGGGAGACACUUGAAUAUUAUGCAUUUGUAAAAGCCAGUUGGAUUUCCAUUUGGCUAACUGG ...((((.(((.((((((.(((((((...((((.(((((((((((((.....)))).........)))))))))....)))))))))))))))))...)))..)))) ( -35.30) >DroSec_CAF1 65643 107 - 1 CUGCCAGCGUUAGUGGAGCAUCUAAUAAAUGGCCACAAAUGCAGGUGGGAGACACUUGAAUAUUAUGCAUUGGUAAAAGUCAGUUGGAUUUCCAUUUGGCUAACUGG .((((((((((.((((..(((.......))).)))).)))))(((((.....))))).............)))))..((((((.(((....))).))))))...... ( -31.20) >DroSim_CAF1 55615 107 - 1 CUGCCAGCGGUAGUGGAGCACCUAAUAAAUGGCCACAGAUGCAGGUGGGAGACACUUGAAUAUUAUGCAUUUGUGAAAGCCAGUUGGAUUUCCAUUUGGCUAACUGG ..((((....(((.(.....)))).....))))((((((((((((((.....)))).........))))))))))..((((((.(((....))).))))))...... ( -35.20) >DroEre_CAF1 66189 106 - 1 CUGCCAGCGGCAGUGGAGCAACUAAUAAAUGGCCACAGUUGCAGGUGGGAGACACUUGACUAUUAUGCAUUUGUAAAAGCUAGUUGGUUUUCGGCUUUC-CAUUUGG (((((...)))))((((((((((.............))))))((((.((((((....(((((((.(((....))).))..))))).)))))).))))))-))..... ( -28.02) >DroYak_CAF1 57226 106 - 1 CUGCCAGCGGUAGUGGAGCAUCUAAUAAAUGGCCACAAAUGCAGGUGGGAGACACUUGACUAUUAUGCAUUUGUUAAAGCCAGUUGGAUUUCCAUUAGG-UAUUUGG .((((.....((((((((.(((((((...((((.((((((((((((((..((...))..))))).)))))))))....)))))))))))))))))))))-))..... ( -33.80) >consensus CUGCCAGCGGUAGUGGAGCAUCUAAUAAAUGGCCACAGAUGCAGGUGGGAGACACUUGAAUAUUAUGCAUUUGUAAAAGCCAGUUGGAUUUCCAUUUGGCUAACUGG ...((((....(((((((.(((((((...((((.(((((((((((((.....)))).........)))))))))....)))))))))))))))))).......)))) (-29.24 = -28.68 + -0.56)

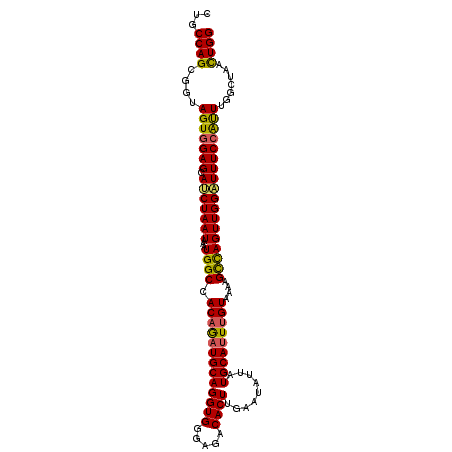

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 12:26:14 2006