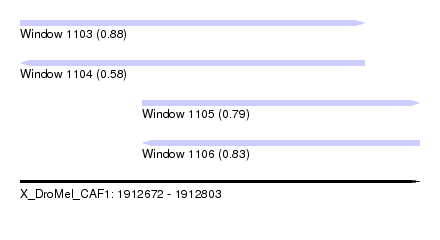
| Sequence ID | X_DroMel_CAF1 |
|---|---|
| Location | 1,912,672 – 1,912,803 |
| Length | 131 |
| Max. P | 0.884228 |

| Location | 1,912,672 – 1,912,785 |
|---|---|
| Length | 113 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 81.85 |
| Mean single sequence MFE | -51.12 |
| Consensus MFE | -37.41 |
| Energy contribution | -36.87 |
| Covariance contribution | -0.55 |
| Combinations/Pair | 1.42 |
| Mean z-score | -2.45 |
| Structure conservation index | 0.73 |
| SVM decision value | 0.93 |
| SVM RNA-class probability | 0.884228 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
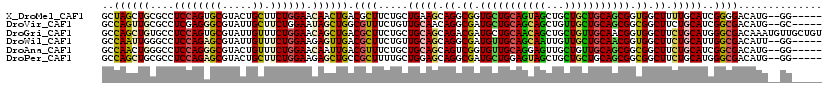
>X_DroMel_CAF1 1912672 113 + 22224390 GCUAGCUGCGCCUCCAGUGCGUACUGCUUCUGGAACAACUGACGCUUCUGCUGAAGCAGGCGGUGCUGCAGUAGCUGCUGCUGCAGCGGUGGCUUUUGCAUCGGGGACAUG--GG----- ((.(((..(((((((((.((.....))..))))).........(((((....))))).)))..(((((((((((...))))))))))))..)))...))...(....)...--..----- ( -49.60) >DroVir_CAF1 4598 113 + 1 GCCAGUUGCGCCUCGAGGGCGUAUUGCUUCUGGAAUAGCUGGCGUUUCUGUUGCAACAGGCGAUGCUGCAGCAGCUGUUGCUGCAGCGGCGGCUUCUGCAUCGGCGACAUG--GC----- ((((((((((((.....)))))(((.(....).))))))))))(((.(((.((((..((.((.(((((((((((...))))))))))).)).))..)))).))).)))...--..----- ( -53.40) >DroGri_CAF1 5439 120 + 1 GCCAGCUGUGCCUCCAGUGCGUAUUGUUUCUGGAACAGCUGACGCUUCUGCUGCAGCAGACGAUGCUGCAACAGCUGCUGUUGCAACGGUGGCUUCUGCAUGGGCGACAAAUGUUGCUGU (((((((((...(((((.((.....))..)))))))))))).((((..((((((((((.....)))))))..(((..((((....))))..)))...)))..)))).........))... ( -50.10) >DroWil_CAF1 3832 113 + 1 GCCAAUUGGGCCUCCAGAGCGUAUUGUUUCUGGAAGAGUUGACGCUUCUGUUGCAGCAGGCGAUGUUGCAGCAAUUGUUGCUGCAACGGUGGCUUCUGCAUUGGCGACAUU--GG----- .(((((.(.(((..((((((((.....(((.....)))...)))).)))).(((((.((((.(((((((((((.....))))))))).)).)))))))))..)))..))))--))----- ( -46.10) >DroAna_CAF1 6480 113 + 1 GCCAACUGGGCCUCCAGGGCGUACUGUUUCUGGAACAAUUGACGUUUCUGCUGCAGCAGUCGGUGUUGCAGGAGUUGCUGUUGCAGCGGCGGCUUCUGCAUCGGCGACAUG--GG----- (((..((((....)))))))...((((..(.(((((.......))))).)..))))..((((.((.(((((((((((((((....))))))))))))))).)).))))...--..----- ( -50.00) >DroPer_CAF1 4240 113 + 1 GCCAGCUGCGCCUCCAGAGCGUACUGCUUCUGGAAGAGCUGCCGCUUUUGCUGGAGCAGGCGAUGCUGGAGUAGCUGCUGCUGCAGCGGCGGCUUCUGCAUGGGCGACAUG--GG----- .(((....(((((((((..(((.(((((((.(.((((((....)))))).).))))))))))...))))((.(((((((((....))))))))).))....)))))...))--).----- ( -57.50) >consensus GCCAGCUGCGCCUCCAGAGCGUACUGCUUCUGGAACAGCUGACGCUUCUGCUGCAGCAGGCGAUGCUGCAGCAGCUGCUGCUGCAGCGGCGGCUUCUGCAUCGGCGACAUG__GG_____ ..((((((....((((((((.....)).)))))).)))))).((((.....(((((.((.((.(((((((((((...))))))))))).)).)).)))))..)))).............. (-37.41 = -36.87 + -0.55)

| Location | 1,912,672 – 1,912,785 |
|---|---|
| Length | 113 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 81.85 |
| Mean single sequence MFE | -41.13 |
| Consensus MFE | -23.58 |
| Energy contribution | -24.12 |
| Covariance contribution | 0.53 |
| Combinations/Pair | 1.29 |
| Mean z-score | -2.13 |
| Structure conservation index | 0.57 |
| SVM decision value | 0.09 |
| SVM RNA-class probability | 0.578276 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 1912672 113 - 22224390 -----CC--CAUGUCCCCGAUGCAAAAGCCACCGCUGCAGCAGCAGCUACUGCAGCACCGCCUGCUUCAGCAGAAGCGUCAGUUGUUCCAGAAGCAGUACGCACUGGAGGCGCAGCUAGC -----..--.........(((((..........(((((((.((...)).))))))).....((((....))))..)))))(((((((((((..((.....)).)))))...))))))... ( -38.50) >DroVir_CAF1 4598 113 - 1 -----GC--CAUGUCGCCGAUGCAGAAGCCGCCGCUGCAGCAACAGCUGCUGCAGCAUCGCCUGUUGCAACAGAAACGCCAGCUAUUCCAGAAGCAAUACGCCCUCGAGGCGCAACUGGC -----((--((((.(((((.(((((.((.((..(((((((((.....)))))))))..)).)).))))).)..........(((........))).............))))))..)))) ( -41.00) >DroGri_CAF1 5439 120 - 1 ACAGCAACAUUUGUCGCCCAUGCAGAAGCCACCGUUGCAACAGCAGCUGUUGCAGCAUCGUCUGCUGCAGCAGAAGCGUCAGCUGUUCCAGAAACAAUACGCACUGGAGGCACAGCUGGC ...(((((.....((((....)).)).......)))))....((..(((((((((((.....)))))))))))..))((((((((((((((............)))))...))))))))) ( -47.30) >DroWil_CAF1 3832 113 - 1 -----CC--AAUGUCGCCAAUGCAGAAGCCACCGUUGCAGCAACAAUUGCUGCAACAUCGCCUGCUGCAACAGAAGCGUCAACUCUUCCAGAAACAAUACGCUCUGGAGGCCCAAUUGGC -----((--(((...(((..(((((..((....(((((((((.....)))))))))...))...))))).((((.((((....((.....))......))))))))..)))...))))). ( -41.30) >DroAna_CAF1 6480 113 - 1 -----CC--CAUGUCGCCGAUGCAGAAGCCGCCGCUGCAACAGCAACUCCUGCAACACCGACUGCUGCAGCAGAAACGUCAAUUGUUCCAGAAACAGUACGCCCUGGAGGCCCAGUUGGC -----..--......((((((......(((.((((((((.((((...............).))).)))))).....(((..((((((.....)))))))))....)).)))...)))))) ( -30.66) >DroPer_CAF1 4240 113 - 1 -----CC--CAUGUCGCCCAUGCAGAAGCCGCCGCUGCAGCAGCAGCUACUCCAGCAUCGCCUGCUCCAGCAAAAGCGGCAGCUCUUCCAGAAGCAGUACGCUCUGGAGGCGCAGCUGGC -----..--......(((...((....(((((.((((.((((((.(((.....)))...).))))).))))....))))).((.((((((((.((.....)))))))))).)).)).))) ( -48.00) >consensus _____CC__CAUGUCGCCGAUGCAGAAGCCACCGCUGCAGCAGCAGCUGCUGCAGCAUCGCCUGCUGCAGCAGAAGCGUCAGCUGUUCCAGAAACAAUACGCACUGGAGGCGCAGCUGGC ...........(((.((....((((..((....(((((((.........)))))))...)))))).)).))).....(((((((.((((((............))))))....))))))) (-23.58 = -24.12 + 0.53)

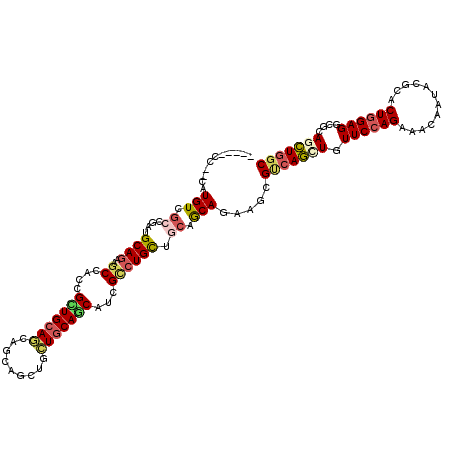
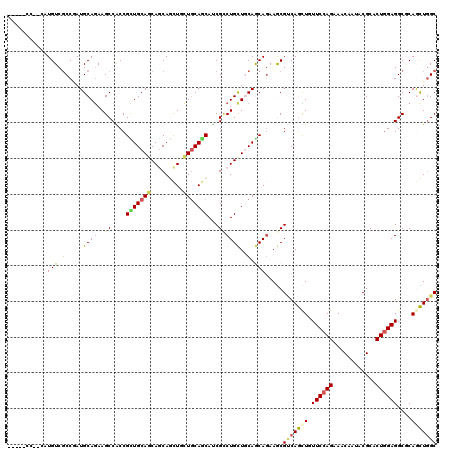
| Location | 1,912,712 – 1,912,803 |
|---|---|
| Length | 91 |
| Sequences | 6 |
| Columns | 106 |
| Reading direction | forward |
| Mean pairwise identity | 81.07 |
| Mean single sequence MFE | -43.65 |
| Consensus MFE | -32.95 |
| Energy contribution | -33.53 |
| Covariance contribution | 0.58 |
| Combinations/Pair | 1.16 |
| Mean z-score | -1.68 |
| Structure conservation index | 0.75 |
| SVM decision value | 0.58 |
| SVM RNA-class probability | 0.787881 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 1912712 91 + 22224390 GACGCUUCUGCUGAAGCAGGCGGUGCUGCAGUAGCUGCUGCUGCAGCGGUGGCUUUUGCAUCGGGGACAUGGGCACGGCCAUACCCACGCC--------------- ...(((((....))))).(((.(((((((((.(((..((((....))))..))).))))...(....)...))))).)))...........--------------- ( -43.80) >DroPse_CAF1 4631 88 + 1 GCCGCUUUUGCUGGAGCAGGCGAUGCUGGAGUAGCUGCUGCUGCAGCGGCGGCUUCUGCAUGGGCGACAUGGGCACCGCCAUACCGAG------------------ ...(((((....))))).((((.((((..((.(((((((((....))))))))).)).((((.....)))))))).))))........------------------ ( -38.60) >DroSec_CAF1 8122 91 + 1 GACGCUUCUGCUGCAGCAGACGGUGCUGCAGGAGCUGCUGCUGCAGCGGUGGCUUUUGCAUCGGGGACAUGGGCACUGCCAUGCCCACGCC--------------- ...(((.((((((((((((.((((.(.....).)))))))))))))))).)))........(((((.(((((......)))))))).))..--------------- ( -46.10) >DroEre_CAF1 5957 106 + 1 GACGCUUCUGCUGCAGCAGGCGGUGCUGGAGUAGCUGCUGCUGCAGCGGUGGCUUUUGCAUCGGCGACAUGGGCACGGCCAUGCCCACACCCACACCCACACCCAC ...(((.((((((((((((.((((((....))).))))))))))))))).)))..((((....))))..((((((......))))))................... ( -44.60) >DroYak_CAF1 8814 91 + 1 GACGCUUCUGCUGCAGCAGGCGGUGCUGCAGGAGUUGCUGCUGCAGCGGUGGCUUUUGCAUCGGCGACAUGGGCACGGCCAUGCCCACACC--------------- ..((((..(((.((((((.....))))))((((((..((((....))))..)))))))))..))))...((((((......))))))....--------------- ( -42.10) >DroAna_CAF1 6520 106 + 1 GACGUUUCUGCUGCAGCAGUCGGUGUUGCAGGAGUUGCUGUUGCAGCGGCGGCUUCUGCAUCGGCGACAUGGGCAUGGCCACGCCCACCCCCACACCAACGCCCAC ..((((.((((....))))..((((((((((((((((((((....)))))))))))))))..((.(...(((((........)))))..))))))))))))..... ( -46.70) >consensus GACGCUUCUGCUGCAGCAGGCGGUGCUGCAGGAGCUGCUGCUGCAGCGGUGGCUUUUGCAUCGGCGACAUGGGCACGGCCAUGCCCACACC_______________ ...((..((((((((((((.((((.........))))))))))))))))..)).........(....).((((((......))))))................... (-32.95 = -33.53 + 0.58)

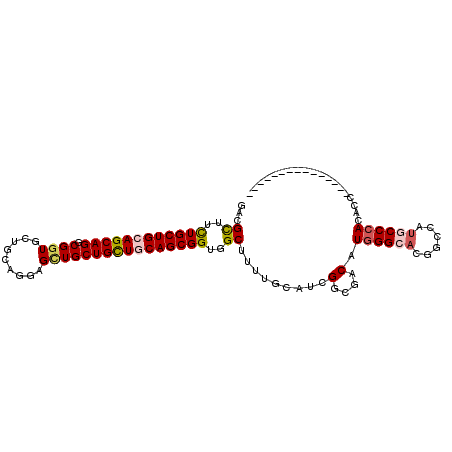
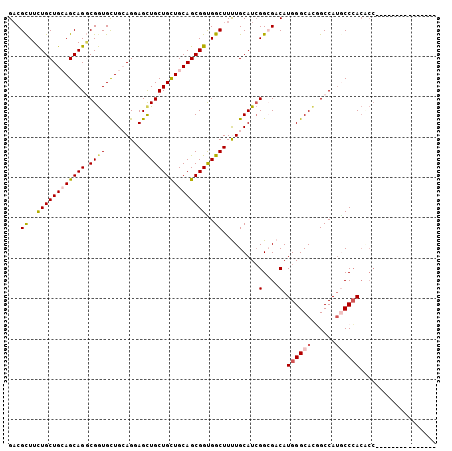
| Location | 1,912,712 – 1,912,803 |
|---|---|
| Length | 91 |
| Sequences | 6 |
| Columns | 106 |
| Reading direction | reverse |
| Mean pairwise identity | 81.07 |
| Mean single sequence MFE | -42.37 |
| Consensus MFE | -31.19 |
| Energy contribution | -33.25 |
| Covariance contribution | 2.06 |
| Combinations/Pair | 1.14 |
| Mean z-score | -1.87 |
| Structure conservation index | 0.74 |
| SVM decision value | 0.71 |
| SVM RNA-class probability | 0.830638 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 1912712 91 - 22224390 ---------------GGCGUGGGUAUGGCCGUGCCCAUGUCCCCGAUGCAAAAGCCACCGCUGCAGCAGCAGCUACUGCAGCACCGCCUGCUUCAGCAGAAGCGUC ---------------(((((((((((....)))))))))))...(((((..........(((((((.((...)).))))))).....((((....))))..))))) ( -40.60) >DroPse_CAF1 4631 88 - 1 ------------------CUCGGUAUGGCGGUGCCCAUGUCGCCCAUGCAGAAGCCGCCGCUGCAGCAGCAGCUACUCCAGCAUCGCCUGCUCCAGCAAAAGCGGC ------------------.((.((((((((..(....)..)).)))))).)).(((((.((((.((((((.(((.....)))...).))))).))))....))))) ( -33.80) >DroSec_CAF1 8122 91 - 1 ---------------GGCGUGGGCAUGGCAGUGCCCAUGUCCCCGAUGCAAAAGCCACCGCUGCAGCAGCAGCUCCUGCAGCACCGUCUGCUGCAGCAGAAGCGUC ---------------(((((((((((....)))))))))))...(((((..........(((((....)))))..((((((((.....)))))))).....))))) ( -45.60) >DroEre_CAF1 5957 106 - 1 GUGGGUGUGGGUGUGGGUGUGGGCAUGGCCGUGCCCAUGUCGCCGAUGCAAAAGCCACCGCUGCAGCAGCAGCUACUCCAGCACCGCCUGCUGCAGCAGAAGCGUC ((((.(....((((.(((((((((((....)))))))...)))).))))...).)))).(((((((((((.(((.....)))...).))))))))))......... ( -49.40) >DroYak_CAF1 8814 91 - 1 ---------------GGUGUGGGCAUGGCCGUGCCCAUGUCGCCGAUGCAAAAGCCACCGCUGCAGCAGCAACUCCUGCAGCACCGCCUGCUGCAGCAGAAGCGUC ---------------((..(((((((....)))))))..))...(((((..........((((...))))..((.((((((((.....)))))))).))..))))) ( -41.10) >DroAna_CAF1 6520 106 - 1 GUGGGCGUUGGUGUGGGGGUGGGCGUGGCCAUGCCCAUGUCGCCGAUGCAGAAGCCGCCGCUGCAACAGCAACUCCUGCAACACCGACUGCUGCAGCAGAAACGUC ...(((((((((((((((((((((((....)))))).....((.(.(((((.........))))).).)).))))))...)))))..((((....)))).)))))) ( -43.70) >consensus _______________GGCGUGGGCAUGGCCGUGCCCAUGUCGCCGAUGCAAAAGCCACCGCUGCAGCAGCAGCUACUGCAGCACCGCCUGCUGCAGCAGAAGCGUC ...............(((((((((((....)))))))))))...(((((..........(((((....)))))..((((((((.....)))))))).....))))) (-31.19 = -33.25 + 2.06)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 09:51:13 2006