
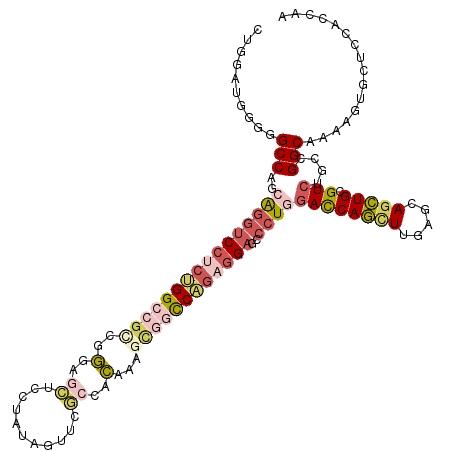
| Sequence ID | X_DroMel_CAF1 |
|---|---|
| Location | 16,976,508 – 16,976,628 |
| Length | 120 |
| Max. P | 0.889077 |

| Location | 16,976,508 – 16,976,628 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 78.56 |
| Mean single sequence MFE | -49.68 |
| Consensus MFE | -29.92 |
| Energy contribution | -31.78 |
| Covariance contribution | 1.87 |
| Combinations/Pair | 1.33 |
| Mean z-score | -1.83 |
| Structure conservation index | 0.60 |
| SVM decision value | 0.63 |
| SVM RNA-class probability | 0.805150 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 16976508 120 + 22224390 CUGGAUGGGGGCCAGCAGGUCCUCUGGCCGCCGGGAGCUCAUAUAGUUCGCCACAAAGCGGCCAGAGGAGACCUGGACCAGCUUGAGCAGCUGCGUCUGCCGGCAAAAGUGCUCAACCAA .(((.((((.(((.(((((((((((((((((.(((((((.....))))).)).....))))))))))))..)))(((((((((.....))))).)))))).))).......)))).))). ( -57.40) >DroVir_CAF1 4071 120 + 1 ACGCAGGGGCGCCAGCGCCUCCUCCGGUUUGCGCGUGCGCCUGUAGUUGCGCAGAAAGUUCUCGACGGUCCCUUCAAUCAAUUUCACUAGUUGCGUCGCCCGGCAAAAUUUCUCAAUUAA ..(((((((((....)))).))).(((((((((((.((.......)))))))))).......((((((......)...(((((.....)))))))))).)))))................ ( -32.70) >DroSim_CAF1 3487 120 + 1 CUGGAUGGGGGCCAGCAGGUCCUCUGGCCGCCGGGAGCUCCUAUAGUUCGCCACAAAGCGGCCAGAGGAACCCUGGACCAGCUUGAGCAGCUGCGUCUGCCGGCAAUAGUGCUCCACUAA .(((.((((((((.((.((((((((((((((.(((((((.....))))).)).....))))))))))).)))..(((((((((.....))))).)))))).))).......)))))))). ( -57.61) >DroEre_CAF1 4731 120 + 1 CUGGAUGGGCGCCAGCAGGUCCUCUGGCCGCCGGGAGCUCCUAUAGUUCGCAACAAAGCGGCCAGAGGAGCCCUGGACCAGCUUGAGCAGCUGCGUCUGCCGGCAAAAGUUCUCCACCAA .((((.(((((((.((.((((((((((((((.(.(((((.....))))).)......)))))))))))).))..(((((((((.....))))).)))))).)))....)))))))).... ( -56.60) >DroYak_CAF1 5176 120 + 1 CUGGAUGGGGGCCAGCAGGUCCUCUGGCCGUCGGCUGCUCCUAUAGUUCGCCACAAAGCGGCCAGAGGAGCCCUGGACCAGCUUGAGCAACUGCGUCUGCCGGCAAAAGUGCUCCACCAG ((((.(((((.((((..((((((((((((((.(((.(((.....)))..))).....)))))))))))).)))))).((.((..((((....)).)).)).))........))))))))) ( -53.60) >DroAna_CAF1 5830 120 + 1 CUGGACGGGUGCCAGCAGGUCCUCGGGACGACGGUGAGUCCUGUAGUUCUCCACAAACUUACCAGCAGAGCCCUCGAUCAGCUUCAGCAGCUGGGUCUCCCGGCAAUACUUCUCCACCAG ((((.((((..((....))..))))(((.((.(((((((..(((.(....).))).))))))).((.((((.........))))..)).((((((...))))))......))))).)))) ( -40.20) >consensus CUGGAUGGGGGCCAGCAGGUCCUCUGGCCGCCGGGAGCUCCUAUAGUUCGCCACAAAGCGGCCAGAGGAGCCCUGGACCAGCUUGAGCAGCUGCGUCUGCCGGCAAAAGUGCUCCACCAA ..........(((..((((((((((((((((..(..((...........))..)...))))))))))))..))))((((((((.....))))).)))....)))................ (-29.92 = -31.78 + 1.87)


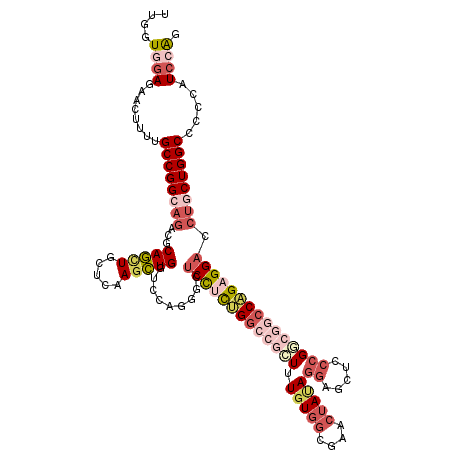
| Location | 16,976,508 – 16,976,628 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 78.56 |
| Mean single sequence MFE | -52.26 |
| Consensus MFE | -33.99 |
| Energy contribution | -37.02 |
| Covariance contribution | 3.03 |
| Combinations/Pair | 1.22 |
| Mean z-score | -1.87 |
| Structure conservation index | 0.65 |
| SVM decision value | 0.95 |
| SVM RNA-class probability | 0.889077 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 16976508 120 - 22224390 UUGGUUGAGCACUUUUGCCGGCAGACGCAGCUGCUCAAGCUGGUCCAGGUCUCCUCUGGCCGCUUUGUGGCGAACUAUAUGAGCUCCCGGCGGCCAGAGGACCUGCUGGCCCCCAUCCAG .(((.((.(.......((((((.(((.(((((.....)))))))).(((..(((((((((((((..(..((...........))..).))))))))))))))))))))))..))).))). ( -55.40) >DroVir_CAF1 4071 120 - 1 UUAAUUGAGAAAUUUUGCCGGGCGACGCAACUAGUGAAAUUGAUUGAAGGGACCGUCGAGAACUUUCUGCGCAACUACAGGCGCACGCGCAAACCGGAGGAGGCGCUGGCGCCCCUGCGU .................((((....(((.......((((.(..((((.(....).))))..).))))(((((........))))).)))....))))(((.((((....))))))).... ( -35.40) >DroSim_CAF1 3487 120 - 1 UUAGUGGAGCACUAUUGCCGGCAGACGCAGCUGCUCAAGCUGGUCCAGGGUUCCUCUGGCCGCUUUGUGGCGAACUAUAGGAGCUCCCGGCGGCCAGAGGACCUGCUGGCCCCCAUCCAG ....((((........((((((.(((.(((((.....))))))))..((((.((((((((((((..(..((...(....)..))..).)))))))))))))))))))))).....)))). ( -60.32) >DroEre_CAF1 4731 120 - 1 UUGGUGGAGAACUUUUGCCGGCAGACGCAGCUGCUCAAGCUGGUCCAGGGCUCCUCUGGCCGCUUUGUUGCGAACUAUAGGAGCUCCCGGCGGCCAGAGGACCUGCUGGCGCCCAUCCAG .((((((.(......(((((((.(((.(((((.....))))))))..(((.(((((((((((((..(((.(........).)))....))))))))))))))))))))))))))).))). ( -61.30) >DroYak_CAF1 5176 120 - 1 CUGGUGGAGCACUUUUGCCGGCAGACGCAGUUGCUCAAGCUGGUCCAGGGCUCCUCUGGCCGCUUUGUGGCGAACUAUAGGAGCAGCCGACGGCCAGAGGACCUGCUGGCCCCCAUCCAG (((((((.(.......((((((.(((.(((((.....))))))))..(((.(((((((((((.....((((...((.....))..)))).))))))))))))))))))))).))).)))) ( -56.81) >DroAna_CAF1 5830 120 - 1 CUGGUGGAGAAGUAUUGCCGGGAGACCCAGCUGCUGAAGCUGAUCGAGGGCUCUGCUGGUAAGUUUGUGGAGAACUACAGGACUCACCGUCGUCCCGAGGACCUGCUGGCACCCGUCCAG (((((((.(......((((((.((.(((((((.....))))).(((.((((...((.(((.(((((((((....))))).)))).))))).)))))))))..)).)))))))))).)))) ( -44.30) >consensus UUGGUGGAGAACUUUUGCCGGCAGACGCAGCUGCUCAAGCUGGUCCAGGGCUCCUCUGGCCGCUUUGUGGCGAACUAUAGGAGCUCCCGGCGGCCAGAGGACCUGCUGGCCCCCAUCCAG ....((((........((((((((...(((((.....))))).........(((((((((((((.(((((....)))))((.....))))))))))))))).)))))))).....)))). (-33.99 = -37.02 + 3.03)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 12:23:42 2006