
| Sequence ID | X_DroMel_CAF1 |
|---|---|
| Location | 16,809,577 – 16,809,707 |
| Length | 130 |
| Max. P | 0.998894 |

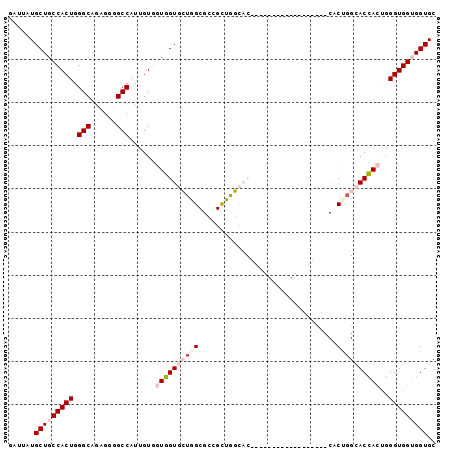
| Location | 16,809,577 – 16,809,667 |
|---|---|
| Length | 90 |
| Sequences | 6 |
| Columns | 99 |
| Reading direction | forward |
| Mean pairwise identity | 71.35 |
| Mean single sequence MFE | -41.08 |
| Consensus MFE | -24.33 |
| Energy contribution | -25.63 |
| Covariance contribution | 1.31 |
| Combinations/Pair | 1.09 |
| Mean z-score | -2.81 |
| Structure conservation index | 0.59 |
| SVM decision value | 2.87 |
| SVM RNA-class probability | 0.997501 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
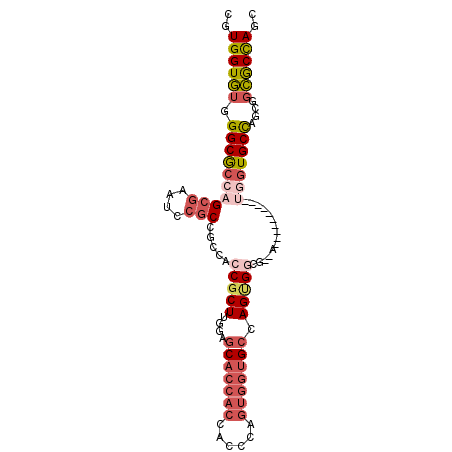
>X_DroMel_CAF1 16809577 90 + 22224390 GAUUAUGCUGCCACUGGGCAAAGGGGCCAUUGUCGUGGUGCUAGCGCCGCUGGCACCAC---------UUGCGCCACUGGCACCACUGGGUGGUGGUGC .........((((.(((((((...(((....)))(((((((((((...)))))))))))---------)))).))).))))((((((....)))))).. ( -44.00) >DroPse_CAF1 4199 71 + 1 AAUGAUGCUGCCACUGGGCAGCGGCGCCAUGGUGCUUGUUGUGGC--UGCUGCU---------------------GCAG--ACGA--UGGUGGAGGUG- .......(..(((.((.(((((((((((((..(....)..)))))--.))))))---------------------))..--.)).--)))..).....- ( -33.30) >DroSec_CAF1 5952 90 + 1 GAUUAUGCUGCCACUGGGCAAAGGGGCCAUUGUCGUGGUGCUGGCGCCGCUGGCACCAU---------UUGCACCACUUGCACCACUGGGUGGUGGUGC (((.(((..(((.((......)).)))))).)))((((((((((.(((...))).))).---------..)))))))..((((((((....)))))))) ( -41.00) >DroEre_CAF1 3055 81 + 1 GAUUAUGCUGCCACUGGGCAGCGGGGCCAUUGUGGUGGUGCUGGCGCCGCUGGCAC------------------CGCUGGCACCACUGGGUGGUGGUGC .....(((((((....)))))))..........((((((((..((...))..))))------------------)))).((((((((....)))))))) ( -43.90) >DroYak_CAF1 2855 99 + 1 GAUUAUGCUGCCACUGGGCAGAGGGGCCAUUGUGGUGGUGCUGGCGCCGUUAGCACCACUGCCACCAUUGCCACCACUGGCACCACUGGGUGGUGGUGC ......((..((((..(((((.(((((......((((((((((((...))))))))))))))).)).))))).(((.((....)).)))))))..)).. ( -48.80) >DroPer_CAF1 7606 71 + 1 AAUGAUGCUGCCACUGGGCAGCGGCGCCAUGGUGCUUGUUGUGGC--UGCUGCU---------------------GCAG--ACGA--CGGUGGAGGUG- ......(((.((((((.(((((((((((((..(....)..)))))--.))))))---------------------))..--....--)))))).))).- ( -35.50) >consensus GAUUAUGCUGCCACUGGGCAGAGGGGCCAUUGUGGUGGUGCUGGCGCCGCUGGCAC__________________CACUGGCACCACUGGGUGGUGGUGC ......(((((((((.(((......)))......((((((((((................................))))))))))..))))))))).. (-24.33 = -25.63 + 1.31)


| Location | 16,809,577 – 16,809,667 |
|---|---|
| Length | 90 |
| Sequences | 6 |
| Columns | 99 |
| Reading direction | reverse |
| Mean pairwise identity | 71.35 |
| Mean single sequence MFE | -33.62 |
| Consensus MFE | -15.94 |
| Energy contribution | -17.67 |
| Covariance contribution | 1.72 |
| Combinations/Pair | 1.11 |
| Mean z-score | -2.63 |
| Structure conservation index | 0.47 |
| SVM decision value | 2.61 |
| SVM RNA-class probability | 0.995767 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 16809577 90 - 22224390 GCACCACCACCCAGUGGUGCCAGUGGCGCAA---------GUGGUGCCAGCGGCGCUAGCACCACGACAAUGGCCCCUUUGCCCAGUGGCAGCAUAAUC (((((((......)))))((((.(((.((((---------(((((((.(((...))).)))))))......(....).))))))).)))).))...... ( -40.80) >DroPse_CAF1 4199 71 - 1 -CACCUCCACCA--UCGU--CUGC---------------------AGCAGCA--GCCACAACAAGCACCAUGGCGCCGCUGCCCAGUGGCAGCAUCAUU -........(((--(.(.--..((---------------------(((.((.--((((............)))))).))))).).)))).......... ( -18.70) >DroSec_CAF1 5952 90 - 1 GCACCACCACCCAGUGGUGCAAGUGGUGCAA---------AUGGUGCCAGCGGCGCCAGCACCACGACAAUGGCCCCUUUGCCCAGUGGCAGCAUAAUC (((((((......)))))))..(((((((..---------.(((((((...)))))))))))))).............(((((....)))))....... ( -41.20) >DroEre_CAF1 3055 81 - 1 GCACCACCACCCAGUGGUGCCAGCG------------------GUGCCAGCGGCGCCAGCACCACCACAAUGGCCCCGCUGCCCAGUGGCAGCAUAAUC (((((((......)))))))..(((------------------(((((...)))))).)).(((......)))....((((((....))))))...... ( -36.30) >DroYak_CAF1 2855 99 - 1 GCACCACCACCCAGUGGUGCCAGUGGUGGCAAUGGUGGCAGUGGUGCUAACGGCGCCAGCACCACCACAAUGGCCCCUCUGCCCAGUGGCAGCAUAAUC (((((((......)))))))....((.(((..((((((...((((((.....))))))...)))))).....))))).(((((....)))))....... ( -44.90) >DroPer_CAF1 7606 71 - 1 -CACCUCCACCG--UCGU--CUGC---------------------AGCAGCA--GCCACAACAAGCACCAUGGCGCCGCUGCCCAGUGGCAGCAUCAUU -..........(--(.(.--((((---------------------....)))--)).)).....((.(....).)).((((((....))))))...... ( -19.80) >consensus GCACCACCACCCAGUGGUGCCAGCG__________________GUGCCAGCGGCGCCAGCACCACCACAAUGGCCCCGCUGCCCAGUGGCAGCAUAAUC (((((((......)))))))...............................((.((((............)))).)).(((((....)))))....... (-15.94 = -17.67 + 1.72)


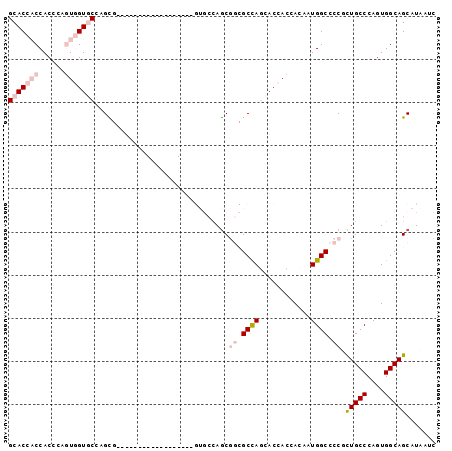
| Location | 16,809,616 – 16,809,707 |
|---|---|
| Length | 91 |
| Sequences | 5 |
| Columns | 100 |
| Reading direction | forward |
| Mean pairwise identity | 75.32 |
| Mean single sequence MFE | -48.78 |
| Consensus MFE | -31.86 |
| Energy contribution | -30.62 |
| Covariance contribution | -1.24 |
| Combinations/Pair | 1.44 |
| Mean z-score | -2.78 |
| Structure conservation index | 0.65 |
| SVM decision value | 3.27 |
| SVM RNA-class probability | 0.998894 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 16809616 91 + 22224390 GCUAGCGCCGCUGGCACCAC---------UUGCGCCACUGGCACCACUGGGUGGUGGUGCUCCAAGCGGUGGCGGCGGAUUCGCUGGCGCCCACACCACG (((((((((((((.((((.(---------(((.(.....(((((((((....)))))))))))))).)))).))))))...)))))))............ ( -50.30) >DroSec_CAF1 5991 91 + 1 GCUGGCGCCGCUGGCACCAU---------UUGCACCACUUGCACCACUGGGUGGUGGUGCUCCAAGCGGUGGCGGCGGAUUCGCUGGCGCCCACACCACG ((..(((((((((.((((.(---------(.(((((((...((((....)))))))))))...))..)))).))))))...)))..))............ ( -46.50) >DroEre_CAF1 3094 82 + 1 GCUGGCGCCGCUGGCAC------------------CGCUGGCACCACUGGGUGGUGGUGCUCCAAGCGGUGGCGGCGGAUUCGCUGGCGCCCACACCACG ((..(((((((((.(((------------------(((((((((((((....)))))))))...))))))).))))))...)))..))............ ( -52.50) >DroYak_CAF1 2894 100 + 1 GCUGGCGCCGUUAGCACCACUGCCACCAUUGCCACCACUGGCACCACUGGGUGGUGGUGCUCCAAGCGGCGGCGGCGGAUUCGCUGGCGCCCACACCACG ...(((((((((((((((((..((.....(((((....)))))......))..))))))))...))))))(.(((((....))))).))))......... ( -51.80) >DroAna_CAF1 5126 85 + 1 GGUGGUAGGACUGGCAAG---------------GACACUGCCCGGGGGAGGUGGUGGCGCCGCCAGGGGUGGUGGUGGGUUGGCCGGUGCCCAUACCACG .((((((((((((((...---------------..(((..(((....).))..))).((((((((....)))))))).....)))))).))..)))))). ( -42.80) >consensus GCUGGCGCCGCUGGCACCA__________U__CACCACUGGCACCACUGGGUGGUGGUGCUCCAAGCGGUGGCGGCGGAUUCGCUGGCGCCCACACCACG ((((((...))))))......................((((.....))))((((((..((.....))((((.(((((....))))).))))..)))))). (-31.86 = -30.62 + -1.24)

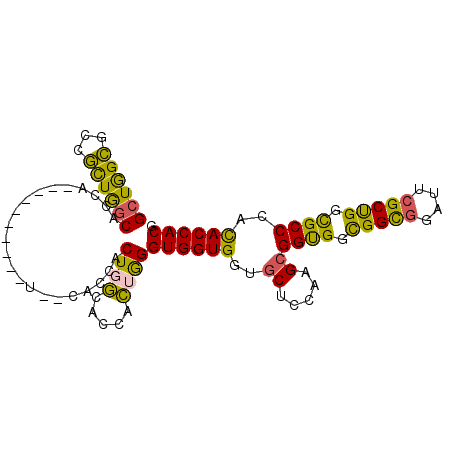
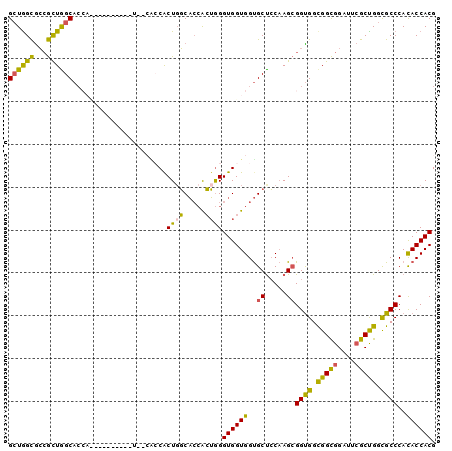
| Location | 16,809,616 – 16,809,707 |
|---|---|
| Length | 91 |
| Sequences | 5 |
| Columns | 100 |
| Reading direction | reverse |
| Mean pairwise identity | 75.32 |
| Mean single sequence MFE | -43.85 |
| Consensus MFE | -27.16 |
| Energy contribution | -29.48 |
| Covariance contribution | 2.32 |
| Combinations/Pair | 1.21 |
| Mean z-score | -2.08 |
| Structure conservation index | 0.62 |
| SVM decision value | 1.62 |
| SVM RNA-class probability | 0.968046 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 16809616 91 - 22224390 CGUGGUGUGGGCGCCAGCGAAUCCGCCGCCACCGCUUGGAGCACCACCACCCAGUGGUGCCAGUGGCGCAA---------GUGGUGCCAGCGGCGCUAGC .(((.(((.((((((((((....)))((((((......(.(((((((......)))))))).))))))...---------.))))))).))).))).... ( -44.70) >DroSec_CAF1 5991 91 - 1 CGUGGUGUGGGCGCCAGCGAAUCCGCCGCCACCGCUUGGAGCACCACCACCCAGUGGUGCAAGUGGUGCAA---------AUGGUGCCAGCGGCGCCAGC ..((((((.((((((((((....))).((.(((((((...(((((((......))))))))))))))))..---------.)))))))....)))))).. ( -47.40) >DroEre_CAF1 3094 82 - 1 CGUGGUGUGGGCGCCAGCGAAUCCGCCGCCACCGCUUGGAGCACCACCACCCAGUGGUGCCAGCG------------------GUGCCAGCGGCGCCAGC (((((((....))))).))....(((((((((((((..(.(((((((......))))))))))))------------------)))...))))))..... ( -42.80) >DroYak_CAF1 2894 100 - 1 CGUGGUGUGGGCGCCAGCGAAUCCGCCGCCGCCGCUUGGAGCACCACCACCCAGUGGUGCCAGUGGUGGCAAUGGUGGCAGUGGUGCUAACGGCGCCAGC ..((((((.(((((((.(....((((((((((((((..(.(((((((......)))))))))))))))))...)))))..))))))))....)))))).. ( -54.30) >DroAna_CAF1 5126 85 - 1 CGUGGUAUGGGCACCGGCCAACCCACCACCACCCCUGGCGGCGCCACCACCUCCCCCGGGCAGUGUC---------------CUUGCCAGUCCUACCACC .((((((.((((....((((...............))))((((....(((..((....))..)))..---------------..)))).)))))))))). ( -30.06) >consensus CGUGGUGUGGGCGCCAGCGAAUCCGCCGCCACCGCUUGGAGCACCACCACCCAGUGGUGCCAGUGGCG__A__________UGGUGCCAGCGGCGCCAGC ..((((((.((((((((((....))).....(((((....(((((((......))))))).)))))...............)))))))....)))))).. (-27.16 = -29.48 + 2.32)


Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 12:22:27 2006