
| Sequence ID | X_DroMel_CAF1 |
|---|---|
| Location | 16,754,715 – 16,755,016 |
| Length | 301 |
| Max. P | 0.991771 |

| Location | 16,754,715 – 16,754,834 |
|---|---|
| Length | 119 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 90.95 |
| Mean single sequence MFE | -30.04 |
| Consensus MFE | -24.60 |
| Energy contribution | -25.04 |
| Covariance contribution | 0.44 |
| Combinations/Pair | 1.03 |
| Mean z-score | -1.73 |
| Structure conservation index | 0.82 |
| SVM decision value | 0.95 |
| SVM RNA-class probability | 0.887695 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 16754715 119 + 22224390 CUCUUUGCCAACUAAUCCCGGCCAUAAUUUUAUGGCA-CUAUUUUAUUGUAUAACGCGCUAUAAAUGAGCUGCUGAUAGCGAUGCGAUACGAAUCAAUCGAUUUAUAGCCGCGUGCAGUU ....((((............((((((....)))))).-...............(((((((((((((..((((....))))(((..(((....))).))).)))))))).))))))))).. ( -30.80) >DroSec_CAF1 41720 119 + 1 CUCUUAGCCAACUAAUCCCGGCCAUAAUUUUAUGGCA-CUAUUUUAUUGUAUAACGCGCUAUAAAUGAGCUGCUGAUAGCGAUACGAUACGAAUCAAUCGAUUUAUAGCCGCGUGCAGUA ....................((((((....)))))).-......((((((...(((((((((((((..((((....))))....((((........)))))))))))).))))))))))) ( -31.00) >DroSim_CAF1 38883 119 + 1 CUCUUUGCCAACUAAUCCCGGUCAUAAUUUUAUGGCA-CUGUUUUAUUGUAUAACGCUCUAUAAAUGAGCUGCUGAUAGCGAUACGAUACGAAUCAAUCGAUUUAUAGCCGCGUGCAGUA ....(((((((.(((..(.(((((((....)))))).-).)..))))))....((((.((((((((..((((....))))....((((........))))))))))))..)))))))).. ( -24.80) >DroEre_CAF1 33213 114 + 1 CUCUAUGGCCCCUAAUCUCGGCCAUAAUUUUAUGGCA-CUUUUUUAUUGUAUAACGCGCUAUAAAGGAGCUGCUGAUAGC-----GAUACGAAUCAAUCGAUUUAUAGCCGCGUGCAGUA ((((((((((.........))))))).(((((((((.-(................).)))))))))))).(((((...((-----(.((..((((....))))..))..)))...))))) ( -31.39) >DroYak_CAF1 33563 119 + 1 CUCUUUGGCACCUAAUCCCGGCCAUAAUUUUAUGGCAUCAUUUUUAUUGUAUAACGCUCUAUAAAGGAGCUGGCGAUAGCUGC-UGAUACGAAUCAAUCGAUUUAUAGCCGCGUGCAGUA .......((((...((((((((((((....))))))...................(((((.....)))))))).))).((.((-((....(((((....))))).)))).)))))).... ( -32.20) >consensus CUCUUUGCCAACUAAUCCCGGCCAUAAUUUUAUGGCA_CUAUUUUAUUGUAUAACGCGCUAUAAAUGAGCUGCUGAUAGCGAU_CGAUACGAAUCAAUCGAUUUAUAGCCGCGUGCAGUA ....................((((((....))))))........((((((...((((((((((((...((((....)))).........(((.....))).))))))).))))))))))) (-24.60 = -25.04 + 0.44)


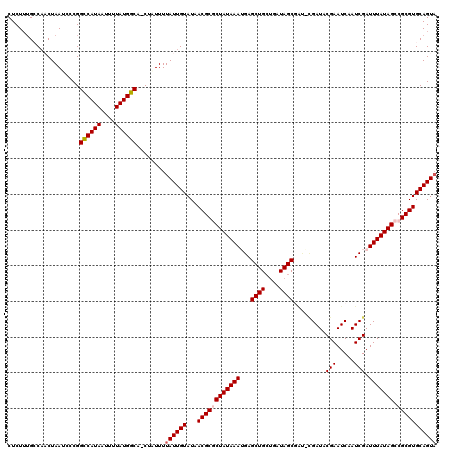
| Location | 16,754,754 – 16,754,874 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 93.99 |
| Mean single sequence MFE | -27.46 |
| Consensus MFE | -22.96 |
| Energy contribution | -23.40 |
| Covariance contribution | 0.44 |
| Combinations/Pair | 1.03 |
| Mean z-score | -1.29 |
| Structure conservation index | 0.84 |
| SVM decision value | -0.02 |
| SVM RNA-class probability | 0.525741 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 16754754 120 + 22224390 AUUUUAUUGUAUAACGCGCUAUAAAUGAGCUGCUGAUAGCGAUGCGAUACGAAUCAAUCGAUUUAUAGCCGCGUGCAGUUAUCUUUACGGCCGCCGCCAAAUCAAUCAAUUUAAAUUUGA .....(((((...(((((((((((((..((((....))))(((..(((....))).))).)))))))).)))))))))).........(((....))).......((((.......)))) ( -26.70) >DroSec_CAF1 41759 120 + 1 AUUUUAUUGUAUAACGCGCUAUAAAUGAGCUGCUGAUAGCGAUACGAUACGAAUCAAUCGAUUUAUAGCCGCGUGCAGUAAUCUUUACGGCCGCCGCCAAAUCAAUCAAUUUAAAUUUGA ...(((((((...(((((((((((((..((((....))))....((((........)))))))))))).)))))))))))).......(((....))).......((((.......)))) ( -29.30) >DroSim_CAF1 38922 120 + 1 GUUUUAUUGUAUAACGCUCUAUAAAUGAGCUGCUGAUAGCGAUACGAUACGAAUCAAUCGAUUUAUAGCCGCGUGCAGUAAUCUUUACGGCCGCCGCCAAAUCAAUCAAUUUAAAUUUGA ....((((((((...((((.......))))(((.....)))))))))))..........(((((...((.(((.((.(((.....))).))))).)).)))))..((((.......)))) ( -26.50) >DroEre_CAF1 33252 115 + 1 UUUUUAUUGUAUAACGCGCUAUAAAGGAGCUGCUGAUAGC-----GAUACGAAUCAAUCGAUUUAUAGCCGCGUGCAGUAAUCUUUACGGCCGCCGCCAAAUCAAUCAAUUUAAAUUUGA ...(((((((...((((((((((((.((((((....))))-----(((....)))..))..))))))).)))))))))))).......(((....))).......((((.......)))) ( -27.00) >DroYak_CAF1 33603 119 + 1 UUUUUAUUGUAUAACGCUCUAUAAAGGAGCUGGCGAUAGCUGC-UGAUACGAAUCAAUCGAUUUAUAGCCGCGUGCAGUAAUCUUUACGGUCGCCGCCAAAUCAAUCAAUUUAAAUUUGA (((..((((......(((((.....))))).((((((.((.((-((....(((((....))))).)))).))(((.((....)).))).))))))...........))))..)))..... ( -27.80) >consensus AUUUUAUUGUAUAACGCGCUAUAAAUGAGCUGCUGAUAGCGAU_CGAUACGAAUCAAUCGAUUUAUAGCCGCGUGCAGUAAUCUUUACGGCCGCCGCCAAAUCAAUCAAUUUAAAUUUGA ...(((((((...((((((((((((...((((....)))).........(((.....))).))))))).)))))))))))).......(((....))).......((((.......)))) (-22.96 = -23.40 + 0.44)

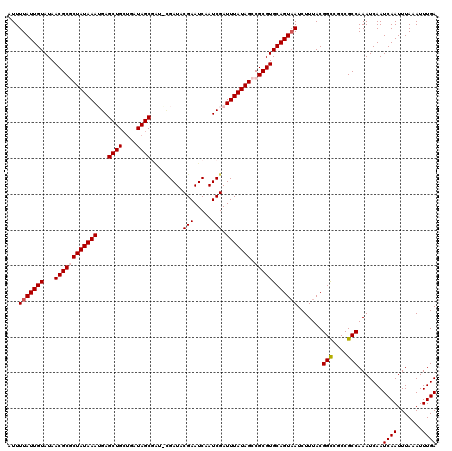
| Location | 16,754,754 – 16,754,874 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 93.99 |
| Mean single sequence MFE | -30.62 |
| Consensus MFE | -24.10 |
| Energy contribution | -25.30 |
| Covariance contribution | 1.20 |
| Combinations/Pair | 1.00 |
| Mean z-score | -1.98 |
| Structure conservation index | 0.79 |
| SVM decision value | 0.82 |
| SVM RNA-class probability | 0.858476 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 16754754 120 - 22224390 UCAAAUUUAAAUUGAUUGAUUUGGCGGCGGCCGUAAAGAUAACUGCACGCGGCUAUAAAUCGAUUGAUUCGUAUCGCAUCGCUAUCAGCAGCUCAUUUAUAGCGCGUUAUACAAUAAAAU .........(((..(((((((((((.(((((.((.......)).)).))).))))..)))))))..))).(((((((...((.....)).(((.......))))))..))))........ ( -31.70) >DroSec_CAF1 41759 120 - 1 UCAAAUUUAAAUUGAUUGAUUUGGCGGCGGCCGUAAAGAUUACUGCACGCGGCUAUAAAUCGAUUGAUUCGUAUCGUAUCGCUAUCAGCAGCUCAUUUAUAGCGCGUUAUACAAUAAAAU .........(((..(((((((((((.(((((.(((.....))).)).))).))))..)))))))..))).(((((((...((.....)).(((.......))))))..))))........ ( -30.70) >DroSim_CAF1 38922 120 - 1 UCAAAUUUAAAUUGAUUGAUUUGGCGGCGGCCGUAAAGAUUACUGCACGCGGCUAUAAAUCGAUUGAUUCGUAUCGUAUCGCUAUCAGCAGCUCAUUUAUAGAGCGUUAUACAAUAAAAC .........(((..(((((((((((.(((((.(((.....))).)).))).))))..)))))))..)))......((((.((.....)).((((.......))))...))))........ ( -32.30) >DroEre_CAF1 33252 115 - 1 UCAAAUUUAAAUUGAUUGAUUUGGCGGCGGCCGUAAAGAUUACUGCACGCGGCUAUAAAUCGAUUGAUUCGUAUC-----GCUAUCAGCAGCUCCUUUAUAGCGCGUUAUACAAUAAAAA .........(((..(((((((((((.(((((.(((.....))).)).))).))))..)))))))..))).(((((-----(((((.((......))..))))))....))))........ ( -30.10) >DroYak_CAF1 33603 119 - 1 UCAAAUUUAAAUUGAUUGAUUUGGCGGCGACCGUAAAGAUUACUGCACGCGGCUAUAAAUCGAUUGAUUCGUAUCA-GCAGCUAUCGCCAGCUCCUUUAUAGAGCGUUAUACAAUAAAAA .........(((..(((((((((((.(((.(.(((.....))).)..))).))))..)))))))..))).((((.(-((.((....))..((((.......)))))))))))........ ( -28.30) >consensus UCAAAUUUAAAUUGAUUGAUUUGGCGGCGGCCGUAAAGAUUACUGCACGCGGCUAUAAAUCGAUUGAUUCGUAUCG_AUCGCUAUCAGCAGCUCAUUUAUAGCGCGUUAUACAAUAAAAA .........(((..(((((((((((.(((((.(((.....))).)).))).))))..)))))))..))).((((.....((((((.............))))))....))))........ (-24.10 = -25.30 + 1.20)

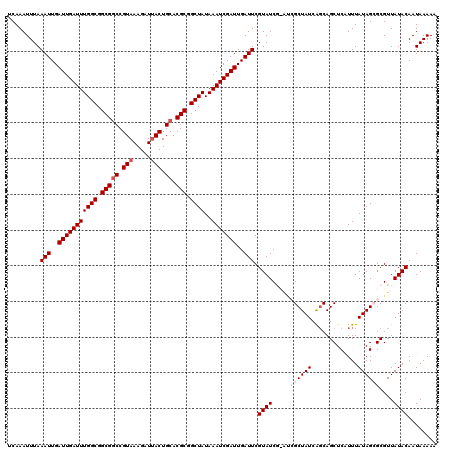
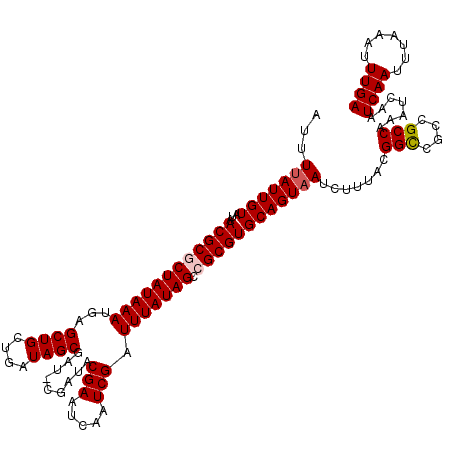
| Location | 16,754,874 – 16,754,976 |
|---|---|
| Length | 102 |
| Sequences | 4 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 79.51 |
| Mean single sequence MFE | -31.35 |
| Consensus MFE | -19.51 |
| Energy contribution | -21.20 |
| Covariance contribution | 1.69 |
| Combinations/Pair | 1.25 |
| Mean z-score | -1.97 |
| Structure conservation index | 0.62 |
| SVM decision value | 0.57 |
| SVM RNA-class probability | 0.786971 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 16754874 102 - 22224390 CAUGCUCGUCAUAUAUCAUAUAGUAUAUACAUAUAUAUAUAUUACCUGUGCG------UGCGUGUAUG----UGUGUGUGCGGGA--------UGGCAGAAAUCAGAAAUCACGGCUUAA ...(((.(((((........(((((((((......)))))))))((((..((------..((......----))..))..)))))--------)))).(....).........))).... ( -24.60) >DroSec_CAF1 41879 112 - 1 CAUGCUCGCCAUAUAUCAUAUAGUAUAUACAUAUA----UAUUACCUGUGUGCACCUGUGCGCACGUG----UGUGUGUGCGGGAUGGUUGGAUGGCAGAAAUCAGAAAUCACGGCUUAA ...(((.(((((..(((((...((((((((((((.----........(((((((....))))))))))----)))))))))...)))))...))))).(....).........))).... ( -36.70) >DroSim_CAF1 39042 112 - 1 CAUGCUCGCCAUAUAUCAUAUAGUAUAUACAUAUA----UAUUAUCUGUGUGCACCUGUGCGCACGUG----UGUGUGUGCGGGAUGGUUGGAUGGCAGAAAUCAGAAAUCACGGCUUAA (((.(((((((((((.((((((((((((....)))----))))))..(((((((....))))))))))----)))))).))))))))(((((((..(........)..))).)))).... ( -36.70) >DroEre_CAF1 33367 97 - 1 CAUGCUCGCCAUAUAUCAUAUAGUAUAUACAUAUU--------ACCCGUGUGCACCUGUGUGUCUGUGUGUCUGUGUGUGCGGGA--------UGGCAGA-------AAUCACGGCUUAA (((.(((((((((((.(((((((.(((((((...(--------((....)))....))))))))))))))..)))))).))))))--------)).....-------............. ( -27.40) >consensus CAUGCUCGCCAUAUAUCAUAUAGUAUAUACAUAUA____UAUUACCUGUGUGCACCUGUGCGCACGUG____UGUGUGUGCGGGA________UGGCAGAAAUCAGAAAUCACGGCUUAA ...(((.(((((..(((((...(((((((((...............((((((((....))))))))......)))))))))...)))))...))))).((.........))..))).... (-19.51 = -21.20 + 1.69)



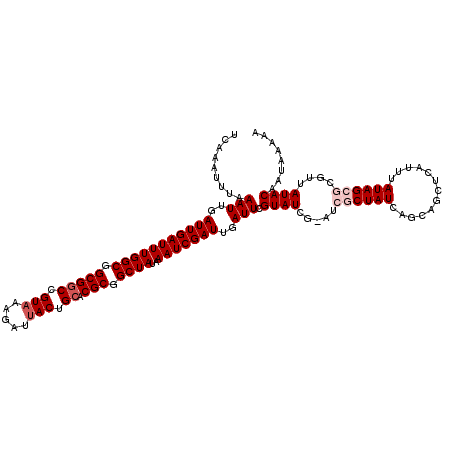
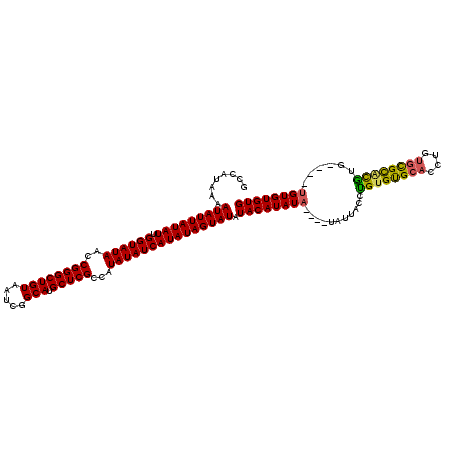
| Location | 16,754,906 – 16,755,016 |
|---|---|
| Length | 110 |
| Sequences | 4 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 87.36 |
| Mean single sequence MFE | -34.33 |
| Consensus MFE | -27.85 |
| Energy contribution | -27.48 |
| Covariance contribution | -0.37 |
| Combinations/Pair | 1.18 |
| Mean z-score | -3.19 |
| Structure conservation index | 0.81 |
| SVM decision value | 2.29 |
| SVM RNA-class probability | 0.991771 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 16754906 110 - 22224390 GCCAUAAAAUAUUAUAUUGGUAUAACCGGGCUGUAAUCGGCAUGCUCGUCAUAUAUCAUAUAGUAUAUACAUAUAUAUAUAUUACCUGUGCG------UGCGUGUAUG----UGUGUGUG ........(((((((((.((((((..((((((((.....))).)))))...))))))))))))))).((((((((((((((((((......)------)).)))))))----)))))))) ( -32.80) >DroSec_CAF1 41919 112 - 1 GCCAUAAAAUAUUAUAUUGGUAUAACCGGGCUGUAAUCGGCAUGCUCGCCAUAUAUCAUAUAGUAUAUACAUAUA----UAUUACCUGUGUGCACCUGUGCGCACGUG----UGUGUGUG ........(((((((((.((((((..((((((((.....))).)))))...))))))))))))))).((((((((----(((.....(((((((....))))))))))----)))))))) ( -37.70) >DroSim_CAF1 39082 112 - 1 GCCAUAAAAUAUUAUAUUGGUAUAACCGGGCUGUAAUCGGCAUGCUCGCCAUAUAUCAUAUAGUAUAUACAUAUA----UAUUAUCUGUGUGCACCUGUGCGCACGUG----UGUGUGUG ........(((((((((.((((((..((((((((.....))).)))))...))))))))))))))).((((((((----(((.....(((((((....))))))))))----)))))))) ( -37.70) >DroEre_CAF1 33392 112 - 1 GCCAUAAAAUAUUAUAUUGGUAUAACCGGGCUGUAAUCGGCAUGCUCGCCAUAUAUCAUAUAGUAUAUACAUAUU--------ACCCGUGUGCACCUGUGUGUCUGUGUGUCUGUGUGUG ........(((((((((.((((((..((((((((.....))).)))))...)))))))))))))))(((((((.(--------((.((.(..((....))..).)).)))..))))))). ( -29.10) >consensus GCCAUAAAAUAUUAUAUUGGUAUAACCGGGCUGUAAUCGGCAUGCUCGCCAUAUAUCAUAUAGUAUAUACAUAUA____UAUUACCUGUGUGCACCUGUGCGCACGUG____UGUGUGUG ........(((((((((.((((((..((((((((.....))).)))))...))))))))))))))).((((((((...........((((((((....))))))))......)))))))) (-27.85 = -27.48 + -0.37)

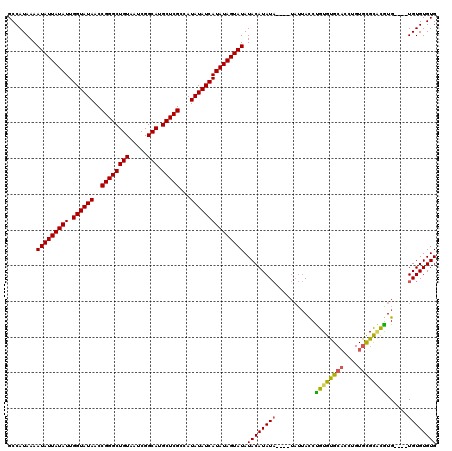
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 12:21:21 2006