| Sequence ID | X_DroMel_CAF1 |
|---|---|
| Location | 1,870,939 – 1,871,038 |
| Length | 99 |
| Max. P | 0.622172 |

| Location | 1,870,939 – 1,871,038 |
|---|---|
| Length | 99 |
| Sequences | 6 |
| Columns | 112 |
| Reading direction | forward |
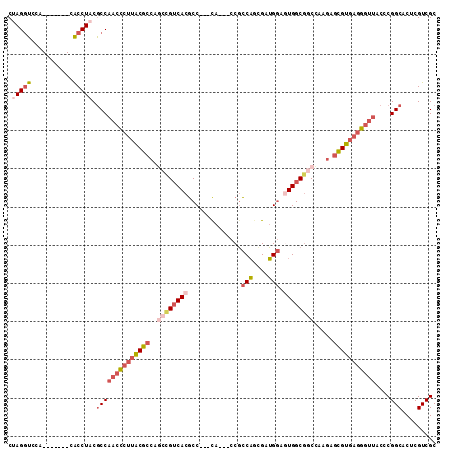
| Mean pairwise identity | 81.61 |
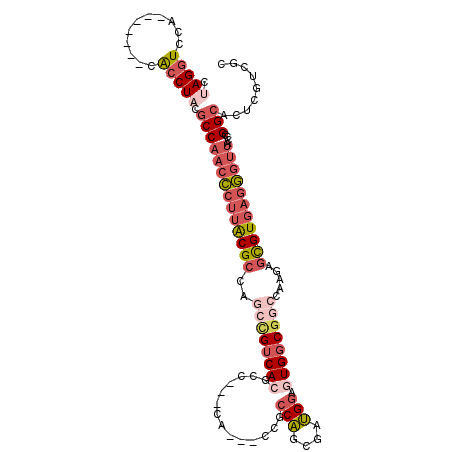
| Mean single sequence MFE | -40.82 |
| Consensus MFE | -26.12 |
| Energy contribution | -29.01 |
| Covariance contribution | 2.89 |
| Combinations/Pair | 1.20 |
| Mean z-score | -1.76 |
| Structure conservation index | 0.64 |
| SVM decision value | 0.18 |
| SVM RNA-class probability | 0.622172 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
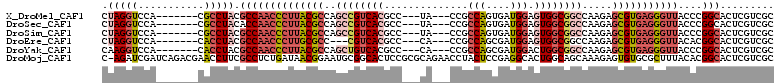
>X_DroMel_CAF1 1870939 99 + 22224390 CUAGGUCCA-------CGCCUACGCCAACCCUUACGCCAGCCGUCACGCC---UA---CCGCCAGUGAUGGAGUGGCGGCCAAGAGCGUGAGGGUUACCCGGCACUCGUCGC .(((((...-------.))))).(((((((((((((((.((((((((...---..---...(((....))).))))))))...).)))))))))))....)))......... ( -43.90) >DroSec_CAF1 8264 99 + 1 CUAGGUCCA-------CGCCUACACCAACCCUUACGCCAGCCGUCACGCC---UA---CCGCCAGUGAUGGAGUGGCGGCCAAGAGCGUGAGGGUUACCCGGCACUCGUCGC .(((((...-------.)))))....((((((((((((.((((((((...---..---...(((....))).))))))))...).)))))))))))...((((....)))). ( -42.80) >DroSim_CAF1 3512 99 + 1 CUAGGUCCA-------CGCCUACGCCAACCCUUACGCCAGCCGUCACGCC---UA---CCGCCAGUGAUGGAGUGGCGGCCAAGAGCGUGAGGGUUACCCGGCACUCGUCGC .(((((...-------.))))).(((((((((((((((.((((((((...---..---...(((....))).))))))))...).)))))))))))....)))......... ( -43.90) >DroEre_CAF1 6884 96 + 1 CUAGGUCCA-------CACCUACGCCAACCCUUGCGCC---CGUCACGCC---CA---CCGCCAGCGAUGGAGUGGCGGCCAAGAGCGUGAGGGUUACACGGCACUCGUCGC .(((((...-------.))))).(((((((((..((((---((((((..(---((---.((....)).))).)))))))......)))..))))))....)))......... ( -42.10) >DroYak_CAF1 8842 99 + 1 CAAGGUCCA-------CACCUACGCCAACCCUUACGCCAGCUGUCACGCC---CA---CCGCCAGCGAUGGACUGGCGGCCAAGAGCGUGAGGGUUACCCGGCACUCGUCGC ..((((...-------.))))..(((((((((((((((.(((((((.(.(---((---.((....)).))).))))))))...).)))))))))))....)))......... ( -40.50) >DroMoj_CAF1 430 111 + 1 C-AGAUCGAUCAGACGAACCUUCGCCUCUGAUAACGGAAUGCGGCACUCCGCGCAGAACCUACUCCGAGGCACUGGCAGCAAAGAGUGUGCGCUUUACACGGCACUCGUCGC .-...((((((((((((....)))..))))))..)))...(((((.....((.(((..(((......)))..)))))......((((((.((.......))))))))))))) ( -31.70) >consensus CUAGGUCCA_______CACCUACGCCAACCCUUACGCCAGCCGUCACGCC___CA___CCGCCAGCGAUGGAGUGGCGGCCAAGAGCGUGAGGGUUACCCGGCACUCGUCGC .(((((...........))))).((((((((((((((..((((((((..............(((....))).)))))))).....)))))))))))....)))......... (-26.12 = -29.01 + 2.89)
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 09:50:50 2006