
| Sequence ID | X_DroMel_CAF1 |
|---|---|
| Location | 16,567,438 – 16,567,535 |
| Length | 97 |
| Max. P | 0.665441 |

| Location | 16,567,438 – 16,567,535 |
|---|---|
| Length | 97 |
| Sequences | 6 |
| Columns | 108 |
| Reading direction | forward |
| Mean pairwise identity | 71.66 |
| Mean single sequence MFE | -24.62 |
| Consensus MFE | -9.28 |
| Energy contribution | -10.84 |
| Covariance contribution | 1.56 |
| Combinations/Pair | 1.06 |
| Mean z-score | -1.93 |
| Structure conservation index | 0.38 |
| SVM decision value | 0.28 |
| SVM RNA-class probability | 0.665441 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
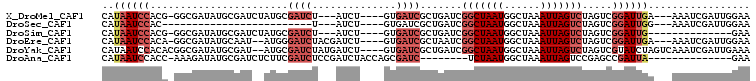
>X_DroMel_CAF1 16567438 97 + 22224390 CAUAAUCCACG-GGCGAUAUGCGAUCUAUGCGAUCU---AUCU----GUGAUCGCUGAUCGGCUAAUGGCUAAAUUAGUCUAGUCGGAUUGA---AAAUCGAUUGGAA .....((((..-..((((...(((((...((((((.---....----..))))))....((((((..(((((...)))))))))))))))).---..))))..)))). ( -31.00) >DroSec_CAF1 7224 73 + 1 CAUAAUCCAC-------------------------U---AUCU----GUGAUCGCUGAUCGGCUAAUGGCUAAAUUAGUCUAGUCGGAUUGG---AAAUCGAUUGGAA .....((((.-------------------------.---(((.----..(((..(..((((((((..(((((...)))))))))).)))..)---..)))))))))). ( -19.00) >DroSim_CAF1 7609 86 + 1 CAUAAUCCACG-GGCGAUAUGCGAUCUAUGCGAUCU---AUCU----GUGAUCGCUGAUCGGCUAAUGGCUAAAUUAGUCUAGUCGGAUUG--------------GAA .....(((((.-(((.......((((...((((((.---....----..)))))).))))(((((((......)))))))..))).)..))--------------)). ( -29.10) >DroEre_CAF1 7310 98 + 1 CAUAAUCCACA-GGCGAUAUGCAAU--AUGGGAUCUACGAUCU----GUGAUCGCUAAUCGGCUAAUGGCUAAAUUAGUCUAGUCGGAUUGA---AAAUCGAUUGGAA .....((((..-..((((...((((--.(((((((.((.....----)))))).))).(((((((..(((((...)))))))))))))))).---..))))..)))). ( -25.10) >DroYak_CAF1 8195 102 + 1 CAUAAUCCACACGGCGAUAUGCGAU--AUGCGAUCUAUGAUCU----GUGAUCGCUGAUCGGCUAAUGGCUAAAUUAGUCUAGUCGUAUCUAGUCAAAUCGAUUGAAA ..(((((.....(((...(((((((--....((((..(((((.----..)))))..))))(((((((......)))))))..)))))))...))).....)))))... ( -28.10) >DroAna_CAF1 13308 85 + 1 CAUAAUCCACC-AAAGAUAUGCGAUCUCUUCGAUCUCCGAUCUACCAGCGAUC--------UCUAAUGGCUAAAUUAGUCCGAGCCGAUUA--------------GAA ...........-...(((.(((.......(((.....))).......))))))--------((((((((((...........)))).))))--------------)). ( -15.44) >consensus CAUAAUCCACA_GGCGAUAUGCGAU__AUGCGAUCU___AUCU____GUGAUCGCUGAUCGGCUAAUGGCUAAAUUAGUCUAGUCGGAUUGA___AAAUCGAUUGGAA ..((((((.......................((((..............)))).......(((((((......))))))).....))))))................. ( -9.28 = -10.84 + 1.56)


Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 12:19:03 2006