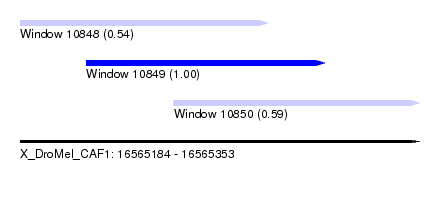
| Sequence ID | X_DroMel_CAF1 |
|---|---|
| Location | 16,565,184 – 16,565,353 |
| Length | 169 |
| Max. P | 0.997236 |

| Location | 16,565,184 – 16,565,289 |
|---|---|
| Length | 105 |
| Sequences | 6 |
| Columns | 109 |
| Reading direction | forward |
| Mean pairwise identity | 82.37 |
| Mean single sequence MFE | -44.88 |
| Consensus MFE | -37.44 |
| Energy contribution | -37.53 |
| Covariance contribution | 0.09 |
| Combinations/Pair | 1.27 |
| Mean z-score | -1.29 |
| Structure conservation index | 0.83 |
| SVM decision value | 0.01 |
| SVM RNA-class probability | 0.539848 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 16565184 105 + 22224390 -CU---UAGGUUUUGGCCGUUCUGGCUGUGAUUGGCGGUUCCCUGGCCGCUCCUCGUCCGGAUGUCAGCCAUCUGGCGGGAUACAGCUACCAGGCUGGUGGUUACAAUG -..---(((.(.(..(((....((((((((...((((((......)))))).((((.(((((((.....))))))))))).))))))))...)))..).).)))..... ( -42.10) >DroPse_CAF1 6060 106 + 1 UGAUUGCAGGUCUUGGCCUGCCUGGCGGUUAUCGGCGGUUCGCUGGCCGCUCCCCGUCCGGACGUGUCCCAUCUG---GGCUACCGCUACCAGGCAGGUGGCUACAACG ........((((...((((((((((((((....((((((......))))))....(((((((.(.....).))))---))).)))))...)))))))))))))...... ( -44.90) >DroEre_CAF1 5121 105 + 1 -UU---AAGGCUUUGGCCGUUCUGGCUGUGAUUGGCGGUUCCCUGGCCGCUCCUCGUCCGGAUGUCAGCCAUCUGGCGGGAUACAGCUAUCAGGCUGGUGGCUACAACG -..---..(((((..(((....((((((((...((((((......)))))).((((.(((((((.....))))))))))).))))))))...)))..).))))...... ( -46.10) >DroYak_CAF1 5927 105 + 1 -CU---UAGGUUUUGGCCGUUCUGGCUGUGAUUGGCGGUUCUCUGGCCGCUCCUCGUCCGGAUGUCAGCCAUCUGGCGGGCUACAGCUACCAGGCUGGUGGCUACAACG -..---...(((.(((((.....(((((((...((((((......)))))).((((.(((((((.....))))))))))).)))))))(((.....)))))))).))). ( -44.00) >DroAna_CAF1 10308 106 + 1 UGA---UAGGUUUUGGCCUGUCUGGCUGUGAUCGGCGGCGCACUGGCUGCCCCCCGUCCGGAUGUUAGCCACUUGGCGGGAUACAGCUACCAGGCUGGCGGCUACAACG ((.---(((.(.(..(((((..((((((((...((((((......)))))).((((((.((.((.....)))).)))))).)))))))).)))))..).).)))))... ( -47.30) >DroPer_CAF1 6325 106 + 1 UGAUUGCAGGUCUUGGCCUGCCUGGCGGUUAUCGGCGGUUCGCUGGCCGCUCCCCGUCCGGACGUGUCCCAUCUG---GGCUACCGCUACCAGGCAGGUGGCUACAACG ........((((...((((((((((((((....((((((......))))))....(((((((.(.....).))))---))).)))))...)))))))))))))...... ( -44.90) >consensus _GA___UAGGUUUUGGCCGGUCUGGCUGUGAUCGGCGGUUCCCUGGCCGCUCCCCGUCCGGAUGUCAGCCAUCUGGCGGGAUACAGCUACCAGGCUGGUGGCUACAACG ........(((((..(((....((((((((...((((((......)))))).((((.(((((((.....))))))))))).))))))))...)))..).))))...... (-37.44 = -37.53 + 0.09)



| Location | 16,565,212 – 16,565,313 |
|---|---|
| Length | 101 |
| Sequences | 6 |
| Columns | 101 |
| Reading direction | forward |
| Mean pairwise identity | 92.21 |
| Mean single sequence MFE | -46.68 |
| Consensus MFE | -44.90 |
| Energy contribution | -44.48 |
| Covariance contribution | -0.41 |
| Combinations/Pair | 1.26 |
| Mean z-score | -1.84 |
| Structure conservation index | 0.96 |
| SVM decision value | 2.82 |
| SVM RNA-class probability | 0.997236 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 16565212 101 + 22224390 UGGCGGUUCCCUGGCCGCUCCUCGUCCGGAUGUCAGCCAUCUGGCGGGAUACAGCUACCAGGCUGGUGGUUACAAUGGCAACCUGGUGGCCAAUCAGGUGC ..((.(((...((((((((((.((.(((((((.....))))))))))))..(((((....)))))))))))).))).)).(((((((.....))))))).. ( -42.60) >DroSec_CAF1 5044 101 + 1 UGGCGGUUCCCUUGCCGCUCCUCGUCCGGAUGUCAGCCAUCUGGCGGGAUACAGCUACCAGGCUGGUGGCUACAACGGCAACCUGGUGGCCAACCAGGUGC .((((((......)))))).((((.(((((((.....)))))))))))....(((((((.....)))))))......(((.((((((.....))))))))) ( -47.20) >DroSim_CAF1 5456 101 + 1 UGGCGGUUCCCUGGCCGCUCCUCGUCCGGAUGUCAGCCAUCUGGCGGGAUACAGCUACCAGGCUGGUGGCUACAACGGCAACCUGGUGGCCAACCAGGUGC ..((.(((...((((((((((.((.(((((((.....))))))))))))..(((((....)))))))))))).))).)).(((((((.....))))))).. ( -48.90) >DroEre_CAF1 5149 101 + 1 UGGCGGUUCCCUGGCCGCUCCUCGUCCGGAUGUCAGCCAUCUGGCGGGAUACAGCUAUCAGGCUGGUGGCUACAACGGCAACCUGGUGGCCAACCAGGUGC ..((.(((...((((((((((.((.(((((((.....))))))))))))..(((((....)))))))))))).))).)).(((((((.....))))))).. ( -48.90) >DroYak_CAF1 5955 101 + 1 UGGCGGUUCUCUGGCCGCUCCUCGUCCGGAUGUCAGCCAUCUGGCGGGCUACAGCUACCAGGCUGGUGGCUACAACGGCAACCUGGUGGCCAACCAGGUGA ..((.(((...(((((((..((((.(((((((.....)))))))))))...(((((....)))))))))))).))).)).(((((((.....))))))).. ( -47.80) >DroAna_CAF1 10337 101 + 1 CGGCGGCGCACUGGCUGCCCCCCGUCCGGAUGUUAGCCACUUGGCGGGAUACAGCUACCAGGCUGGCGGCUACAACGGAGGCUUGGUGGCCAACCAGGUGG .((((((......))))))..((....)).......((((((((..(......(((((((((((..((.......))..))))))))))))..)))))))) ( -44.70) >consensus UGGCGGUUCCCUGGCCGCUCCUCGUCCGGAUGUCAGCCAUCUGGCGGGAUACAGCUACCAGGCUGGUGGCUACAACGGCAACCUGGUGGCCAACCAGGUGC ..((.(((...(((((((..((((.(((((((.....)))))))))))...(((((....)))))))))))).))).)).(((((((.....))))))).. (-44.90 = -44.48 + -0.41)


| Location | 16,565,249 – 16,565,353 |
|---|---|
| Length | 104 |
| Sequences | 6 |
| Columns | 104 |
| Reading direction | forward |
| Mean pairwise identity | 89.68 |
| Mean single sequence MFE | -41.27 |
| Consensus MFE | -34.86 |
| Energy contribution | -34.20 |
| Covariance contribution | -0.66 |
| Combinations/Pair | 1.26 |
| Mean z-score | -1.65 |
| Structure conservation index | 0.84 |
| SVM decision value | 0.11 |
| SVM RNA-class probability | 0.587182 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 16565249 104 + 22224390 CAUCUGGCGGGAUACAGCUACCAGGCUGGUGGUUACAAUGGCAACCUGGUGGCCAAUCAGGUGCUCAGUGCUCCCUUGACCACGGUGACCACCUCGUACCAGCC .....(((.((.(((((((....))))(((((((((..((((((((((((.....)))))))((.....)).....)).)))..)))))))))..))))).))) ( -42.80) >DroSec_CAF1 5081 104 + 1 CAUCUGGCGGGAUACAGCUACCAGGCUGGUGGCUACAACGGCAACCUGGUGGCCAACCAGGUGCUCAGUGCUCCCCUGACCACGGUGACCACCUCGUACCAGCC ...((((.((((((((((((((.....))))))).....((((.((((((.....))))))))))..))).))))........(((....))).....)))).. ( -40.60) >DroSim_CAF1 5493 104 + 1 CAUCUGGCGGGAUACAGCUACCAGGCUGGUGGCUACAACGGCAACCUGGUGGCCAACCAGGUGCUCAGUGCUCCCCUGACCACGGUGACCACCUCGUACCAGCC ...((((.((((((((((((((.....))))))).....((((.((((((.....))))))))))..))).))))........(((....))).....)))).. ( -40.60) >DroEre_CAF1 5186 104 + 1 CAUCUGGCGGGAUACAGCUAUCAGGCUGGUGGCUACAACGGCAACCUGGUGGCCAACCAGGUGCUCAGUGCUCCCCUGACCACGGUGUCCUCCUCGUAUCAGCC .....((.(((((((.....(((((.((((((((((...(....)...)))))).))))((.((.....)).))))))).....)))))))))........... ( -39.60) >DroYak_CAF1 5992 104 + 1 CAUCUGGCGGGCUACAGCUACCAGGCUGGUGGCUACAACGGCAACCUGGUGGCCAACCAGGUGAUCAGUGCUCCCCUGACCACGGUGACCACCUCGUAUCAGCC (((((((..(((((((((((((.....))))))).....(....)...))))))..)))))))....(((.((....)).)))(((....)))........... ( -39.90) >DroAna_CAF1 10374 98 + 1 CACUUGGCGGGAUACAGCUACCAGGCUGGCGGCUACAACGGAGGCUUGGUGGCCAACCAGGUGGUGAGCACCCCGG------UGGUGAGCACCUCGUACCAGCC .....(((.((.(((.(((((((((((..((.......))..))))))))))).......(.((((..((((....------.))))..)))).)))))).))) ( -44.10) >consensus CAUCUGGCGGGAUACAGCUACCAGGCUGGUGGCUACAACGGCAACCUGGUGGCCAACCAGGUGCUCAGUGCUCCCCUGACCACGGUGACCACCUCGUACCAGCC .....(((.((.(((.(((((((((.((.((.......)).)).))))))))).....(((((.(((.((............)).))).))))).))))).))) (-34.86 = -34.20 + -0.66)



Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 12:19:00 2006