| Sequence ID | X_DroMel_CAF1 |
|---|---|
| Location | 16,495,628 – 16,495,748 |
| Length | 120 |
| Max. P | 0.872341 |

| Location | 16,495,628 – 16,495,748 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 76.83 |
| Mean single sequence MFE | -48.98 |
| Consensus MFE | -26.53 |
| Energy contribution | -27.71 |
| Covariance contribution | 1.17 |
| Combinations/Pair | 1.35 |
| Mean z-score | -2.03 |
| Structure conservation index | 0.54 |
| SVM decision value | 0.88 |
| SVM RNA-class probability | 0.872341 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
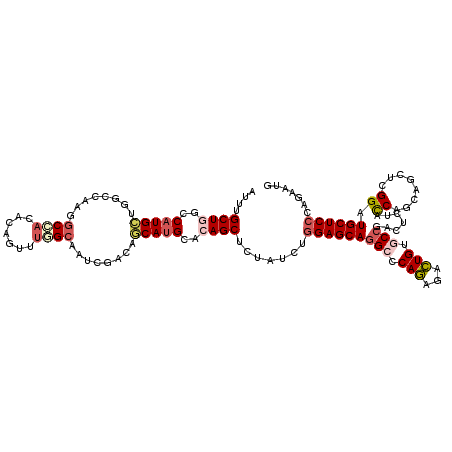
>X_DroMel_CAF1 16495628 120 + 22224390 CCUGCUGCGAGCAACCGAGGUGCUGAUAAAGGCGGCACAAUCUUUGCCCAUGCUCCAGAUAAAGCUGUGCAUGUCCACGGUUGCCAAACCGAAUGGCACGAGCCAACAUGGCCAGCGAAU ..(((((.(.(((((((..((((((.......))))))......((..(((((..(((......))).)))))..))))))))))...(((..((((....))))...))).)))))... ( -42.60) >DroVir_CAF1 5329 120 + 1 CAUACUGGGAGCAGCCCAGCUGCUGGUAGAGCCGGCACAGGCGCUGCGCCUGCUCCAAAUAGAGCUGGGCGUGCUGUGCAUUGCGAAACUCCGUCGCCUUGGCCAGCAUGGCCAGCAUGU .......((((((((.((((((((((.....)))))).....)))).).))))))).......(((((.(((((((.((...((((.......))))....))))))))).))))).... ( -59.50) >DroPse_CAF1 5767 120 + 1 CAUUCUGGGAGCAUCCGAGCUUCUGAUACAGUCGGCACAGCCUCUGGGCCUGCUCCAAAAAGAGCUCCGCAUGCAUCCGAUUGCCAAACUGUGUGGCCUUGGCCAGCAUGGCCAGCAAAU ....(((((((((((.(((((..(.....(((.(((.(((...))).))).))).....)..))))).).)))).)))....((((...(((.((((....))))))))))))))..... ( -40.80) >DroYak_CAF1 4549 120 + 1 CCUGCUGGGAGCAACCGAGGUGCUGAUAGAGGCGGCACAAUCUUUGGCCCUGCUCCAGAUAAAGCUGUGCAUGGGUACGGUUGCCAAAUCGGGUGGCACACGCCAACAUGGCCAGCGAAU ..(((((((.(((((((..((((((.......))))))........(((((((..(((......))).))).)))).))))))))...(((..((((....))))...)))))))))... ( -48.70) >DroMoj_CAF1 5634 120 + 1 CGUAUUGGGAGCAGCCCAGCUGCUGGUAGAGCCGGCACAGGCGCUGCGCCUGCUCCAGGUAGAGCUGGGCAUGCUGUGCAUUCCGGAACUCCGUUGCCUUGGCCAGCAUGGCCAGCAUAU ..(((..((((((((.((((((((((.....)))))).....)))).).)))))))..)))..(((((.(((((((.((.(..(((....)))..).....))))))))).))))).... ( -59.00) >DroPer_CAF1 3680 120 + 1 CAUUCUGGGAGCAUCCGAGCUUCUGGUACAGUCGGCACAGCCUCUGGGCCUGCUCCAAAAAGAGCUCCGCAUGCAGCCGAUUGCCAAACUGUGUUGCCUUGGCCAGCAUGGCCAGCAGAU ....(..(((((......)))))..)..((((((((...(((....)))..((((......))))..........))))))))..........((((..(((((.....))))))))).. ( -43.30) >consensus CAUACUGGGAGCAACCGAGCUGCUGAUAGAGCCGGCACAGCCUCUGCGCCUGCUCCAAAUAGAGCUGCGCAUGCAGACGAUUGCCAAACUGCGUGGCCUUGGCCAGCAUGGCCAGCAAAU .......((((((((.((((((((((.....)))))...))))).))...)))))).......((((..(((((........((((.......))))........)))))..)))).... (-26.53 = -27.71 + 1.17)


| Location | 16,495,628 – 16,495,748 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 76.83 |
| Mean single sequence MFE | -49.47 |
| Consensus MFE | -22.97 |
| Energy contribution | -23.89 |
| Covariance contribution | 0.92 |
| Combinations/Pair | 1.15 |
| Mean z-score | -1.98 |
| Structure conservation index | 0.46 |
| SVM decision value | 0.16 |
| SVM RNA-class probability | 0.611604 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 16495628 120 - 22224390 AUUCGCUGGCCAUGUUGGCUCGUGCCAUUCGGUUUGGCAACCGUGGACAUGCACAGCUUUAUCUGGAGCAUGGGCAAAGAUUGUGCCGCCUUUAUCAGCACCUCGGUUGCUCGCAGCAGG ....(((((((....((((....))))...)))..((((((((.((.(((((.(((......)))..)))))((((.......))))((........)).)).))))))))..))))... ( -45.90) >DroVir_CAF1 5329 120 - 1 ACAUGCUGGCCAUGCUGGCCAAGGCGACGGAGUUUCGCAAUGCACAGCACGCCCAGCUCUAUUUGGAGCAGGCGCAGCGCCUGUGCCGGCUCUACCAGCAGCUGGGCUGCUCCCAGUAUG .((((((((...(((((((....((((.......))))...)).))))).((((((((((...((((((.(((((((...))))))).))))))..)).)))))))).....)))))))) ( -62.50) >DroPse_CAF1 5767 120 - 1 AUUUGCUGGCCAUGCUGGCCAAGGCCACACAGUUUGGCAAUCGGAUGCAUGCGGAGCUCUUUUUGGAGCAGGCCCAGAGGCUGUGCCGACUGUAUCAGAAGCUCGGAUGCUCCCAGAAUG .....(((((((.((((((....)))....))).))))....(((.((((.(.(((((((...((.((..(((.(((...))).)))..)).))..)).))))).))))))))))).... ( -43.80) >DroYak_CAF1 4549 120 - 1 AUUCGCUGGCCAUGUUGGCGUGUGCCACCCGAUUUGGCAACCGUACCCAUGCACAGCUUUAUCUGGAGCAGGGCCAAAGAUUGUGCCGCCUCUAUCAGCACCUCGGUUGCUCCCAGCAGG ....(((((.((.(((((.(((...)))))))).))(((((((..(((.(((.(((......)))..)))))).........((((...........))))..)))))))..)))))... ( -40.80) >DroMoj_CAF1 5634 120 - 1 AUAUGCUGGCCAUGCUGGCCAAGGCAACGGAGUUCCGGAAUGCACAGCAUGCCCAGCUCUACCUGGAGCAGGCGCAGCGCCUGUGCCGGCUCUACCAGCAGCUGGGCUGCUCCCAAUACG ......(((((.....)))))..(((.(((....)))...)))..((((.((((((((((...((((((.(((((((...))))))).))))))..)).))))))))))))......... ( -58.00) >DroPer_CAF1 3680 120 - 1 AUCUGCUGGCCAUGCUGGCCAAGGCAACACAGUUUGGCAAUCGGCUGCAUGCGGAGCUCUUUUUGGAGCAGGCCCAGAGGCUGUGCCGACUGUACCAGAAGCUCGGAUGCUCCCAGAAUG .((((.(((((.....))))).((((..((((((.((((..(((((...((.((.(((((....)))))...))))..))))))))))))))).((........)).))))..))))... ( -45.80) >consensus AUUUGCUGGCCAUGCUGGCCAAGGCCACACAGUUUGGCAAUCGACAGCAUGCACAGCUCUAUCUGGAGCAGGCCCAGAGACUGUGCCGACUCUACCAGCAGCUCGGAUGCUCCCAGAAUG ....((((..(((((........((((.......))))........)))))..)))).......(((((((((.(((...))).))).......((........)).))))))....... (-22.97 = -23.89 + 0.92)


Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 12:18:24 2006