
| Sequence ID | X_DroMel_CAF1 |
|---|---|
| Location | 16,491,986 – 16,492,146 |
| Length | 160 |
| Max. P | 0.572951 |

| Location | 16,491,986 – 16,492,106 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 79.94 |
| Mean single sequence MFE | -53.63 |
| Consensus MFE | -35.06 |
| Energy contribution | -37.15 |
| Covariance contribution | 2.09 |
| Combinations/Pair | 1.27 |
| Mean z-score | -1.37 |
| Structure conservation index | 0.65 |
| SVM decision value | 0.08 |
| SVM RNA-class probability | 0.572951 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
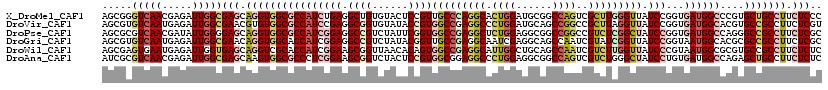
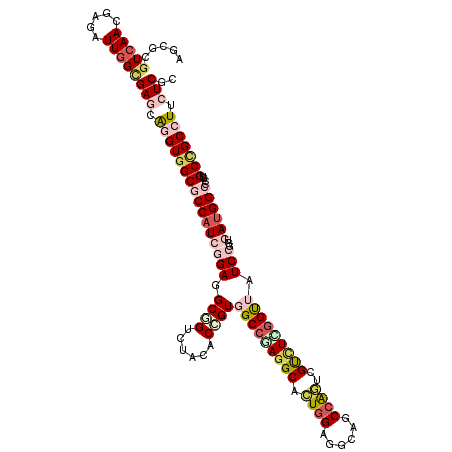
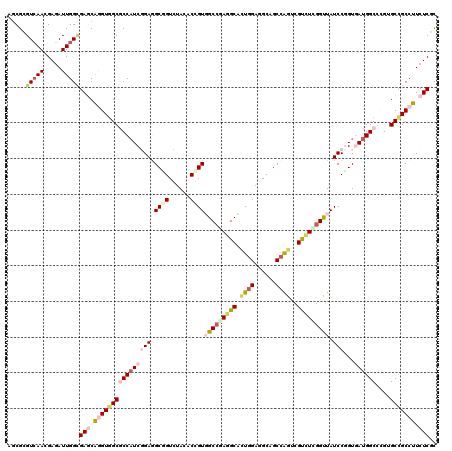
>X_DroMel_CAF1 16491986 120 + 22224390 AGCGGGUCAACGAGAUUGGCGAGCAGGUGGCGCCAUCUGAGGCUGUGUACUCCGUUGCCCAGGCACUGGAUGCGGCCAGUCGCUUGGGUUAUCCGGUGAUGGCCCGUGCUGCCUUCUCCC .((.((((.....)))).))(((.(((..((((.....(.((((((.((((..((.(((((((((((((......))))).)))))))).))..)))))))))))))))..))).))).. ( -54.40) >DroVir_CAF1 1562 120 + 1 AGCGUGUCAAUGAGAUUGGCGAACGUGUGGCGCCAUCCGAGGCGGUGUAUACCGUGGCCGAGGCCCUGGAUGCAGCCGGCCGCUUAGGUUAUCCGGUGAUGGCACGUGCCGCCUUCUCGU .........((((((..((((.((((((.(((((......((((((((((.(((.(((....))).)))))))).)).))).....((....)))))).).))))))..)))).)))))) ( -53.20) >DroPse_CAF1 1581 120 + 1 AGCGCGUCAACGAUAUUGGGGAGCAGGUGGCGCCAUCGGAGGCCGUCUAUUCGGUGGCCGAGGCUCUGGAGGCGGCCGGCCGUCUCGGCUAUCCGGUGAUGGCCAGGGCCGCCUUCUCGC .((.(.((((.....)))).).))....((.(((.((((..((((......))))..))))))).))(((((((((((((((((((((....)))).)))))))..)))))))))).... ( -63.20) >DroGri_CAF1 1581 120 + 1 AGCGUGUCAAUGAGAUUGGCGAACAGGUGGCACCAUCGGAGGCCGUCUAUACGGUUGCCGAGGCAAUCGAGGCAGCCAAUCGUAUCGGUUAUCCGGUAAUGGCACGCGCCGCCUUCUCGC .((((((((....(((((((...(((..(((..(....)..)))..))...(((((((....))))))).)...))))))).((((((....)))))).))))))))............. ( -52.10) >DroWil_CAF1 4515 120 + 1 AGCGAGUGAAUGAGAUUGGUGAGCAGGUCGCACCAUCGGAAGCGGUUAACACAGUGGCCGAGGCAUUGGCUGCAGCCAAUCGUCUUGGUUAUCCCGUAAUGGCGCGUGCCGCCUUCUCUC ...........((((..((((.(((.(.(((.....(((..(.(.....).).(((((((((((((((((....)))))).))))))))))).))).....)))).))))))).)))).. ( -48.20) >DroAna_CAF1 831 120 + 1 AUCGCGUCAACGAGAUUGGCGAGCAAGUGGCGCCCUCGGAAGCGGUCUACUCCGUGGCGGAGGCCCUGGAGGCGGCCAGUCGUCUGGGCUAUCCUGUGAUGGCCAGAGCUGCCUUCUCUC ...........((((..(((.(((...(((((((..(((..((((......))))(((....))))))..))).))))....(((((.(((((....)))))))))))))))).)))).. ( -50.70) >consensus AGCGCGUCAACGAGAUUGGCGAGCAGGUGGCGCCAUCGGAGGCGGUCUACACCGUGGCCGAGGCACUGGAGGCAGCCAGUCGUCUCGGUUAUCCGGUGAUGGCCCGUGCCGCCUUCUCGC .....(((((.....)))))(((.((((((((((((((((.((((......))))(((((((((.((((......))))..))))))))).)))...))))))....))))))).))).. (-35.06 = -37.15 + 2.09)
| Location | 16,492,026 – 16,492,146 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 82.00 |
| Mean single sequence MFE | -57.48 |
| Consensus MFE | -37.53 |
| Energy contribution | -36.07 |
| Covariance contribution | -1.47 |
| Combinations/Pair | 1.36 |
| Mean z-score | -1.57 |
| Structure conservation index | 0.65 |
| SVM decision value | 0.00 |
| SVM RNA-class probability | 0.534286 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
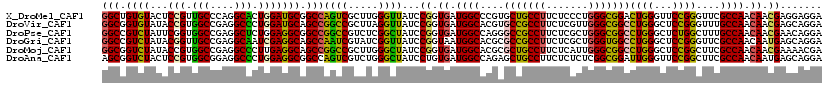
>X_DroMel_CAF1 16492026 120 + 22224390 GGCUGUGUACUCCGUUGCCCAGGCACUGGAUGCGGCCAGUCGCUUGGGUUAUCCGGUGAUGGCCCGUGCUGCCUUCUCCCUGGGCGGACUGGGUUCCGGGUUCGCCAACAACGAGGAGGA (((((((...((((.(((....))).)))))))))))....((.(((((((((....))))))))).))..(((((((....((((((((.(....).))))))))......))))))). ( -58.70) >DroVir_CAF1 1602 120 + 1 GGCGGUGUAUACCGUGGCCGAGGCCCUGGAUGCAGCCGGCCGCUUAGGUUAUCCGGUGAUGGCACGUGCCGCCUUCUCGUUGGGCGGCCUGGGCUCCGGGUUUGCCAACAACGAGCAGGA ((((((((.(((((((((((..((.......))...)))))))...((....))))))...)))).)))).(((.(((((((((((((((((...))))).)))))..))))))).))). ( -58.40) >DroPse_CAF1 1621 120 + 1 GGCCGUCUAUUCGGUGGCCGAGGCUCUGGAGGCGGCCGGCCGUCUCGGCUAUCCGGUGAUGGCCAGGGCCGCCUUCUCGCUGGGCGGCCUGGGCUCUGGCUUUGCCAACAACGAACAGGA (((((((...((((((((((((((.((((......))))..)))))))))).)))).))))))).(((((((((.......))))))))).(((.........))).............. ( -64.40) >DroGri_CAF1 1621 120 + 1 GGCCGUCUAUACGGUUGCCGAGGCAAUCGAGGCAGCCAAUCGUAUCGGUUAUCCGGUAAUGGCACGCGCCGCCUUCUCGCUGGGUGGCCUGGGCUCCGGGUUCGCCAACAAUGAGCAGGA ((((((((...(((((((....))))))))))).((((....((((((....)))))).))))..).))).(((.((((.((((((((((((...))))).)))))..)).)))).))). ( -52.50) >DroMoj_CAF1 1603 120 + 1 GGCGGUCUAUACCGUGGCCGAGGCCCUUGAGGCAGCCGGCCGCUUGGGCUAUCCGGUGAUGGCACGCGCUGCCUUCUCAUUGGGCGGCCUGGGCUCCGGCUUCGCCAACAACGAAAACGA ((((((((.....(.(((....))))...))))((((((..(((..((((..(((((((.((((.....))))...)))))))..))))..))).)))))).)))).............. ( -58.40) >DroAna_CAF1 871 120 + 1 AGCGGUCUACUCCGUGGCGGAGGCCCUGGAGGCGGCCAGUCGUCUGGGCUAUCCUGUGAUGGCCAGAGCUGCCUUCUCUCUCGGCGGAUUGGGUUCCGGCUUCGCCAACAAUGAGCAGGA .((((......)).(((((((((((..((((((((((((....)))(((((((....)))))))...)))))))))......)))(((......)))..)))))))).......)).... ( -52.50) >consensus GGCGGUCUAUACCGUGGCCGAGGCCCUGGAGGCAGCCAGCCGCUUGGGCUAUCCGGUGAUGGCACGCGCCGCCUUCUCGCUGGGCGGCCUGGGCUCCGGCUUCGCCAACAACGAGCAGGA (((.((((...(((.(((....))).))))))).)))((((.....))))...((.((.((((....(((((((.......)))))))((((...))))....)))).)).))....... (-37.53 = -36.07 + -1.47)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 12:18:21 2006