
| Sequence ID | X_DroMel_CAF1 |
|---|---|
| Location | 16,486,214 – 16,486,414 |
| Length | 200 |
| Max. P | 0.939684 |

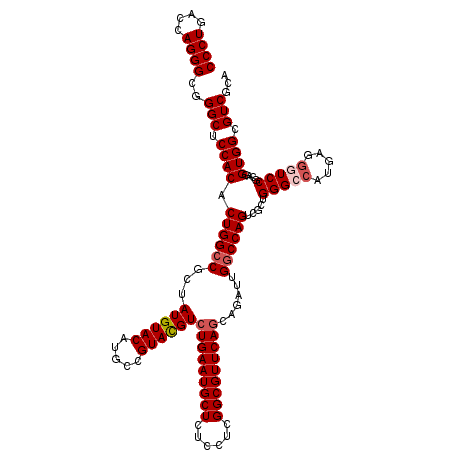
| Location | 16,486,214 – 16,486,334 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 95.67 |
| Mean single sequence MFE | -51.96 |
| Consensus MFE | -44.42 |
| Energy contribution | -45.26 |
| Covariance contribution | 0.84 |
| Combinations/Pair | 1.03 |
| Mean z-score | -1.55 |
| Structure conservation index | 0.85 |
| SVM decision value | 0.73 |
| SVM RNA-class probability | 0.833903 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 16486214 120 - 22224390 CCCUGACCAGGGCGGGCUCCACACUGGCCGCUAUGUACAUACCGUAUGUCUGAAUGCUCUCCUCGGCGUUCAACAGAUUGGCCAGCCGCUGGGCCAUGAGGGUCCGCAGGUGGCGUCGCA ((((....))))..(((.((((.(((((((...((((((((...))))).((((((((......)))))))))))...)))))))..((.(((((.....)))))))..)))).)))... ( -49.00) >DroSec_CAF1 24353 120 - 1 CCCUGACCAGGGCGGGCUCCACACUGGCCGCUAUGUACAUGCCGUACGUCUGAAUGCUCUCCUCGGCGUUCAGCAGAUUGGCCAGUCGCUGGGCCAUCAGGGUCCGCAGGUGGCGUCGCA ((((....))))..(((.(((((((((((.....((((.....))))(.(((((((((......)))))))))).....))))))).((.(((((.....)))))))..)))).)))... ( -54.30) >DroSim_CAF1 21462 120 - 1 CCCUGACCAGGGCGGGCUCCACACUGGCCGCUAUGUACAUGCCGUACGUCUGAAUGCUCUCCUCGGCGUUCAGCAGAUUGGCCAGUCGCUGGGCCAUGAGGGUCCGCAGGUGGCGUCGCA ((((....))))..(((.(((((((((((.....((((.....))))(.(((((((((......)))))))))).....))))))).((.(((((.....)))))))..)))).)))... ( -54.30) >DroEre_CAF1 15204 120 - 1 CCCUGACCAGGGCGGGCUCCACACUGGACGCUAUGUACAUGCCGUACGUCUGAAUGCUCUCCUCGGCGUUCAGCAGAUUGGCCAGCCGCUGGGCCAUGAGGCUCCUCAGGUGGCGUCUCA ......(((((((.((((.....(((......((((((.....))))))(((((((((......))))))))))))...)))).))).))))((((((((....))))..))))...... ( -46.80) >DroYak_CAF1 16370 120 - 1 CCCUGACCAGGGCGGGCUCCACACUGGCCGCUAUGUACAUGCCGUACGUCUGAAUGCUCUCCUCGGCGUUCAGCAUAUUGGCCAGUCGCUGGGCCAUGAGACUCCUCAGGUGGCGUCUCA ((((....))))..(((.((((((((((((.(((((((.....))))(.(((((((((......))))))))))))).)))))))).).)))))).((((((.((......)).)))))) ( -55.40) >consensus CCCUGACCAGGGCGGGCUCCACACUGGCCGCUAUGUACAUGCCGUACGUCUGAAUGCUCUCCUCGGCGUUCAGCAGAUUGGCCAGUCGCUGGGCCAUGAGGGUCCGCAGGUGGCGUCGCA ((((....))))..(((.((((.((((((...((((((.....))))))(((((((((......)))))))))......)))))).....(((((.....)))))....)))).)))... (-44.42 = -45.26 + 0.84)

| Location | 16,486,294 – 16,486,414 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 97.00 |
| Mean single sequence MFE | -50.39 |
| Consensus MFE | -44.08 |
| Energy contribution | -44.32 |
| Covariance contribution | 0.24 |
| Combinations/Pair | 1.03 |
| Mean z-score | -1.64 |
| Structure conservation index | 0.87 |
| SVM decision value | 1.01 |
| SVM RNA-class probability | 0.899360 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 16486294 120 + 22224390 AUGUACAUAGCGGCCAGUGUGGAGCCCGCCCUGGUCAGGGACAUUCUGGUGCACGCCAUCGAGGACGUGUACUUCCAUCGACCGCUGGAGAAGCUGGUGCAGCAGCUGGAGCAACUGUAC ..(((((((((((((((.((((...)))).)))))).((........((((((((((.....)).))))))))........))))))....(((((......)))))........))))) ( -45.19) >DroSec_CAF1 24433 120 + 1 AUGUACAUAGCGGCCAGUGUGGAGCCCGCCCUGGUCAGGGACAUUCUGGUGCACGCCAUCGAGGACGUGUACUUCCAUCGCCCGCAGGAGAAGCUGGUCCAGCAGCUGGAGCAACUGUAC ..((((...((((((((.((((...)))).)))))).(((.......((((((((((.....)).)))))))).......)))))......((.((.(((((...))))).)).)))))) ( -48.84) >DroSim_CAF1 21542 120 + 1 AUGUACAUAGCGGCCAGUGUGGAGCCCGCCCUGGUCAGGGACAUUCUGGUGCACGCCAUCGAGGACGUGUACUUCCAUCGCCCGCUGGAGAAGCUGGUCCAGCAGCUGGAGCAACUGUAC ..((((.((((((((((.((((...)))).)))))).(((.......((((((((((.....)).)))))))).......)))))))....((.((.(((((...))))).)).)))))) ( -50.64) >DroEre_CAF1 15284 120 + 1 AUGUACAUAGCGUCCAGUGUGGAGCCCGCCCUGGUCAGGGACAUUCUGGUGCACGCCAUUGAGGACGUGUAUUUCCAUCGGCCGCUGGAGAAGCUGGUCCAGCAGCUGGAGCAGCUGUAC ..((((.(((((.((...((((((...(((((....)))).)......(((((((((.....)).))))))))))))).)).)))))....(((((.(((((...))))).))))))))) ( -48.90) >DroYak_CAF1 16450 120 + 1 AUGUACAUAGCGGCCAGUGUGGAGCCCGCCCUGGUCAGGGACAUUCUGGUGCACGCCAUUGAGGACGUGUACUUCCAUCGGCCGCUGGAGAAGCUGGUCCAGCAGCUGGAGCAGCUGUAC ..((((.((((((((...(((((....(((((....)))).).....((((((((((.....)).))))))))))))).))))))))....(((((.(((((...))))).))))))))) ( -58.40) >consensus AUGUACAUAGCGGCCAGUGUGGAGCCCGCCCUGGUCAGGGACAUUCUGGUGCACGCCAUCGAGGACGUGUACUUCCAUCGCCCGCUGGAGAAGCUGGUCCAGCAGCUGGAGCAACUGUAC ..(((((..((..((((((((((....(((((....)))).).....((((((((((.....)).))))))))))))).....((((((........)))))).))))).))...))))) (-44.08 = -44.32 + 0.24)



| Location | 16,486,294 – 16,486,414 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 97.00 |
| Mean single sequence MFE | -46.60 |
| Consensus MFE | -42.92 |
| Energy contribution | -43.80 |
| Covariance contribution | 0.88 |
| Combinations/Pair | 1.05 |
| Mean z-score | -1.54 |
| Structure conservation index | 0.92 |
| SVM decision value | 1.28 |
| SVM RNA-class probability | 0.939684 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 16486294 120 - 22224390 GUACAGUUGCUCCAGCUGCUGCACCAGCUUCUCCAGCGGUCGAUGGAAGUACACGUCCUCGAUGGCGUGCACCAGAAUGUCCCUGACCAGGGCGGGCUCCACACUGGCCGCUAUGUACAU (((((........(((((......))))).....((((((((.((((.((.((((((......)))))).))..(..((.((((....))))))..)))))...)))))))).))))).. ( -42.40) >DroSec_CAF1 24433 120 - 1 GUACAGUUGCUCCAGCUGCUGGACCAGCUUCUCCUGCGGGCGAUGGAAGUACACGUCCUCGAUGGCGUGCACCAGAAUGUCCCUGACCAGGGCGGGCUCCACACUGGCCGCUAUGUACAU (((((...((.((((.((.((((...(((..(((((((((.((((...((.((((((......)))))).)).....))))))))..)))))..)))))))))))))..))..))))).. ( -45.10) >DroSim_CAF1 21542 120 - 1 GUACAGUUGCUCCAGCUGCUGGACCAGCUUCUCCAGCGGGCGAUGGAAGUACACGUCCUCGAUGGCGUGCACCAGAAUGUCCCUGACCAGGGCGGGCUCCACACUGGCCGCUAUGUACAU (((((...((.((((((((((((........))))))))..(.((((.((.((((((......)))))).))..(..((.((((....))))))..)))))).))))..))..))))).. ( -45.60) >DroEre_CAF1 15284 120 - 1 GUACAGCUGCUCCAGCUGCUGGACCAGCUUCUCCAGCGGCCGAUGGAAAUACACGUCCUCAAUGGCGUGCACCAGAAUGUCCCUGACCAGGGCGGGCUCCACACUGGACGCUAUGUACAU (((((...(((((((((((((((........))))))))).(.((((....((((((......)))))).....(..((.((((....))))))..))))))..)))).))..))))).. ( -47.60) >DroYak_CAF1 16450 120 - 1 GUACAGCUGCUCCAGCUGCUGGACCAGCUUCUCCAGCGGCCGAUGGAAGUACACGUCCUCAAUGGCGUGCACCAGAAUGUCCCUGACCAGGGCGGGCUCCACACUGGCCGCUAUGUACAU (((((((((.(((((...))))).))))......((((((((.((((.((.((((((......)))))).))..(..((.((((....))))))..)))))...)))))))).))))).. ( -52.30) >consensus GUACAGUUGCUCCAGCUGCUGGACCAGCUUCUCCAGCGGCCGAUGGAAGUACACGUCCUCGAUGGCGUGCACCAGAAUGUCCCUGACCAGGGCGGGCUCCACACUGGCCGCUAUGUACAU (((((((((.(((((...))))).))))......((((((((.((((.((.((((((......)))))).))..(..((.((((....))))))..)))))...)))))))).))))).. (-42.92 = -43.80 + 0.88)


Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 12:18:13 2006