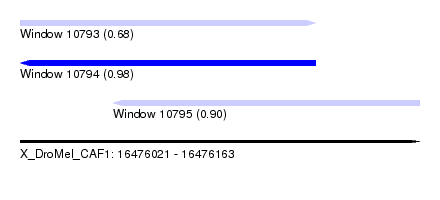
| Sequence ID | X_DroMel_CAF1 |
|---|---|
| Location | 16,476,021 – 16,476,163 |
| Length | 142 |
| Max. P | 0.981207 |

| Location | 16,476,021 – 16,476,126 |
|---|---|
| Length | 105 |
| Sequences | 6 |
| Columns | 118 |
| Reading direction | forward |
| Mean pairwise identity | 75.24 |
| Mean single sequence MFE | -38.78 |
| Consensus MFE | -14.47 |
| Energy contribution | -16.34 |
| Covariance contribution | 1.87 |
| Combinations/Pair | 1.25 |
| Mean z-score | -2.19 |
| Structure conservation index | 0.37 |
| SVM decision value | 0.30 |
| SVM RNA-class probability | 0.677348 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
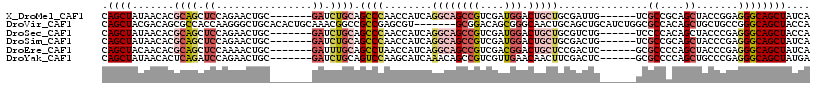
>X_DroMel_CAF1 16476021 105 + 22224390 CAGCUAUAACACGCAGCUCCAGAACUGC-------GAUCUGCAGCCCAACCAUCAGGCAGCCGUCGAUGGACUGCUGCGAUUG------UCGCCGCAGCUACCGGAGGGCAGCUAUCA .((((.......((((.((((....)).-------)).)))).((((..(((((.(((....))))))))...((((((....------....)))))).......)))))))).... ( -40.80) >DroVir_CAF1 24890 111 + 1 CAGCUACGACAGCGCCACCAAGGGCUGCACACUGCAAACGGCCGCCGAGCGU-------GCGGACAGCGGGCAACUGCAGCUGCAUCUGGCGCCACAGCUGCUGCCGGGCAGCUACCA ..((.....((((.((.....))))))......))....(((.(((.((.((-------((((...((((....))))..))))))))))))))..(((((((....))))))).... ( -47.10) >DroSec_CAF1 14231 105 + 1 CAGCUAUAACACGCAGCUCCAGAACUGC-------GAUCUGCAGCCCAACCAUCAGGCAGCCGUCGAUGGACUGCUGCGUCUG------UCCCCACAGCUACCCGAGGGCAGCUACCA .((((.......((((.((((....)).-------)).)))).((((.........(((((.(((....))).)))))(.(((------(....))))).......)))))))).... ( -36.20) >DroSim_CAF1 11414 105 + 1 CAGCUAUAACACGCAGCUCCAGAACUGC-------GAUCUGCAGCCCAACCAUCAGGCAGCCGUCGAUGGACUGCUGCGACUG------UCGCCGCAGCUACCCGAGGGCAGCUAUCA .((((.......((((.((((....)).-------)).)))).((((..(((((.(((....))))))))...((((((....------....)))))).......)))))))).... ( -40.80) >DroEre_CAF1 5093 105 + 1 CAGCUACAACACGCAGCUCCAAAACUGC-------GAUUUGCAGCCUAACCAUCAGGCAGCCGUCGACGGACUGCUCCGACUC------GCGCCCCAGCUACCCGAGGGCAGCUAUCA .((((......(((((........))))-------).......((((.......)))).(((.(((.((((....))))....------((......))....))).))))))).... ( -33.00) >DroYak_CAF1 5950 105 + 1 CAGCUAUAACACUCAGAUCCAGAACUGC-------GAUCUGCAGUCCAAGCAUCAAACAGCCGUCGUUGAACAACUUCGACUC------GCGCCCCAGCUGCCCGAGGGCAGCUAUGA ..(((.....(((((((((((....)).-------)))))).)))...))).(((....((((..((((((....)))))).)------).))...(((((((....))))))).))) ( -34.80) >consensus CAGCUAUAACACGCAGCUCCAGAACUGC_______GAUCUGCAGCCCAACCAUCAGGCAGCCGUCGAUGGACUGCUGCGACUG______GCGCCACAGCUACCCGAGGGCAGCUAUCA .((((.......((((.((................)).)))).((((........((((((((....))).)))))...............((....)).......)))))))).... (-14.47 = -16.34 + 1.87)


| Location | 16,476,021 – 16,476,126 |
|---|---|
| Length | 105 |
| Sequences | 6 |
| Columns | 118 |
| Reading direction | reverse |
| Mean pairwise identity | 75.24 |
| Mean single sequence MFE | -48.18 |
| Consensus MFE | -29.64 |
| Energy contribution | -30.70 |
| Covariance contribution | 1.06 |
| Combinations/Pair | 1.35 |
| Mean z-score | -2.22 |
| Structure conservation index | 0.62 |
| SVM decision value | 1.88 |
| SVM RNA-class probability | 0.981207 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 16476021 105 - 22224390 UGAUAGCUGCCCUCCGGUAGCUGCGGCGA------CAAUCGCAGCAGUCCAUCGACGGCUGCCUGAUGGUUGGGCUGCAGAUC-------GCAGUUCUGGAGCUGCGUGUUAUAGCUG ..((((((((.((((((.((((((((((.------....))).(((((((((((.(((....))).))).))))))))....)-------))))))))))))..))).)))))..... ( -47.70) >DroVir_CAF1 24890 111 - 1 UGGUAGCUGCCCGGCAGCAGCUGUGGCGCCAGAUGCAGCUGCAGUUGCCCGCUGUCCGC-------ACGCUCGGCGGCCGUUUGCAGUGUGCAGCCCUUGGUGGCGCUGUCGUAGCUG ...(((((((..((((((.((....))(((...(((((((((((.((.((((((..(..-------..)..)))))).)).))))))).))))(((...))))))))))))))))))) ( -51.80) >DroSec_CAF1 14231 105 - 1 UGGUAGCUGCCCUCGGGUAGCUGUGGGGA------CAGACGCAGCAGUCCAUCGACGGCUGCCUGAUGGUUGGGCUGCAGAUC-------GCAGUUCUGGAGCUGCGUGUUAUAGCUG ..(((((((((.((((((((((((.((..------..(((......)))..)).)))))))))))).)))..)))))).((((-------((((((....))))))).)))....... ( -48.50) >DroSim_CAF1 11414 105 - 1 UGAUAGCUGCCCUCGGGUAGCUGCGGCGA------CAGUCGCAGCAGUCCAUCGACGGCUGCCUGAUGGUUGGGCUGCAGAUC-------GCAGUUCUGGAGCUGCGUGUUAUAGCUG ....(((((((....)))))))(((((..------..))))).(((((((((((.(((....))).))).)))))))).((((-------((((((....))))))).)))....... ( -46.70) >DroEre_CAF1 5093 105 - 1 UGAUAGCUGCCCUCGGGUAGCUGGGGCGC------GAGUCGGAGCAGUCCGUCGACGGCUGCCUGAUGGUUAGGCUGCAAAUC-------GCAGUUUUGGAGCUGCGUGUUGUAGCUG ........(((.(((((((((((.(((..------..)))(((....))).....))))))))))).)))..((((((((..(-------((((((....)))))))..)))))))). ( -51.40) >DroYak_CAF1 5950 105 - 1 UCAUAGCUGCCCUCGGGCAGCUGGGGCGC------GAGUCGAAGUUGUUCAACGACGGCUGUUUGAUGCUUGGACUGCAGAUC-------GCAGUUCUGGAUCUGAGUGUUAUAGCUG ((.((((((((....)))))))).(((..------..))))).((((.....))))((((((..((((((..((.(.((((..-------.....)))).)))..)))))))))))). ( -43.00) >consensus UGAUAGCUGCCCUCGGGUAGCUGCGGCGA______CAGUCGCAGCAGUCCAUCGACGGCUGCCUGAUGGUUGGGCUGCAGAUC_______GCAGUUCUGGAGCUGCGUGUUAUAGCUG ...((((((((....)))))))).(((................(((((((((((.(((....))).)))).)))))))............((((((....))))))........))). (-29.64 = -30.70 + 1.06)

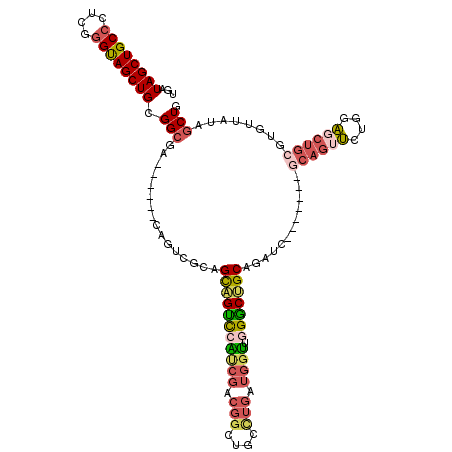
| Location | 16,476,054 – 16,476,163 |
|---|---|
| Length | 109 |
| Sequences | 6 |
| Columns | 118 |
| Reading direction | reverse |
| Mean pairwise identity | 78.21 |
| Mean single sequence MFE | -54.72 |
| Consensus MFE | -33.73 |
| Energy contribution | -36.97 |
| Covariance contribution | 3.23 |
| Combinations/Pair | 1.36 |
| Mean z-score | -1.97 |
| Structure conservation index | 0.62 |
| SVM decision value | 0.99 |
| SVM RNA-class probability | 0.895645 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 16476054 109 - 22224390 ACCGGGUUGGCC---ACAGUCGGGCAGGCCGUAGCUAGUCUGAUAGCUGCCCUCCGGUAGCUGCGGCGA------CAAUCGCAGCAGUCCAUCGACGGCUGCCUGAUGGUUGGGCUGC ..(((.(..(((---(...((((((((.(((((((((......)))))))....((((.(((((.(((.------....))).)))))..))))..))))))))))))))..).))). ( -53.60) >DroVir_CAF1 24930 111 - 1 GCUGCCAUUGCCCAGGCACUCGGGCAGGCCGUAGCUGGUCUGGUAGCUGCCCGGCAGCAGCUGUGGCGCCAGAUGCAGCUGCAGUUGCCCGCUGUCCGC-------ACGCUCGGCGGC ((((((...((...((.((.(((((((.(.((((((((((((((....(((((((....)))).)))))))))).))))))).))))))))..))))..-------..))..)))))) ( -59.40) >DroSec_CAF1 14264 109 - 1 ACCGGGUUGGGC---ACAGUCGGGCAGGCCAUAGCUGGUCUGGUAGCUGCCCUCGGGUAGCUGUGGGGA------CAGACGCAGCAGUCCAUCGACGGCUGCCUGAUGGUUGGGCUGC ....((((.(((---...(((((((((.((...(((((((((.((((((((....))))))))......------))))).)))).(((....)))))))))))))).))).)))).. ( -54.40) >DroSim_CAF1 11447 109 - 1 ACCGGGUUGGCC---ACAGUCGGGCAGGCCGUAGCUGGUCUGAUAGCUGCCCUCGGGUAGCUGCGGCGA------CAGUCGCAGCAGUCCAUCGACGGCUGCCUGAUGGUUGGGCUGC ..(((.(..(((---(...((((((((.((((.(.(((.(((..(((((((....)))))))(((((..------..)))))..))).))).).))))))))))))))))..).))). ( -61.10) >DroEre_CAF1 5126 109 - 1 ACCGGGUUGUGC---ACAUUCGGGCAGGCCGUAGCUGGUCUGAUAGCUGCCCUCGGGUAGCUGGGGCGC------GAGUCGGAGCAGUCCGUCGACGGCUGCCUGAUGGUUAGGCUGC ..(((.(((.((---.(((.(((((((.((((.(.(((.(((.((((((((....)))))))).(((..------..)))....))).))).).))))))))))))))))))).))). ( -51.40) >DroYak_CAF1 5983 109 - 1 ACCGGGCUGGGC---GCAGUCGGGCAGGCCGUAGCUGGUCUCAUAGCUGCCCUCGGGCAGCUGGGGCGC------GAGUCGAAGUUGUUCAACGACGGCUGUUUGAUGCUUGGACUGC ..(((.(..(((---...(((((((((.((((.(((.(((((.((((((((....))))))))))).))------.)))....(((....))).))))))))))))))))..).))). ( -48.40) >consensus ACCGGGUUGGCC___ACAGUCGGGCAGGCCGUAGCUGGUCUGAUAGCUGCCCUCGGGUAGCUGCGGCGA______CAGUCGCAGCAGUCCAUCGACGGCUGCCUGAUGGUUGGGCUGC ..(((.(..(((......(((((((((.((((.(.(((.(((..(((((((....)))))))(((((..........)))))..))).))).).))))))))))))))))..).))). (-33.73 = -36.97 + 3.23)

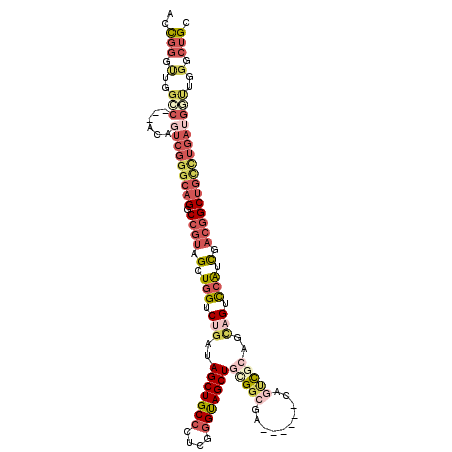
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 12:18:10 2006