
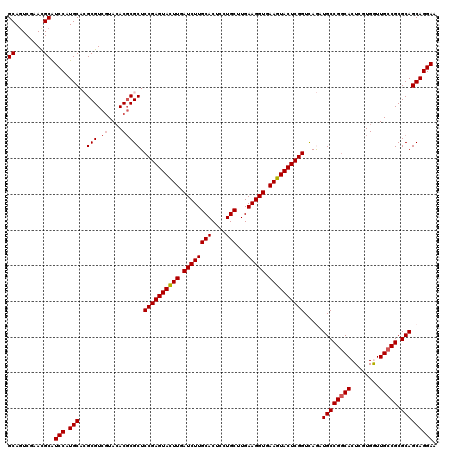
| Sequence ID | X_DroMel_CAF1 |
|---|---|
| Location | 16,392,594 – 16,392,754 |
| Length | 160 |
| Max. P | 0.966939 |

| Location | 16,392,594 – 16,392,714 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 94.94 |
| Mean single sequence MFE | -46.88 |
| Consensus MFE | -43.35 |
| Energy contribution | -43.63 |
| Covariance contribution | 0.28 |
| Combinations/Pair | 1.03 |
| Mean z-score | -1.54 |
| Structure conservation index | 0.92 |
| SVM decision value | 1.60 |
| SVM RNA-class probability | 0.966939 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 16392594 120 + 22224390 GCAGUCGAAUGCGUCCAUGCAGGCGUCGUACACGCGCUCCGAGUACUUGAUCUUGCACUCCUGCUUGAAGGUGAAGUACUCGGUUAAGUGCCGGCACUCGUGGUUGCCGCGCAGCAGGAA (((......))).(((.(((.((((.((....))))))((((((((((.((((((((....)))...))))).)))))))))).....((((((((........))))).))))))))). ( -49.50) >DroVir_CAF1 19257 120 + 1 GCAGUCGAACGCAUCCAUGCACGCGUCGUACACCCGCUCCGAGUAUUUGAUCUUGCACUCCUGCUUGAAGGUGAAGUACUCGGUCAGAUGCCGACACUCGUGAUUGCCGCGCAGCAGGAA ((.((((...(((....)))..(((((((......)).((((((((((.((((((((....)))...))))).))))))))))...)))))))))...((((.....))))..))..... ( -38.90) >DroGri_CAF1 11095 120 + 1 GCAAUCGAACGCAUCCAUGCACGCGUCGUACACGCGCUCCGAGUACUUGAUCUUGCACUCCUGCUUGAAGGUGAAGUACUCGGUCAGAUGCCGGCACUCGUGGUUGCCGCGCAGCAGGAA ((........)).(((.(((.(((((.....)))))..((((((((((.((((((((....)))...))))).)))))))))).....((((((((........))))).))))))))). ( -47.50) >DroYak_CAF1 10001 120 + 1 GCAGUCGAAUGCGUCCAUGCAGGCGUCGUACACGCGCUCCGAGUACUUGAUCUUGCACUCCUGCUUGAAGGUGAAGUACUCGGUUAAGUGCCGGCACUCGUGGUUGCCGCGCAGCAGGAA (((......))).(((.(((.((((.((....))))))((((((((((.((((((((....)))...))))).)))))))))).....((((((((........))))).))))))))). ( -49.50) >DroMoj_CAF1 11565 120 + 1 GCAGUCGAACGCAUCCAUGCACGCGUCGUACACGCGCUCCGAGUAUUUGAUCUUGCACUCCUGCUUGAAGGUGAAGUACUCGGUCAGAUGCCGGCACUCGUGAUUGCCGCGCAGCAGGAA ((........)).(((.(((.(((((.....)))))..((((((((((.((((((((....)))...))))).)))))))))).....((((((((........))))).))))))))). ( -45.20) >DroAna_CAF1 9003 120 + 1 GCAGUCGAACGCGUCCAUGCACGCGUCAUAGACGCGCUCCGAGUACUUGAUCUUGCACUCCUGCUUGAAGGUGAAGUACUCGGUCAGGUGCCGGCACUCGUGGUUGCCGCGCAGCAGGAA ((.(((..((((((......))))))....)))((((.((((((((((.((((((((....)))...))))).))))))))))...(((..(.((....)).)..))))))).))..... ( -50.70) >consensus GCAGUCGAACGCAUCCAUGCACGCGUCGUACACGCGCUCCGAGUACUUGAUCUUGCACUCCUGCUUGAAGGUGAAGUACUCGGUCAGAUGCCGGCACUCGUGGUUGCCGCGCAGCAGGAA ((........)).(((.(((..(((.((....))))).((((((((((.((((((((....)))...))))).)))))))))).....((((((((........))))).))))))))). (-43.35 = -43.63 + 0.28)


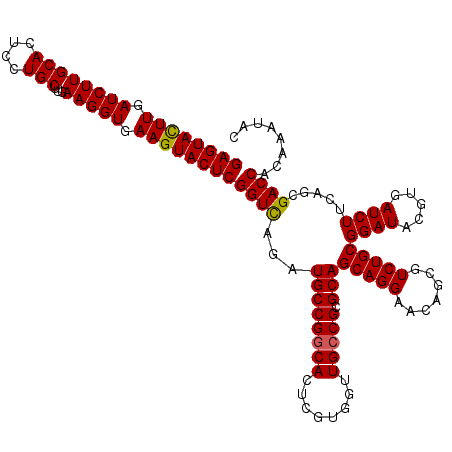
| Location | 16,392,634 – 16,392,754 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 95.44 |
| Mean single sequence MFE | -41.60 |
| Consensus MFE | -39.28 |
| Energy contribution | -39.00 |
| Covariance contribution | -0.28 |
| Combinations/Pair | 1.05 |
| Mean z-score | -0.99 |
| Structure conservation index | 0.94 |
| SVM decision value | 0.49 |
| SVM RNA-class probability | 0.758401 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 16392634 120 + 22224390 GAGUACUUGAUCUUGCACUCCUGCUUGAAGGUGAAGUACUCGGUUAAGUGCCGGCACUCGUGGUUGCCGCGCAGCAGGAACAGCGUCUGCGGAUAGGUGAUCUUUAGCGACCACAAAUAC ((((((((.((((((((....)))...))))).)))))))).....((((....)))).((((((((..(((((.(.(.....).))))))(((.....)))....))))))))...... ( -41.50) >DroVir_CAF1 19297 120 + 1 GAGUAUUUGAUCUUGCACUCCUGCUUGAAGGUGAAGUACUCGGUCAGAUGCCGACACUCGUGAUUGCCGCGCAGCAGGAACAGCGUCUGCGGAUACGUGAUCUUCAGCGACCACAAAUAG ((((((((.((((((((....)))...))))).))))))))((((....((.(((.((.((..((((......))))..)))).))).))((((.....)))).....))))........ ( -36.60) >DroGri_CAF1 11135 120 + 1 GAGUACUUGAUCUUGCACUCCUGCUUGAAGGUGAAGUACUCGGUCAGAUGCCGGCACUCGUGGUUGCCGCGCAGCAGGAACAGCGUCUGCGGAUACGUGAUCUUCAGCGACCACAAAUAG ((((((((.((((((((....)))...))))).))))))))((((...((((((((........))))).)))((((((...((((........))))..))))..))))))........ ( -42.30) >DroSec_CAF1 13720 120 + 1 GAGUACUUGAUCUUGCACUCCUGCUUGAAGGUGAAGUACUCGGUUAAGUGCCGGCACUCGUGGUUGCCGCGCAGCAGGAACAGCGUCUGCGGAUAGGUGAUCUUUAGCGACCACAAAUAC ((((((((.((((((((....)))...))))).)))))))).....((((....)))).((((((((..(((((.(.(.....).))))))(((.....)))....))))))))...... ( -41.50) >DroMoj_CAF1 11605 120 + 1 GAGUAUUUGAUCUUGCACUCCUGCUUGAAGGUGAAGUACUCGGUCAGAUGCCGGCACUCGUGAUUGCCGCGCAGCAGGAACAGCGUCUGCGGAUACGUGAUCUUCAGCGACCACAAAUAG ((((((((.((((((((....)))...))))).))))))))((((...((((((((........))))).)))((((((...((((........))))..))))..))))))........ ( -40.00) >DroAna_CAF1 9043 120 + 1 GAGUACUUGAUCUUGCACUCCUGCUUGAAGGUGAAGUACUCGGUCAGGUGCCGGCACUCGUGGUUGCCGCGCAGCAGGAACAGCGUCUGCGGAUAGCUGAUCUUCAGCGACCACAGAUAC ((((((((.((((((((....)))...))))).))))))))((((.(.((((((((........))))).))).)((((.(((((((....))).)))).))))....))))........ ( -47.70) >consensus GAGUACUUGAUCUUGCACUCCUGCUUGAAGGUGAAGUACUCGGUCAGAUGCCGGCACUCGUGGUUGCCGCGCAGCAGGAACAGCGUCUGCGGAUACGUGAUCUUCAGCGACCACAAAUAC ((((((((.((((((((....)))...))))).))))))))((((...((((((((........))))).)))(((((.......)))))((((.....)))).....))))........ (-39.28 = -39.00 + -0.28)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 12:16:31 2006