
| Sequence ID | X_DroMel_CAF1 |
|---|---|
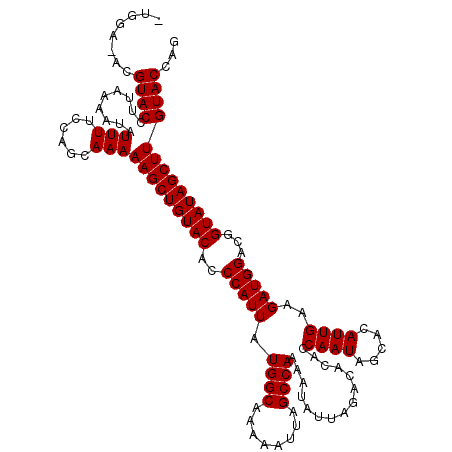
| Location | 16,309,553 – 16,309,672 |
| Length | 119 |
| Max. P | 0.996905 |

| Location | 16,309,553 – 16,309,672 |
|---|---|
| Length | 119 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 97.99 |
| Mean single sequence MFE | -24.41 |
| Consensus MFE | -23.00 |
| Energy contribution | -23.00 |
| Covariance contribution | 0.00 |
| Combinations/Pair | 1.00 |
| Mean z-score | -1.90 |
| Structure conservation index | 0.94 |
| SVM decision value | 2.77 |
| SVM RNA-class probability | 0.996905 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 16309553 119 + 22224390 UUGGC-ACGUACCUUAAAAUAUUUUCCAGCAAAAAGCUGUACACCCAUUAUGGCAAAAAUUAGCCAAAAUAUUAGACACACCAAUAGCACAUUGAAGAUGGACGGUAUAGCUUGUACCAG .(((.-((((..................))...(((((((((..(((((.((((........))))...............((((.....))))..)))))...))))))))))).))). ( -24.87) >DroVir_CAF1 13379 118 + 1 -UUGA-ACGUACCUUAAAAUAUUUUCCAGCAAAAAGCUGUACACCCAUUAUGGCAAAAAUUAGCCAAAAUAUUAGACACACCAAUAGCACAUUGAAGAUGGACGGUAUAGCUUGUACCAG -....-..((((.........(((......)))(((((((((..(((((.((((........))))...............((((.....))))..)))))...)))))))))))))... ( -23.00) >DroGri_CAF1 13750 118 + 1 -UUGA-ACGUACCUUAAAAUAUUUUCCAGCAAAAAGCUGUACACCCAUUAUGGCAAAAAUUAGCCAAAAUAUUAGACACACCAAUAGCACAUUGAAGAUGGACGGUAUAGCUUGUACCAG -....-..((((.........(((......)))(((((((((..(((((.((((........))))...............((((.....))))..)))))...)))))))))))))... ( -23.00) >DroWil_CAF1 14854 120 + 1 UUGGCAACGUACCUUAAAAUAUUUUCCAGCAAAAAGCUGUACACCCAUUAUGGCAAAAAUUAGCCAAAAUAUUAGACACACCAAUAGCACAUUGAAGAUGGACGGUAUAGCUUGUACCAG ..(....)((((.........(((......)))(((((((((..(((((.((((........))))...............((((.....))))..)))))...)))))))))))))... ( -24.00) >DroMoj_CAF1 13523 118 + 1 -GGGA-ACGUACCUUAAAAUAUUUUCCAGCAAAAAGCUGUACACCCAUUAUGGCAAAAAUUAGCCAAAAUAUUAGACACACCAAUAGCACAUUGAAGAUGGACGGUAUAGCUUGUACCAG -.(((-(.(((........))).))))......(((((((((..(((((.((((........))))...............((((.....))))..)))))...)))))))))....... ( -26.70) >DroAna_CAF1 11596 119 + 1 UUGGC-ACGUACCUUAAAAUAUUUUCCAGCAAAAAGCUGUACACCCAUUAUGGCAAAAAUUAGCCAAAAUAUUAGACACACCAAUAGCACAUUGAAGAUGGACGGUAUAGCUUGUACCAG .(((.-((((..................))...(((((((((..(((((.((((........))))...............((((.....))))..)))))...))))))))))).))). ( -24.87) >consensus _UGGA_ACGUACCUUAAAAUAUUUUCCAGCAAAAAGCUGUACACCCAUUAUGGCAAAAAUUAGCCAAAAUAUUAGACACACCAAUAGCACAUUGAAGAUGGACGGUAUAGCUUGUACCAG ........((((.........(((......)))(((((((((..(((((.((((........))))...............((((.....))))..)))))...)))))))))))))... (-23.00 = -23.00 + 0.00)

| Location | 16,309,553 – 16,309,672 |
|---|---|
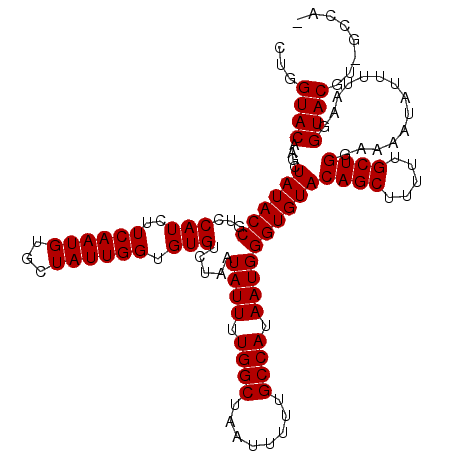
| Length | 119 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 97.99 |
| Mean single sequence MFE | -27.68 |
| Consensus MFE | -26.00 |
| Energy contribution | -26.00 |
| Covariance contribution | 0.00 |
| Combinations/Pair | 1.00 |
| Mean z-score | -1.03 |
| Structure conservation index | 0.94 |
| SVM decision value | 0.53 |
| SVM RNA-class probability | 0.770815 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 16309553 119 - 22224390 CUGGUACAAGCUAUACCGUCCAUCUUCAAUGUGCUAUUGGUGUGUCUAAUAUUUUGGCUAAUUUUUGCCAUAAUGGGUGUACAGCUUUUUGCUGGAAAAUAUUUUAAGGUACGU-GCCAA .((((((....((((((...(((..((((((...)))))).))).....((((.((((........)))).))))))))))((((.....))))..................))-)))). ( -30.40) >DroVir_CAF1 13379 118 - 1 CUGGUACAAGCUAUACCGUCCAUCUUCAAUGUGCUAUUGGUGUGUCUAAUAUUUUGGCUAAUUUUUGCCAUAAUGGGUGUACAGCUUUUUGCUGGAAAAUAUUUUAAGGUACGU-UCAA- ...((((....((((((...(((..((((((...)))))).))).....((((.((((........)))).))))))))))((((.....))))..............))))..-....- ( -26.00) >DroGri_CAF1 13750 118 - 1 CUGGUACAAGCUAUACCGUCCAUCUUCAAUGUGCUAUUGGUGUGUCUAAUAUUUUGGCUAAUUUUUGCCAUAAUGGGUGUACAGCUUUUUGCUGGAAAAUAUUUUAAGGUACGU-UCAA- ...((((....((((((...(((..((((((...)))))).))).....((((.((((........)))).))))))))))((((.....))))..............))))..-....- ( -26.00) >DroWil_CAF1 14854 120 - 1 CUGGUACAAGCUAUACCGUCCAUCUUCAAUGUGCUAUUGGUGUGUCUAAUAUUUUGGCUAAUUUUUGCCAUAAUGGGUGUACAGCUUUUUGCUGGAAAAUAUUUUAAGGUACGUUGCCAA ..((((.......)))).........(((((((((....(((..((((.((...((((........)))))).))))..)))(((.....)))..............))))))))).... ( -27.30) >DroMoj_CAF1 13523 118 - 1 CUGGUACAAGCUAUACCGUCCAUCUUCAAUGUGCUAUUGGUGUGUCUAAUAUUUUGGCUAAUUUUUGCCAUAAUGGGUGUACAGCUUUUUGCUGGAAAAUAUUUUAAGGUACGU-UCCC- ...((((....((((((...(((..((((((...)))))).))).....((((.((((........)))).))))))))))((((.....))))..............))))..-....- ( -26.00) >DroAna_CAF1 11596 119 - 1 CUGGUACAAGCUAUACCGUCCAUCUUCAAUGUGCUAUUGGUGUGUCUAAUAUUUUGGCUAAUUUUUGCCAUAAUGGGUGUACAGCUUUUUGCUGGAAAAUAUUUUAAGGUACGU-GCCAA .((((((....((((((...(((..((((((...)))))).))).....((((.((((........)))).))))))))))((((.....))))..................))-)))). ( -30.40) >consensus CUGGUACAAGCUAUACCGUCCAUCUUCAAUGUGCUAUUGGUGUGUCUAAUAUUUUGGCUAAUUUUUGCCAUAAUGGGUGUACAGCUUUUUGCUGGAAAAUAUUUUAAGGUACGU_GCCA_ ...((((....((((((...(((..((((((...)))))).))).....((((.((((........)))).))))))))))((((.....))))..............))))........ (-26.00 = -26.00 + 0.00)


Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 12:15:34 2006