
| Sequence ID | X_DroMel_CAF1 |
|---|---|
| Location | 16,302,470 – 16,302,563 |
| Length | 93 |
| Max. P | 0.849843 |

| Location | 16,302,470 – 16,302,563 |
|---|---|
| Length | 93 |
| Sequences | 6 |
| Columns | 113 |
| Reading direction | forward |
| Mean pairwise identity | 83.49 |
| Mean single sequence MFE | -52.87 |
| Consensus MFE | -36.63 |
| Energy contribution | -36.38 |
| Covariance contribution | -0.25 |
| Combinations/Pair | 1.12 |
| Mean z-score | -3.16 |
| Structure conservation index | 0.69 |
| SVM decision value | 0.78 |
| SVM RNA-class probability | 0.849843 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
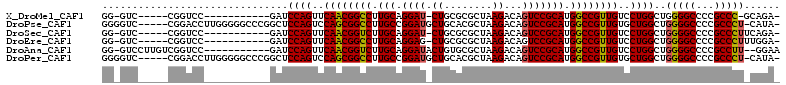
>X_DroMel_CAF1 16302470 93 + 22224390 GG-GUC-----CGGUCC-----------GAUCCAGUUCAACGGCCUUGCAGGAU-CUGCGCGCUAAGACAGUCCGCAUGGCCGUUGUCCUGGCUGGGGCCCCGCCC-GCAGA- ((-((.-----.(((((-----------.(.((((..((((((((.(((.(((.-((((.......).))))))))).))))))))..)))).).)))))..))))-.....- ( -52.10) >DroPse_CAF1 5779 106 + 1 GGGGUC-----CGGACCUUGGGGGCCCGGCUCCAGUCCAGCGGCCUUGCCGGAUGCUGCACGCUAAGACAGUCCGCAUGGCCGUUGUGCUGGCUGGGGCCCCGCCCU-CAUA- (((((.-----...)))))((((((..((((((((((((((((((.(((.((((.((........))...))))))).))))))))....))))))))))..)))))-)...- ( -60.10) >DroSec_CAF1 4244 94 + 1 GG-GUC-----CGGUCC-----------GAUCCAGUUCAACGGUCUUGCAGGAU-CUGCGCGCUAAGACAGUCCGCAUGGCCGUUGUCCUGGCUGGGGCCCCGCCCUUCAGA- ((-((.-----.(((((-----------.(.((((..((((((((.(((.(((.-((((.......).))))))))).))))))))..)))).).)))))..))))......- ( -48.90) >DroEre_CAF1 4730 94 + 1 GG-GUC-----CGGUCC-----------GAUCCAGUUCAACGGCCUUGCAGGAG-CUGCGCGCUAAGACAGUCCGCAUGGCCGUUGUCCUGGCUGGGGCCCCGCCCUUUGGA- ((-((.-----.(((((-----------.(.((((..((((((((.(((.(((.-((((.......).))))))))).))))))))..)))).).)))))..))))......- ( -51.60) >DroAna_CAF1 4667 99 + 1 GG-GUCCUUGUCGGUCC-----------GAUCCAGUUCAACGGUCUUGCAGGAUACUGUGCGCUAAGACAGUCCGCAUGGCCGUUGUCCUGGCUGGGGCCCCGCCUU--GGAA ..-.(((..((.(((((-----------.(.((((..((((((((.(((.(((..((((.(.....))))))))))).))))))))..)))).).)))))..))...--))). ( -44.40) >DroPer_CAF1 2611 106 + 1 GGGGUC-----CGGACCUUGGGGGCCCGGCUCCAGUCCAGCGGCCUUGCCGGAUGCUGCACGCUAAGACAGUCCGCAUGGCCGUUGUGCUGGCUGGGGCCCCGCCCU-CAUA- (((((.-----...)))))((((((..((((((((((((((((((.(((.((((.((........))...))))))).))))))))....))))))))))..)))))-)...- ( -60.10) >consensus GG_GUC_____CGGUCC___________GAUCCAGUUCAACGGCCUUGCAGGAU_CUGCGCGCUAAGACAGUCCGCAUGGCCGUUGUCCUGGCUGGGGCCCCGCCCU_CAGA_ ...............................((((..((((((((.(((.((((.((........))...))))))).))))))))..))))..(((((...)))))...... (-36.63 = -36.38 + -0.25)

| Location | 16,302,470 – 16,302,563 |
|---|---|
| Length | 93 |
| Sequences | 6 |
| Columns | 113 |
| Reading direction | reverse |
| Mean pairwise identity | 83.49 |
| Mean single sequence MFE | -45.85 |
| Consensus MFE | -34.06 |
| Energy contribution | -34.58 |
| Covariance contribution | 0.53 |
| Combinations/Pair | 1.04 |
| Mean z-score | -1.90 |
| Structure conservation index | 0.74 |
| SVM decision value | 0.25 |
| SVM RNA-class probability | 0.652844 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
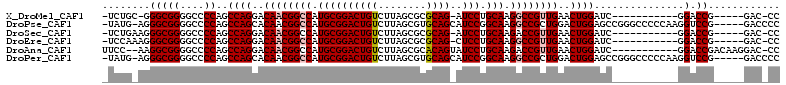
>X_DroMel_CAF1 16302470 93 - 22224390 -UCUGC-GGGCGGGGCCCCAGCCAGGACAACGGCCAUGCGGACUGUCUUAGCGCGCAG-AUCCUGCAAGGCCGUUGAACUGGAUC-----------GGACCG-----GAC-CC -.....-((((.((..((...((((..((((((((.((((((((((........))))-.))).))).))))))))..))))...-----------)).)).-----).)-)) ( -46.90) >DroPse_CAF1 5779 106 - 1 -UAUG-AGGGCGGGGCCCCAGCCAGCACAACGGCCAUGCGGACUGUCUUAGCGUGCAGCAUCCGGCAAGGCCGCUGGACUGGAGCCGGGCCCCCAAGGUCCG-----GACCCC -....-.(((((((((((...((((..((.(((((.((((((((((........))))..))).))).))))).))..))))....)))))))....)))).-----...... ( -52.10) >DroSec_CAF1 4244 94 - 1 -UCUGAAGGGCGGGGCCCCAGCCAGGACAACGGCCAUGCGGACUGUCUUAGCGCGCAG-AUCCUGCAAGACCGUUGAACUGGAUC-----------GGACCG-----GAC-CC -......((((.((..((...((((..((((((.(.((((((((((........))))-.))).))).).))))))..))))...-----------)).)).-----).)-)) ( -39.70) >DroEre_CAF1 4730 94 - 1 -UCCAAAGGGCGGGGCCCCAGCCAGGACAACGGCCAUGCGGACUGUCUUAGCGCGCAG-CUCCUGCAAGGCCGUUGAACUGGAUC-----------GGACCG-----GAC-CC -......((((.((..((...((((..((((((((.((((((((((........))))-.))).))).))))))))..))))...-----------)).)).-----).)-)) ( -46.80) >DroAna_CAF1 4667 99 - 1 UUCC--AAGGCGGGGCCCCAGCCAGGACAACGGCCAUGCGGACUGUCUUAGCGCACAGUAUCCUGCAAGACCGUUGAACUGGAUC-----------GGACCGACAAGGAC-CC .(((--....(((..((....((((..((((((.(.((((((((((........)))))...))))).).))))))..))))...-----------)).)))....))).-.. ( -37.50) >DroPer_CAF1 2611 106 - 1 -UAUG-AGGGCGGGGCCCCAGCCAGCACAACGGCCAUGCGGACUGUCUUAGCGUGCAGCAUCCGGCAAGGCCGCUGGACUGGAGCCGGGCCCCCAAGGUCCG-----GACCCC -....-.(((((((((((...((((..((.(((((.((((((((((........))))..))).))).))))).))..))))....)))))))....)))).-----...... ( -52.10) >consensus _UCUG_AGGGCGGGGCCCCAGCCAGGACAACGGCCAUGCGGACUGUCUUAGCGCGCAG_AUCCUGCAAGGCCGUUGAACUGGAUC___________GGACCG_____GAC_CC ........(((((....))..((((..((((((((.((((((((((........))))..))).))).))))))))..))))...............).))............ (-34.06 = -34.58 + 0.53)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 12:15:23 2006