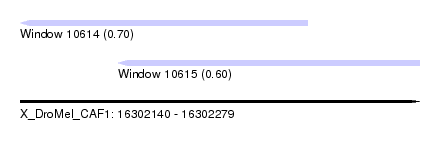
| Sequence ID | X_DroMel_CAF1 |
|---|---|
| Location | 16,302,140 – 16,302,279 |
| Length | 139 |
| Max. P | 0.700293 |

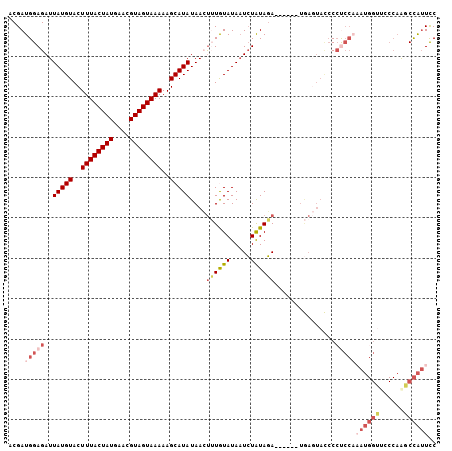
| Location | 16,302,140 – 16,302,240 |
|---|---|
| Length | 100 |
| Sequences | 6 |
| Columns | 106 |
| Reading direction | reverse |
| Mean pairwise identity | 83.13 |
| Mean single sequence MFE | -22.08 |
| Consensus MFE | -14.01 |
| Energy contribution | -15.98 |
| Covariance contribution | 1.98 |
| Combinations/Pair | 1.13 |
| Mean z-score | -2.60 |
| Structure conservation index | 0.63 |
| SVM decision value | 0.35 |
| SVM RNA-class probability | 0.700293 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 16302140 100 - 22224390 ACGAUGGAGAUUAUGUACUUUACUAUGAACGUAGUAAAAAGCAUAUAACUUUAUAUAAUCUAUAGA------UGAGUACCCAUCCAAAUGGUUCCCAAGCCAUUUC ..(((((....(((((..((((((((....))))))))..)))))..((((..((((...))))..------.))))..))))).(((((((......))))))). ( -24.80) >DroSec_CAF1 3928 99 - 1 ACGAUGGAGAUUAUGUACUUUACUAUGAACGUAGUAAAAAGCAUAUAACUUUAUAUAAUCUAUAGA------UGAGUACCCCUCCAAAUGGU-CCCAAGCCAUUCC ....(((((..(((((..((((((((....))))))))..)))))..((((..((((...))))..------.))))....)))))((((((-.....)))))).. ( -23.80) >DroEre_CAF1 4391 100 - 1 ACGAUGGAGAUUAUGUACUUUACUAUGAACGUAGUAAAAAGCAUAUAACUUUGUAUAAUCUAUAGA------UGAGUACCCCUCCACAUGGUUCCCAAGCCAUUCC ..(.(((((..(((((..((((((((....))))))))..))))).......((((.(((....))------)..))))..))))))(((((......)))))... ( -24.40) >DroYak_CAF1 3643 100 - 1 ACGAUGGAGAUUAUGUACUUUACUAUGAACGUAGUAAAAAGCAUAUAACUUUGUAUAUUCUAUAGA------UGAGUACCCCUCCAAAUGGUUCCCAAGCCAUUCC ....(((((..(((((..((((((((....))))))))..)))))..........(((((......------.)))))...)))))((((((......)))))).. ( -24.80) >DroAna_CAF1 4378 92 - 1 ACGAUGGAGAUUAUGUACUUUACUAUGAACGUAGUAAAAAGCAUAUAAACGUGUAUAAUCUAUAGA--------------CCCCCCAAUGGUUCCCAGACCAUUUC ..((((((((((((((((((((((((....))))))))..(........))))))))))))...((--------------((.......))))......))))).. ( -19.10) >DroPer_CAF1 2347 94 - 1 ACGAUGGAGAUUAUGUACUUUACUAUGAACGUAGUAAAAAGCAUAUAAAUGUGUAUAAUAUGUAUAACCAAAUAACUAUCCCCA------------CAGCUGAUCC ..(((((....(((((..((((((((....))))))))..)))))......(((((.....))))).........)))))....------------.......... ( -15.60) >consensus ACGAUGGAGAUUAUGUACUUUACUAUGAACGUAGUAAAAAGCAUAUAACUUUGUAUAAUCUAUAGA______UGAGUACCCCUCCAAAUGGUUCCCAAGCCAUUCC ....(((((..(((((..((((((((....))))))))..)))))....((((((.....))))))...............)))))((((((......)))))).. (-14.01 = -15.98 + 1.98)

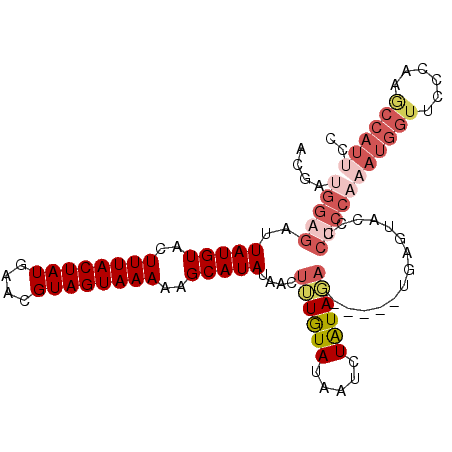
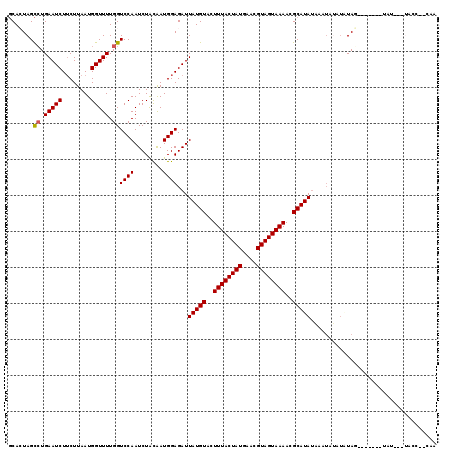
| Location | 16,302,174 – 16,302,279 |
|---|---|
| Length | 105 |
| Sequences | 6 |
| Columns | 119 |
| Reading direction | reverse |
| Mean pairwise identity | 79.57 |
| Mean single sequence MFE | -21.47 |
| Consensus MFE | -17.12 |
| Energy contribution | -17.15 |
| Covariance contribution | 0.03 |
| Combinations/Pair | 1.04 |
| Mean z-score | -1.06 |
| Structure conservation index | 0.80 |
| SVM decision value | 0.13 |
| SVM RNA-class probability | 0.596633 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 16302174 105 - 22224390 GCAAUAGCCUGAAUCUUCUUAAUGGUUUUGGUCCAAUCUACGAUGGAGAUUAUGUACUUUACUAUGAACGUAGUAAAAAGCAUAUAACUUUAUAUAA-----UCUAUA--GA------- ......(((.(((((........))))).)))..........((((((..(((((..((((((((....))))))))..)))))...))))))....-----......--..------- ( -20.50) >DroVir_CAF1 5340 109 - 1 GCACUAGCCUGAAUCUUCUUAAUGGUUUUGGUCCAAUCUGUAAUGGAGAUUAUGUACUUUACUAUGAACGUAGUAAAACGCAUAUUAAUAGGUUUAGAUA----------UGCCUCCAA ......(((.(((((........))))).)))...........(((((.........((((((((....))))))))..(((((((..........))))----------)))))))). ( -24.90) >DroGri_CAF1 5850 99 - 1 GCACUAGCCUGAAUCUUCUUAAUGGUUUUGGUCCAAUCUGCAAUGGAGAUUAUGUACUUUACUAUGAACGUAGUAAAACGCAUAU--------AUAGAUA----------UACC--CAA (((...(((.(((((........))))).)))......)))..(((..(((((((..((((((((....))))))))..))))).--------......)----------)..)--)). ( -18.91) >DroWil_CAF1 6099 102 - 1 UCAGUAGUAUGAAUCUACUUAAUGGUUUUGGUCCAAUCUACAAUGGAGAUUAUGUACUUUACUAUGAACGUAGUAAAACGCAUAUAUAUAUAUGUAU------GUAU----A------- ...((((..((.(((.(((....)))...))).))..)))).........(((((..((((((((....))))))))..)))))(((((((....))------))))----)------- ( -19.10) >DroMoj_CAF1 5607 118 - 1 GCACUAGCCUGAAUCUUCUUAAUGGUUUUGGUCCAAUCUGCAAUGGAGAUUAUGUACUUUACUAUGAACGUAGUAAAACGCAUACUA-UAUAUUUAGAUAGAUCUACCUAUACCUGGUA ..(((((((.(((((........))))).)).............(((((((((((..((((((((....))))))))..)))).(((-......)))...))))).)).....))))). ( -24.30) >DroPer_CAF1 2370 110 - 1 GCACUAGCCUGAAUCUUCUUAAUGGUUUUGGUCCAAUCUACGAUGGAGAUUAUGUACUUUACUAUGAACGUAGUAAAAAGCAUAUAAAUGUGUAUAA-----UAUGUA--UAAC--CAA ((((...((.(((((........))))).))((((........))))...(((((..((((((((....))))))))..))))).....))))....-----......--....--... ( -21.10) >consensus GCACUAGCCUGAAUCUUCUUAAUGGUUUUGGUCCAAUCUACAAUGGAGAUUAUGUACUUUACUAUGAACGUAGUAAAACGCAUAUAAAUAUAUAUAG_______UAU___UACC__CAA .......((.(((((........))))).))((((........))))...(((((..((((((((....))))))))..)))))................................... (-17.12 = -17.15 + 0.03)


Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 12:15:22 2006