
| Sequence ID | X_DroMel_CAF1 |
|---|---|
| Location | 15,990,540 – 15,990,651 |
| Length | 111 |
| Max. P | 0.897898 |

| Location | 15,990,540 – 15,990,651 |
|---|---|
| Length | 111 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 83.29 |
| Mean single sequence MFE | -38.01 |
| Consensus MFE | -24.72 |
| Energy contribution | -25.44 |
| Covariance contribution | 0.72 |
| Combinations/Pair | 1.14 |
| Mean z-score | -2.37 |
| Structure conservation index | 0.65 |
| SVM decision value | 0.97 |
| SVM RNA-class probability | 0.892186 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 15990540 111 + 22224390 -------GAUGGCAGUGCAGCCGCGGGGAAUCGGGAUCGAAUGCUAUAGAUCACCGAUCUCCCACAAUAGGUGGCCCAUCUGCAGAUACACUGGGGAA-AAAUUCUCCAAGAGCCAUGC- -------.(((((..((((((((((((..(((((((((..........)))).)))))..))).(....)))))).....)))).......((((((.-....))))))...)))))..- ( -43.30) >DroSec_CAF1 58462 107 + 1 -------GAUGGCAGUGCAGCC--GGGGAAUCGGGAUCGAAUGCUAUAGAUCACCGAU--CCCACAAUAGGUGGCCCAUCUGCAGAUACACUGGAAAA-AAGUUCUUCAAGAGCCAUGC- -------.(((((..(((((..--.(((.(((((((((..........)))).)))))--.((((.....)))))))..)))))((...(((......-.)))...))....)))))..- ( -35.80) >DroSim_CAF1 49518 114 + 1 GAUUGGGGAUGGCAGUGCAGCC--GGGGAAUCGGGAUCGAAUGCUAUAGAUCACCGAU--CCCACAAUAGGUGGCCCAUCUGCAGAUACACUGUGAAA-AAUUUCUUCAAGAGUCAUGC- ..(((.((((((....((.(((--.((((.((((((((..........)))).)))))--)))......))).)))))))).)))....(((.((((.-......))))..))).....- ( -34.70) >DroEre_CAF1 57367 109 + 1 -------GAUGGCAUUGCAACC--GGGGGAUCGGGAUCGAAUGCUAUAGAUCACCGAU--CCCACAAUAGGUGGCCCCUCUGCAGAUACACCGGGAAAAAAACUCUUCAAGUGUCGAUUU -------..(((((((((.(((--.(((((((((((((..........)))).)))))--))).)....))).))(((..((......))..)))..............))))))).... ( -36.30) >DroYak_CAF1 41487 107 + 1 -------GAUGGGAAUGGGAAU--G-GGGAUCGGGAUCGAAUGCUAUAGAUCACCGAU--CCCACAUAAGGUGGCCCAUCUGCAGAUGCACAGGGAAA-AAAUGCUGCAAGUGCCGUUUU -------((((((.......((--(-((((((((((((..........)))).)))))--))).))).......)))))).(((..((((((......-...)).))))..)))...... ( -39.94) >consensus _______GAUGGCAGUGCAGCC__GGGGAAUCGGGAUCGAAUGCUAUAGAUCACCGAU__CCCACAAUAGGUGGCCCAUCUGCAGAUACACUGGGAAA_AAAUUCUUCAAGAGCCAUGC_ ........(((((..(((((.....(((.(((((((((..........)))).)))))...((((.....)))))))..)))))..........(((........)))....)))))... (-24.72 = -25.44 + 0.72)

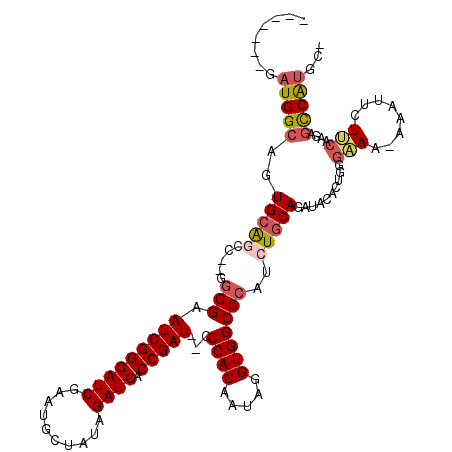
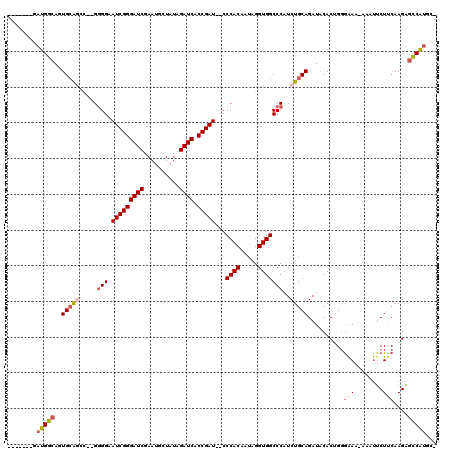
| Location | 15,990,540 – 15,990,651 |
|---|---|
| Length | 111 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 83.29 |
| Mean single sequence MFE | -38.01 |
| Consensus MFE | -21.14 |
| Energy contribution | -22.06 |
| Covariance contribution | 0.92 |
| Combinations/Pair | 1.12 |
| Mean z-score | -2.80 |
| Structure conservation index | 0.56 |
| SVM decision value | 1.00 |
| SVM RNA-class probability | 0.897898 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 15990540 111 - 22224390 -GCAUGGCUCUUGGAGAAUUU-UUCCCCAGUGUAUCUGCAGAUGGGCCACCUAUUGUGGGAGAUCGGUGAUCUAUAGCAUUCGAUCCCGAUUCCCCGCGGCUGCACUGCCAUC------- -..(((((....(((((...)-))))..((((((.((((.((((((...))))))..(((..(((((.((((..........)))))))))..))))))).))))))))))).------- ( -44.60) >DroSec_CAF1 58462 107 - 1 -GCAUGGCUCUUGAAGAACUU-UUUUCCAGUGUAUCUGCAGAUGGGCCACCUAUUGUGGG--AUCGGUGAUCUAUAGCAUUCGAUCCCGAUUCCCC--GGCUGCACUGCCAUC------- -..(((((....(((((....-))))).((((((.(((..((((((...))))))..(((--(((((.((((..........)))))))))))).)--)).))))))))))).------- ( -36.00) >DroSim_CAF1 49518 114 - 1 -GCAUGACUCUUGAAGAAAUU-UUUCACAGUGUAUCUGCAGAUGGGCCACCUAUUGUGGG--AUCGGUGAUCUAUAGCAUUCGAUCCCGAUUCCCC--GGCUGCACUGCCAUCCCCAAUC -(((..((.((((((((...)-))))).)).))...)))...((((.........(.(((--(((((.((((..........)))))))))))).)--(((......)))...))))... ( -34.90) >DroEre_CAF1 57367 109 - 1 AAAUCGACACUUGAAGAGUUUUUUUCCCGGUGUAUCUGCAGAGGGGCCACCUAUUGUGGG--AUCGGUGAUCUAUAGCAUUCGAUCCCGAUCCCCC--GGUUGCAAUGCCAUC------- ......(((((.(((((....)))))..)))))...(((((.((((((((.....))))(--(((((.((((..........))))))))))))))--..)))))........------- ( -36.00) >DroYak_CAF1 41487 107 - 1 AAAACGGCACUUGCAGCAUUU-UUUCCCUGUGCAUCUGCAGAUGGGCCACCUUAUGUGGG--AUCGGUGAUCUAUAGCAUUCGAUCCCGAUCCC-C--AUUCCCAUUCCCAUC------- ......(((..((((.((...-......))))))..))).((((((.......(((.(((--(((((.((((..........))))))))))))-)--)).......))))))------- ( -38.54) >consensus _GCAUGGCUCUUGAAGAAUUU_UUUCCCAGUGUAUCUGCAGAUGGGCCACCUAUUGUGGG__AUCGGUGAUCUAUAGCAUUCGAUCCCGAUUCCCC__GGCUGCACUGCCAUC_______ ...........................(((((((...((....(((((((.....))))...(((((.((((..........))))))))).)))....)))))))))............ (-21.14 = -22.06 + 0.92)



Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 12:10:54 2006