
| Sequence ID | X_DroMel_CAF1 |
|---|---|
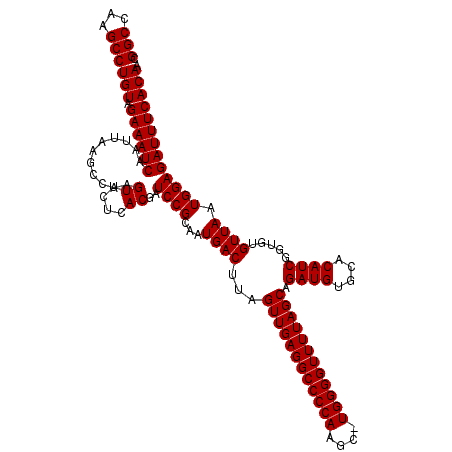
| Location | 15,986,051 – 15,986,211 |
| Length | 160 |
| Max. P | 0.955138 |

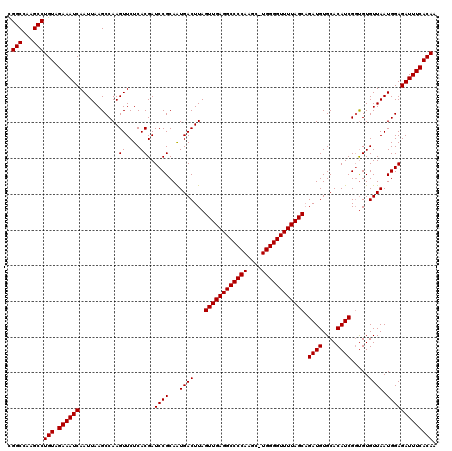
| Location | 15,986,051 – 15,986,171 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 97.32 |
| Mean single sequence MFE | -36.68 |
| Consensus MFE | -35.18 |
| Energy contribution | -35.18 |
| Covariance contribution | -0.00 |
| Combinations/Pair | 1.00 |
| Mean z-score | -1.50 |
| Structure conservation index | 0.96 |
| SVM decision value | 1.45 |
| SVM RNA-class probability | 0.955138 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 15986051 120 + 22224390 CGGCCAAGCCUGUAGAAAUCAAUUAAGCCAAGUUCUCACGAUCCGCAAUGACUUAGUUGAGGCCCCAAGCCUGGGGUUUUAGCAGAUGUGCACAUCGGUGUGUUAAUGGAGAUUUCACAA .(((...)))(((.((((((.(((((((((((((....((...))....))))).((((((((((((....)))))))))))).((((....)))))))...)))))...))))))))). ( -36.00) >DroSec_CAF1 54033 119 + 1 CGGCCAAGCCUGUAGAAAUCAAUUAAGCCAAGUUCUCACGAUCCGCAAUGACUUAGUUGAGGCCCCAAGC-UGGGGUUUUAGCUGAUGUGCACAUCGGUGUGUUAAUGGAGAUUUCACAA .(((...)))(((.((((((.......(((....(.((((((..(((.....((((((((((((((....-.))))))))))))))..)))..))).))).)....))).))))))))). ( -37.40) >DroSim_CAF1 45105 119 + 1 CGGCCAAGCCUGUAGAAAUCAAUUAAGCCAAGUUCUCACGAUCCGCAAUGACUUAGUUGAGGCCCCAAGC-UGGGGUUUUAGCAGAUGUGCACAUCGGUGUGUUAAUGGAGAUUUCACAA .(((...)))(((.((((((.(((((((((((((....((...))....))))).(((((((((((....-.))))))))))).((((....)))))))...)))))...))))))))). ( -35.20) >DroEre_CAF1 52752 119 + 1 UGGCCAGGCCUGUAGAAAUCAAUUAAGCCAAGUUCUCACGAUCCGCAGUGACUUAGUUGAGGCCCCAAGC-UGGGGUUUUAGCAGAUGUGCACAUCGGUGUGUUAAUGGAGAUUUCACAA .(((...)))(((.((((((.((((((((......((((........))))....(((((((((((....-.))))))))))).((((....)))))))...)))))...))))))))). ( -37.60) >DroYak_CAF1 36679 119 + 1 UGGCCAAGCCUGUAGAAAUCAAUUAAGCCAAGUUCUCACGAUCCGCAGUGACUUAGUUGAGGCCCCAAGC-UGGGGUUUUAGCAGAUGUGCACAUCGUCGUGUUAAUGGAGAUUUCACAA .(((...)))(((.((((((.......(((......((((((.............(((((((((((....-.))))))))))).((((....))))))))))....))).))))))))). ( -37.20) >consensus CGGCCAAGCCUGUAGAAAUCAAUUAAGCCAAGUUCUCACGAUCCGCAAUGACUUAGUUGAGGCCCCAAGC_UGGGGUUUUAGCAGAUGUGCACAUCGGUGUGUUAAUGGAGAUUUCACAA .(((...)))(((.((((((...........((....))..((((...((((...((((((((((((....)))))))))))).((((....)))).....)))).))))))))))))). (-35.18 = -35.18 + -0.00)

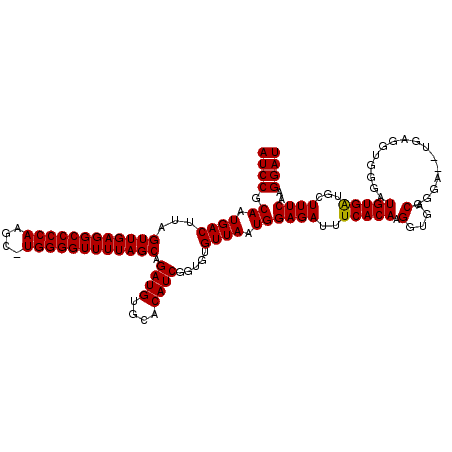
| Location | 15,986,091 – 15,986,211 |
|---|---|
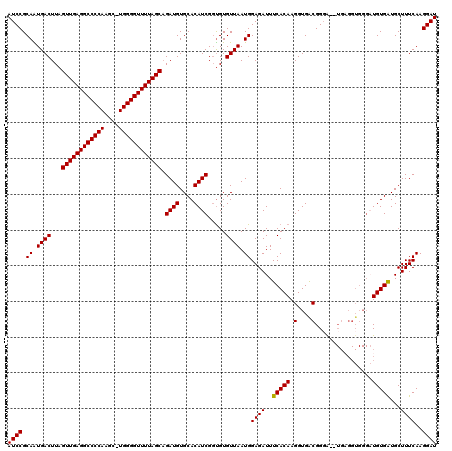
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 91.75 |
| Mean single sequence MFE | -38.02 |
| Consensus MFE | -32.22 |
| Energy contribution | -31.98 |
| Covariance contribution | -0.24 |
| Combinations/Pair | 1.03 |
| Mean z-score | -1.75 |
| Structure conservation index | 0.85 |
| SVM decision value | 1.34 |
| SVM RNA-class probability | 0.943504 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 15986091 120 + 22224390 AUCCGCAAUGACUUAGUUGAGGCCCCAAGCCUGGGGUUUUAGCAGAUGUGCACAUCGGUGUGUUAAUGGAGAUUUCACAAGGUGACGGGAUGUGAGGUGGGAUGUGAUGCUUUCAAGGAU ((((...........((((((((((((....)))))))))))).((.(((((((((.((......))....((((((((.(....)....))))))))..)))))).)))..))..)))) ( -38.40) >DroSec_CAF1 54073 107 + 1 AUCCGCAAUGACUUAGUUGAGGCCCCAAGC-UGGGGUUUUAGCUGAUGUGCACAUCGGUGUGUUAAUGGAGAUUUCACAAGGUGACGGGA------------UGUGAUGCUUUCAAGGAU ((((.((.((((((((((((((((((....-.))))))))))))))...((((....)))))))).))((((..(((((.(....)....------------)))))...))))..)))) ( -38.00) >DroSim_CAF1 45145 119 + 1 AUCCGCAAUGACUUAGUUGAGGCCCCAAGC-UGGGGUUUUAGCAGAUGUGCACAUCGGUGUGUUAAUGGAGAUUUCACAAGGUGACGGGAUGUGAGGUGGGAUGUGAUGCUUUCAAGGAU ((((...........(((((((((((....-.))))))))))).((.(((((((((.((......))....((((((((.(....)....))))))))..)))))).)))..))..)))) ( -37.60) >DroEre_CAF1 52792 117 + 1 AUCCGCAGUGACUUAGUUGAGGCCCCAAGC-UGGGGUUUUAGCAGAUGUGCACAUCGGUGUGUUAAUGGAGAUUUCACAAGGUGGCGGUG--UGAGGUGGGAUGUGGUGCUUUCAAGGAU ..(((((.(.((((.(((((((((((....-.))))))))))).......(((((((.((((..(((....))).))))......)))))--)))))).)..)))))............. ( -36.20) >DroYak_CAF1 36719 117 + 1 AUCCGCAGUGACUUAGUUGAGGCCCCAAGC-UGGGGUUUUAGCAGAUGUGCACAUCGUCGUGUUAAUGGAGAUUUCACAAGGUGACGGGA--UGUGGUGGGCUGUGGUGCUUUCAAGGAU ..(((((((......(((((((((((....-.))))))))))).......(((..((((.(((((.(((......).))...))))).))--))..))).)))))))............. ( -39.90) >consensus AUCCGCAAUGACUUAGUUGAGGCCCCAAGC_UGGGGUUUUAGCAGAUGUGCACAUCGGUGUGUUAAUGGAGAUUUCACAAGGUGACGGGA__UGAGGUGGGAUGUGAUGCUUUCAAGGAU ((((.((.((((...((((((((((((....)))))))))))).((((....)))).....)))).))((((..(((((.......................)))))...))))..)))) (-32.22 = -31.98 + -0.24)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 12:10:42 2006