
| Sequence ID | X_DroMel_CAF1 |
|---|---|
| Location | 15,952,986 – 15,953,106 |
| Length | 120 |
| Max. P | 0.824488 |

| Location | 15,952,986 – 15,953,106 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 93.33 |
| Mean single sequence MFE | -49.84 |
| Consensus MFE | -43.04 |
| Energy contribution | -42.96 |
| Covariance contribution | -0.08 |
| Combinations/Pair | 1.14 |
| Mean z-score | -1.43 |
| Structure conservation index | 0.86 |
| SVM decision value | 0.69 |
| SVM RNA-class probability | 0.824488 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 15952986 120 + 22224390 GUCAUGAUAUGCGAUCGGGACUUUGCCGCUGGCCCGAAUCUUCGGUAAUGCCCGUGGUAUCACCGGCAUGCGCCGGAUGUACACAAUGAUCCGGUGAUCUGGACUGCGAUCGGGAUCUCU (((.((((.((((.((.(((...((((((.(((((((....))))....))).))))))((((((((....)))((((..........)))))))))))).)).)))))))).))).... ( -44.70) >DroSec_CAF1 21354 120 + 1 GUCAUGAUAUGCGAUCGGGACUUUGCCGCUGGCCCGGAUCUUCGGUGAUGUCCGUGGUGUCACCGGCAUGCGCCGGAUGUACACAAUGAUCCGGUGAUCUGGACUGCGAUCGCGAUCUCU .....(((.(((((((.((......))((.((.(((((((.(((((((..(.....)..))))))).....(((((((..........)))))))))))))).))))))))))))))... ( -49.60) >DroSim_CAF1 15875 120 + 1 GUCAUGAUAUGCGAUCGGGACUUUGCCGCUGGCCCGGAUCUUCGGUGAUGCCCGUGGUGUCACCGGCAUGCGCCGGAUGUACACAAUGAUCCGGUGAUCUGGACUGCGAUCGCGAUCUCU .....(((.(((((((.((......))((.((.(((((((.(((((((((((...))))))))))).....(((((((..........)))))))))))))).))))))))))))))... ( -52.60) >DroEre_CAF1 19953 120 + 1 GUCAUGAUAUGCGAUCGGGACUCUGCCGCUGGCCCGAAUCCUCGGUGAUGCCCGUGGUAUCGCCGGCAUGCGCCGGAUGGGCACAGUGAUCCGGUGACCUGGACUGCGAUCGGGAUCUCU .....(((.(.((((((.........(((((((((..((((..(((((((((...)))))))))(((....))))))))))).))))).(((((....)))))...)))))).))))... ( -53.50) >DroYak_CAF1 3414 120 + 1 GUCAUGAUAUGCGAUCGGGACUUUGCCGCUGGCCCGAAUCCUCGGUGAUGCCGGUGGCAUCGACGGCAUGCGCCGGAUGUGCACAGUGAUCCGGUGAUCUGGACUGCGAACGUGAUCUCU ((((((...(((((((....(..((((((((((((((....))))....))))))))))..).((((....))))))).))))..((..(((((....)))))..))...)))))).... ( -48.80) >consensus GUCAUGAUAUGCGAUCGGGACUUUGCCGCUGGCCCGAAUCUUCGGUGAUGCCCGUGGUAUCACCGGCAUGCGCCGGAUGUACACAAUGAUCCGGUGAUCUGGACUGCGAUCGCGAUCUCU .....(((.((((((((......((((((.(((((((....))))....))).))))))...((((....((((((((..........))))))))..))))....)))))))))))... (-43.04 = -42.96 + -0.08)

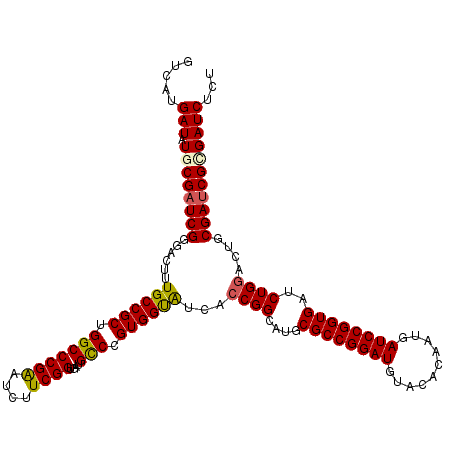

| Location | 15,952,986 – 15,953,106 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 93.33 |
| Mean single sequence MFE | -44.38 |
| Consensus MFE | -36.18 |
| Energy contribution | -36.54 |
| Covariance contribution | 0.36 |
| Combinations/Pair | 1.09 |
| Mean z-score | -1.60 |
| Structure conservation index | 0.82 |
| SVM decision value | 0.59 |
| SVM RNA-class probability | 0.790516 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 15952986 120 - 22224390 AGAGAUCCCGAUCGCAGUCCAGAUCACCGGAUCAUUGUGUACAUCCGGCGCAUGCCGGUGAUACCACGGGCAUUACCGAAGAUUCGGGCCAGCGGCAAAGUCCCGAUCGCAUAUCAUGAC ...(((..((((((..((....((((((((.....(((((.......)))))..))))))))...))((((....((((....))))(((...)))...))))))))))...)))..... ( -40.70) >DroSec_CAF1 21354 120 - 1 AGAGAUCGCGAUCGCAGUCCAGAUCACCGGAUCAUUGUGUACAUCCGGCGCAUGCCGGUGACACCACGGACAUCACCGAAGAUCCGGGCCAGCGGCAAAGUCCCGAUCGCAUAUCAUGAC ...(((.(((((((............(((((((...((((.((.(((((....)))))))))))..(((......)))..)))))))(((...))).......)))))))..)))..... ( -46.60) >DroSim_CAF1 15875 120 - 1 AGAGAUCGCGAUCGCAGUCCAGAUCACCGGAUCAUUGUGUACAUCCGGCGCAUGCCGGUGACACCACGGGCAUCACCGAAGAUCCGGGCCAGCGGCAAAGUCCCGAUCGCAUAUCAUGAC ...(((.(((((((............(((((((...((((.((.(((((....)))))))))))..(((......)))..)))))))(((...))).......)))))))..)))..... ( -47.90) >DroEre_CAF1 19953 120 - 1 AGAGAUCCCGAUCGCAGUCCAGGUCACCGGAUCACUGUGCCCAUCCGGCGCAUGCCGGCGAUACCACGGGCAUCACCGAGGAUUCGGGCCAGCGGCAGAGUCCCGAUCGCAUAUCAUGAC ...(((..((((((..((((..(((.((((.....((((((.....))))))..)))).))).....))))........((((((..(((...))).))))))))))))...)))..... ( -44.70) >DroYak_CAF1 3414 120 - 1 AGAGAUCACGUUCGCAGUCCAGAUCACCGGAUCACUGUGCACAUCCGGCGCAUGCCGUCGAUGCCACCGGCAUCACCGAGGAUUCGGGCCAGCGGCAAAGUCCCGAUCGCAUAUCAUGAC .(.((((..((.((((((...((((....)))))))))).))....((.((.((((((.((((((...)))))).(((......)))....))))))..)).)))))).).......... ( -42.00) >consensus AGAGAUCCCGAUCGCAGUCCAGAUCACCGGAUCAUUGUGUACAUCCGGCGCAUGCCGGUGAUACCACGGGCAUCACCGAAGAUUCGGGCCAGCGGCAAAGUCCCGAUCGCAUAUCAUGAC ...(((..(((((((.((((.((((..(((.....(((((.......)))))((((.(((....))).))))...)))..)))).))))..))((.......)))))))...)))..... (-36.18 = -36.54 + 0.36)



Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 12:09:34 2006